胶质母细胞瘤中异常甲基化调控的蛋白编码基因识别研究
2021-02-05嵇江淮赵潇潇李乾鹏李冬果
嵇江淮 赵潇潇李乾鹏 安 奕 赵 磊 李冬果*
(1.首都医科大学生物医学工程学院生物医学信息学系,北京 100069;2.首都医科大学宣武医院麻醉手术科,北京 100053;3. 国家老年疾病临床研究中心,北京 100053)
胶质母细胞瘤(glioblastoma, GBM)是人类最常见且致死率极高的一种中枢神经系统肿瘤,发生率约占胶质瘤的69%[1]。这种脑瘤具有高浸润性,预后差,患者的中位生存期大约只有一年[2]。近年来,随着大量的蛋白编码基因(protein-coding genes, PCGs)被发现,编码基因表达的失调被证明与疾病的进程有密切关系[3-4]。有研究[5]表明GBM中EGFR基因表达显著升高,促进GBM患者细胞的凋亡。Meng等[6]发现TCTN1基因的过表达促进GBM细胞的增生,并且TCTN1的过表达可以作为预测GBM患者的独立预后因素。这些研究表明PCGs在GBM进程中发挥非常重要的作用,但是这些研究主要关注PCGs的表达模式。目前,GBM中绝大部分PCGs的调控机制(特别是DNA甲基化调控机制)尚不清楚。
DNA甲基化是表观遗传调控的一种重要模式。在基因启动子处的DNA甲基化对基因的表达具有重要的作用,并且参与到许多疾病的进程中[7]。一般来说,基因启动子处的高甲基化往往抑制转录因子的组合并且下调甚至沉默癌症抑制基因,基因启动子的低甲基化往往激活致癌基因[8]。研究[9]表明GBM中MGMT基因启动子甲基化和MGMT的表达具有很强的相关性。然而,这些研究仅仅分析一些特殊的PCGs的DNA甲基化模式,并没有系统分析肿瘤中PCGs的DNA甲基化的模式以及全面评估肿瘤中DNA甲基化对基因表达的调控关系。
随着高通量测序技术的发展,RNA-seq和Infnium 450k等众多数据已经应用于癌症分析。本研究系统整合多组学数据,通过重新注释DNA甲基化阵列,系统分析GBM进程中PCGs的DNA甲基化调控模式。本研究将会帮助理解GBM中DNA甲基化的调控机制,为识别GBM生物标志物和潜在的治疗靶点提供新的见解。
1 数据来源及方法
1.1 数据的来源,整合和标准化
GBM的DNA甲基化数据来自Infinium HM450k平台[10],本研究从TCGA中下载GBM肿瘤样本的HM450k数据,从GEO(GSE41826[11-12],其中包含58个正常胶质细胞样本)中下载正常样本的HM450k数据。正常数据是通过同一平台用同样方法获得的。GBM表达谱数据来自于TCGA数据库,包括5个正常样本数据和152个临床样本信息。为了保证数据的质量,本研究挑选50个同时具有DNA甲基化数据和表达谱数据的样本用作进一步的分析。人类全基因组的注释数据(V19)收集于GENCODE数据库[13]。
对下载的RNA-seq(counts)数据进行log2转换并使用R包“edgeR”[14]对数据进行标准化,最终获得PCGs的表达谱数据。由于HM450k数据存在一定的缺失值,所以,在构建甲基化谱之前需要对含有缺失的数据进行处理。K最近邻填充算法 (K-nearest neighbor, KNN) 是用数据集中缺失数据的k个最近邻来估计缺失值[15]。本研究计算在所有肿瘤样本中具有缺失值的探针数量,并用“DMwR”包中的knnImputation函数补全剩余的缺失值,以评估甲基化探针的质量。最终有89 512个探针被移除,获得392 867个甲基化位点探针。
1.2 PCGs甲基化谱的构建
本研究用映射到PCGs启动子区域探针的甲基化水平来构建PCGs的甲基化谱。采用Zhi等[16]的重注释方法将392 867个探针映射到PCGs的启动子区域(转录开始位点到上游10 kb区域内),使用最接近每个转录开始位点的探针来确定PCGs启动子的DNA甲基化水平[17],从而构建GBM相关的PCGs甲基化谱。
1.3 不同甲基化模式PCGs功能的预测
本研究使用基于线性模型设计的R包“limma”[18]计算肿瘤和正常样本之间的差异甲基化和差异表达。通过Benjamini-Hochberg方法校正P值。为了全面分析表达受对应启动子异常甲基化调控的PCGs,分别将高甲基化的PCGs和低表达的PCGs及低甲基化的PCGs和高表达的PCGs取交集,得到两种具有不同生物学意义的情况:高甲基化且低表达的PCGs及低甲基化且高表达的PCGs。
为了预测不同甲基化模式下PCGs的功能,采用富集分析方法,对挑选出的PCGs进行功能和通路的显著性分析,使用“clusterProfiler”[19]包来预测不同甲基化模式下PCGs的功能。通过Benjamini-Hochberg方法校正P值,如果矫正后的P值≤0.05,该GO项和富集通路就认为是显著的。
1.4 生存分析
为了鉴别挑选出的PCGs是否具有良好的预后效果,本研究基于152个GBM患者的表达谱信息,依据中值将患者分为两组。Kaplan-Meier生存分析和log-rank检测被用来评估两组患者的生存差异。
所有的研究均使用R3.5.1完成。
2 结果
2.1 重注释Infinium HM 450K阵列并构建PCGs的DNA甲基化谱
为了描绘PCGs的DNA甲基化调控模式,本研究将甲基化数据重新注释到人类PCGs相关的启动子区域,共有125 442个探针落在14 684个PCGs启动子区域,通常每一个PCG都有几个探针落在其启动子区域,在此仅保留最接近每个转录开始位点的探针来确定PCGs启动子的DNA甲基化状态。
2.2 整合差异甲基化和差异表达的PCGs
为了能够有效地识别GBM相关的风险标志物,研究差异甲基化可能的生物学意义,基于鉴别出的3 561个差异甲基化的PCGs,其中高甲基化的PCGs有873个(24.5%),低甲基化的PCGs有2 688个(75.5%)。显然低甲基化的PCGs的数量远远多于高甲基化的PCGs的数量。这些结果显示,在GBM的发生发展中PCGs呈现为更多的低甲基化模式,并且这种全局的低甲基化可能导致致癌基因的激活和影响基因组的稳定性。进一步地,本研究鉴别出 6 586 个差异表达的PCGs,其中有2 788个PCGs表达上调,3 798个PCGs表达下调。
为了分析异常甲基化对PCGs表达的调控机制,针对两种不同生物学意义的情况,识别出240个高甲基化且低表达的PCGs及390个低甲基化且高表达的PCGs。结果表明,PCGs在肿瘤样本和正常样本中表现出差异甲基化和差异表达两种模式。
2.3 不同甲基化模式下PCGs的富集分析
对不同甲基化模式下的PCGs,采用富集分析方法进行功能和通路的显著性分析。结果显示,高甲基化的PCGs主要富集在神经元系统发展、细胞-细胞信号传导等生物过程;低甲基化的PCGs富集在许多与肿瘤进程相关的生物过程,比如细胞黏附、细胞迁移、免疫相关的细胞增生和血管生成(图1A、1B)。因此,低甲基化的PCGs可能是影响GBM进程的一个重要模块。对于KEGG通路富集分析,高甲基化的PCGs主要富集在GABAergic突触、胰岛素分泌等通路中,而低甲基化的PCGs主要富集在金黄色葡萄球菌感染、补体和凝血级联等通路中(图1C、1D)。
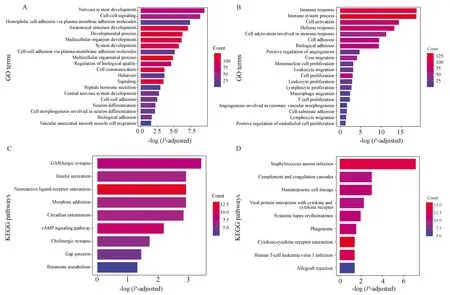
图1 不同甲基化模式下PCGs的富集分析Fig.1 Enrichment analysis of PCGs under different methylation patternsA and B are the GO function analysis of hypermethylated-low expressed PCGs and hypomethylated-highly expressed PCGs,respectively. C and D are KEGG pathway analysis of hypermethylated-low expressed PCGs and hypomethylated-highly expressed PCGs,respectively.The depth of color represents the number of PCGs enriched in the GO item or pathway; PCGs: protein-coding gens.
2.4 GBM中预后良好的PCGs的鉴别
为了评估这些表达受对应启动子异常甲基化影响的PCGs是否可以作为GBM的预后因子,本研究将表达谱数据与临床信息相结合,进行基因生存分析(图2),基于生存分析的结果,获得了51个与GBM整体存活显著相关的PCGs(P<0.05),如表1所示。
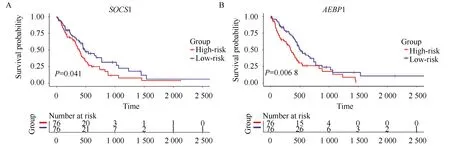
图2 SOCS1和AEBP1基因生存分析Fig.2 Survival analysis of SOCS1 and AEBP1 genesA and B represent Kaplan-Meier curves of SOCS1 and AEBP1 genes in GBM, respectively.The red and blue lines at the bottom represent the number of patients who survived.
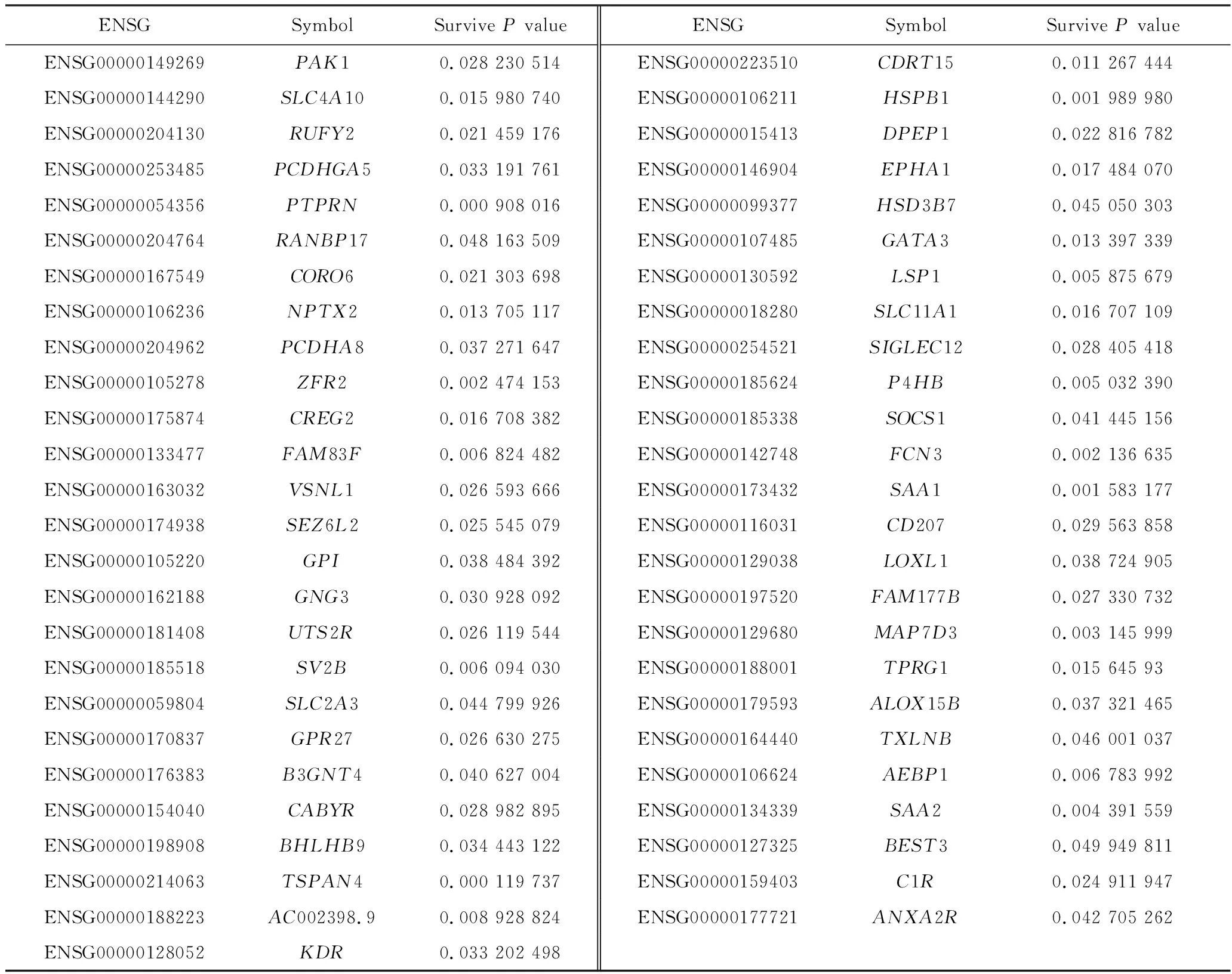
表1 预后良好的PCGsTab.1 PCGs with good prognosis
3 讨论
DNA甲基化的变化可能破坏癌症中特定启动子的调控[20]。近年来,随着高通量测序技术的发展,表观遗传调控已经成为生物医学研究的一大热点。一项研究[21]表明GBM中B3GNT5、FABP7等13个基因启动子的甲基化和表达水平之间呈现负相关。
本研究对重注释得到的PCGs甲基化谱进行差异甲基化分析,结果显示,GBM中多呈现一种低甲基化的模式。为了验证这个结果,本研究重新注释了GBM中lncRNA的甲基化谱,结果发现在鉴别出的5 567个异常甲基化的lncRNAs中高甲基化的有1 214个(21.8%),低甲基化的有4 353个(78.2%)。为了评估是否不同甲基化模式的PCGs对应着不同的生物学功能,对这些基因进行功能富集分析。结果显示低甲基化的PCGs参与了许多与肿瘤发生及进程相关的生物过程。由此推断这些低甲基化的PCGs更有可能成为GBM诊断和治疗的生物标志物。有研究[22-23]表明AEBP1和SOCS1在GBM中过表达,沉默他们的表达可以抑制GBM细胞的增生。在本研究中,AEBP1和SOCS1均属于低甲基化且高表达的基因,进一步地,AEBP1和SOCS1基因表达水平较低的GBM患者具有更加良好的预后。结果表明AEBP1和SOCS1在GBM中表达的上调可能是受它们基因启动子的低甲基化调控,而抑制它们的表达可以提高GBM患者的生存概率。
当然,由于GBM不同于一般的肿瘤,样本的获取是一个重大的难题。本研究需要进一步搜集大规模样本进行重复性计算或者动物实验来验证挖掘出的表达受异常甲基化调控的PCGs以及潜在的治疗靶点。
综上,本研究通过重新注释DNA甲基化阵列,系统识别GBM中潜在的表达受异常甲基化调控的PCGs,加深对GBM中PCGs甲基化调控模式的理解,并对识别GBM风险标志物和潜在的治疗靶点提出了新的认识。
