狼疮性肾炎相关基因的共表达网络构建与分析
2020-07-27梁钊林栩覃卿杜秀日唐志明王晨韦美理
梁钊 林栩 覃卿 杜秀日 唐志明 王晨 韦美理


【摘要】目的基于基因表达综合数据库(GEO)挖掘与狼疮性肾炎(LN)相关的潜在基因。
方法从GEO中搜集LN相关的样本,获得GSE32591、GSE81622、GSE99967共3个数据集。利用GEO2R平台对这3个数据集进行分析,筛选出共同差异基因,并利用在线分析工具DAVID完成GO富集分析和KEGG通路富集分析。将筛选出的共同差异基因导入STRING在线数据库,构建共同差异基因的蛋白蛋白相互作用网络,利用Cytoscape软件进行模块分析并识别枢纽基因。
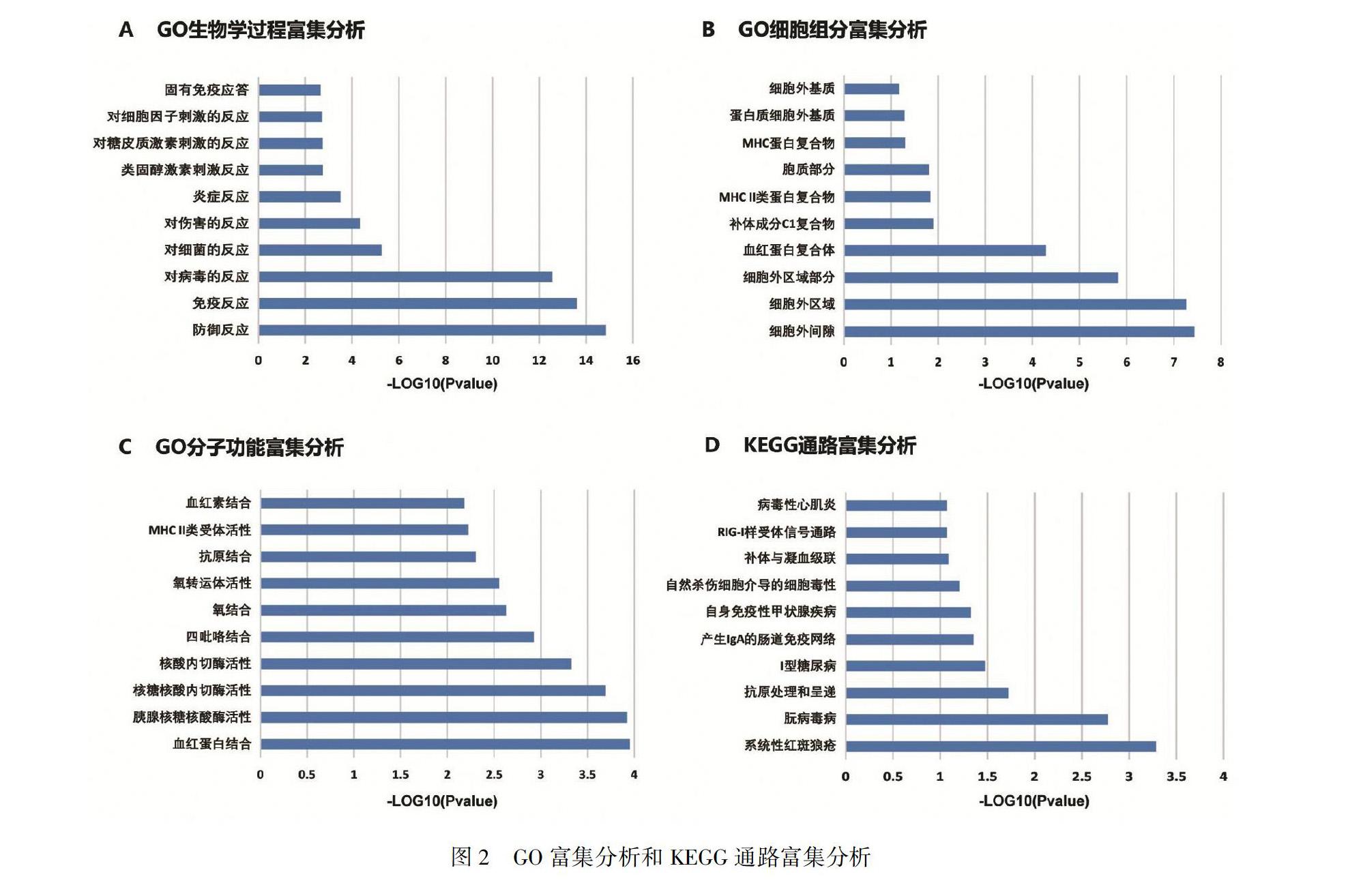
结果筛选得到110个共同差异基因。GO富集分析发现这些基因主要参与了防御反应、免疫反应、对病毒的反应、对细菌的反应、对伤害的反应、炎症反应等生物学过程。KEGG通路富集分析主要包括了系统性红斑狼疮、抗原处理和呈递、补体与凝血级联、RIGI样受体等信号通路。从最显著基因模块中识别出10个枢纽基因:RSAD2、OAS1、MX1、ISG15、DDX58、IFIH1、IFI44、IFI44L、IFIT1、IFIT3。
结论生物信息学分析显示RSAD2、OAS1、MX1、ISG15、DDX58、IFIH1、IFI44、IFI44L、IFIT1、IFIT3可能是与LN相关的枢纽基因,为LN的机制研究提供了一种全新的思路。
【关键词】狼疮性肾炎;生物信息学;基因;共表达网络
中图分类号:R593.24+2文献标志码:ADOI:10.3969/j.issn.10031383.2020.06.002
【Abstract】ObjectiveTo explore potential the genes associated with lupus nephritis(LN) based on Gene Expression Omnibus(GEO).
MethodsLN related samples were collected from the GEO database,and three data sets(GSE32591,GSE81622 and GSE99967) were obtained.GEO2R platform was applied to analyze these three data sets,and common differential genes were screened.In addition,online analysis tool DAVID was used to complete GO enrichment analysis and KEGG pathway enrichment analysis.The common differential genes were imported into the STRING online database,and the proteinprotein interaction network of the common differential genes was constructed.The module analysis and the pivot genes were identified by the Cytoscape software.
Results110 common differential genes were identified.The GO functional enrichment analysis showed that these genes were mainly involved in the biological processes of defense response,immune response,response to virus,response to bacteria,response to injury,and inflammatory response,etc.The KEGG signal pathway enrichment analysis mainly included systemic lupus erythematosus,antigen processing and presentation,complement and coagulation cascades,RIGIlike receptor signaling pathway,etc.Ten pivot genes were identified from the most significant modules:RSAD2,OAS1,MX1,ISG15,DDX58,IFIH1,IFI44,IFI44L,IFIT1 and IFIT3.
ConclusionBioinformatics analysis shows that RSAD2,OAS1,MX1,ISG15,DDX58,IFIH1,IFI44,IFI44L,IFIT1 and IFIT3 may be the pivot genes related to LN,which provides a new idea for the pathogenesis research of LN.
【Key words】LN;bioinformatics;gene;coexpression network
狼疮性肾炎(lupus nephritis,LN)是系统性红斑狼疮(systemic lupus erythematosus,SLE)累及腎脏所引起的一种免疫复合物性肾炎,该病临床表现多样,主要包括血尿、蛋白尿、轻度肾功能损伤、急进性肾功能衰竭等,其发病机制错综复杂[1]。随着基因芯片、RNAseq等高通量测序技术的发展,利用生物信息学方法挖掘公共数据库中与LN相关的基因,对LN的机制研究有着重要的意义。美国国立生物技术信息中心(national center for biotechnology information,NCBI)的基因表达综合数据库(gene expression omnibus,GEO)是一个公共基因表达数据库,收录和储存了大量的高通量实验数据,研究人员可以免费下载这些数据进行分析研究[2]。本研究拟通过挖掘GEO数据库,筛选在LN中有差异表达的基因,构建共表达基因的蛋白互作网络并识别与LN相关的枢纽基因,为LN的机制研究提供一种全新的思路。
1对象与方法
1.1研究对象
对公共的基因芯片数据库GEO(https://www.ncbi.nlm.nih.gov/geo/)进行LN相关样本检索,得到GEO数据库中GSE32591、GSE81622、GSE99967共3个LN人群研究的数据集。GSE32591为来自LN患者肾小球组织和正常对照组、肾间质组织和正常对照组进行对比的表达谱芯片,GSE81622为将LN患者与正常对照组的外周血单个核细胞(peripheral blood mononuclear cell,PBMC)进行比较的表达谱芯片,GSE99967为LN患者与正常对照组的外周血进行比较的表达谱芯片。
1.2研究方法
1.2.1差异基因选取
GEO2R程序是NCBI中基于R语言的在线分析程序,我们使用GEO2R平台(https://www.ncbi.nlm.nih.gov/geo/geo2r/)将样本分组并筛选差异基因,按平台默认方案选出每个芯片中表达差异明显的基因,设定P<0.05、差异倍数绝对值≥1(|logFC|≥1)作为筛选差异基因的阈值,符合条件的差异基因数据认为其表达具有显著性差异。
1.2.2共同差异基因选取
根据3个数据集分组的不同,将GSE32591中LN患者肾小球与正常肾小球对照的芯片数据设为“肾小球组”,将GSE32591中LN患者肾间质与正常肾间质对照的芯片数据设为“肾间质组”,将GSE81622的芯片数据设为“PBMC组”,将GSE99967设为“外周血组”。利用在线分析工具(https://bioinfogp.cnb.csic.es/tools/venny/index.html)筛选出四组数据中的共同差异基因。
1.2.3共同差异基因的GO和KEGG通路分析
对共同差异基因使用在线工具DAVID 6.7(https://davidd.ncifcrf.gov/)进行基因本体(gene ontology,GO)富集分析和京都基因与基因组百科全书通路(kyoto encyclopedia of genes and genomes pathway,KEGG)富集分析。
1.2.4蛋白蛋白相互作用网络的构建(proteinprotein interaction network,PPI网络) 及关键基因的识别
将筛选出的共同差异基因导入在线数据库STRING(https://stringdb.org/)构建PPI网络并进行聚类分析,综合得分>0.4认为蛋白间的相互作用显著。随后,用生物信息学绘图软件Cytoscape的MCODE插件筛选PPI网络中关键节点基因聚类形成的显著基因模块。之后,用Cytoscape的cytoHubba插件从显著基因模块中识别与LN相关的枢纽基因。
2结果
2.1差异基因和共同差异基因的选取结果
肾小球组共筛选出361个差异表达基因,肾间质组共筛选出133个差异表达基因,PBMC组共筛选出158个差异表达基因,外周血组共筛选出122个差异表达基因。用韦恩图筛选出这4组数据的共同表达差异基因,共110个。见图1。
2.2GO富集分析和KEGG通路富集分析
GO富集分析包括生物学功能、细胞组分和分子功能三个方面。将110个共同表达差异基因导入DAVID数据库进行GO富集分析和KEGG通路富集分析,发现富集到的生物学过程主要包括防御反应、免疫反应、对病毒的反应、对细菌的反应、对伤害的反应、炎症反应等过程;参与的细胞组分包括细胞外间隙、细胞外区域、细胞外区域部分、血红蛋白复合体、补体成分C1复合物等;发挥了血红蛋白结合、核糖核酸内切酶活性、核酸内切酶活性、氧结合、抗原结合等分子功能;KEGG通路主要包括了系统性红斑狼疮、抗原处理和呈递、补体与凝血级联、RIGI样受体等信号通路。见图2。
2.3PPI网络
共同差异基因编码蛋白之间的相互作用网络及聚类分析,发现有5个不同类别的聚类,其中不同颜色代表蛋白的不同类别。见图3。
将PPI网络导入Cytoscape,使用MCODE插件筛选显著基因模块,用cytoHubba插件从最显著基因模块中筛选出10个hub基因:RSAD2、OAS1、MX1、ISG15、DDX58、IFIH1、IFI44、IFI44L、IFIT1、IFIT3。见图4。
3讨论
LN是SLE累及肾脏的自身免疫性疾病,遗传、环境、病毒感染、性激素等多种因素均可导致,其发病机制尚未完全明确[3]。GEO数据库收录了大量与疾病相关的高通量数据,对LN的机制研究具有十分重要的意义。本研究纳入GEO数据库中LN患者肾组织、PBMC和外周血的基因表达数据,筛选出共同差异基因110个。
GO富集分析发现,这些共同差异基因在防御反应、免疫反应、对病毒的反应、对细菌的反应、对伤害的反应、炎症反应、类固醇激素刺激的反应、对糖皮质激素刺激的反应、对细胞因子刺激的反应和固有免疫应答等生物学过程发挥作用。有研究显示,LN的发病是免疫复合物沉积于肾组织引起腎脏损伤的过程,这个过程包括炎症细胞募集、细胞因子产生、氧化损伤、补体吸收和成纤维细胞活化增殖等,其中,炎症和纤维化是LN发生发展的关键过程,它们涉及先天性免疫和适应性免疫的免疫细胞与常驻肾细胞的相互作用[4~5]。SLE的发病可能与多种病毒感染相关,如EB病毒、人类疱疹病毒、巨细胞病毒、水痘带状疱疹病毒等[6]。YU等[7]研究发现,在LN患者的肾组织标本中EB病毒阳性率显著高于对照组肾组织标本,且肾组织EB病毒标记阳性组抗Sm阳性率也高于肾组织EB病毒标记阴性组,这些发现提示抗Sm的产生可能与EB病毒感染有关,肾EB病毒感染可能通过诱导产生抗Sm抗体而参与LN的发病机制。
KEGG通路富集分析显示,这些共同差异基因主要富集在系统性红斑狼疮、抗原处理和呈递、补体与凝血级联、RIGI样受体等信号通路。当SLE累及肾脏时,免疫复合物沉积于肾小球内皮细胞和系膜区引起炎症细胞募集、补体激活等过程,最终导致不同程度的肾脏损伤[4]。有研究显示,RIGI在LN患者的肾脏组织标本中有显著的表达[8],此外,还有研究发现LN患者的尿沉渣中RIGI mRNA的表达水平高于IgA肾病患者[9],这些结果表明RIGI可能参与了LN的发生过程。
通过构建PPI网络筛选显著基因模块,我们从最显著基因模块中识别出了10个可能与LN发生发展高度相关的枢纽基因:RSAD2、OAS1、MX1、ISG15、DDX58、IFIH1、IFI44、IFI44L、IFIT1、IFIT3,这些基因均参与Ⅰ型干扰素信号通路。环境因素与SLE的发病密切相关,其中EB病毒感染被认为是SLE的环境危险因素之一。在感染EB病毒后,机体通过多种途径诱导Ⅰ型干扰素信号,并激活免疫系统产生炎症介质和自身抗体参与SLE的发病过程,自身抗体沉积于肾脏引起肾脏损害便可导致LN[10~13]。最近的研究表明,与正常人相比,SLE患者的Ⅰ型干扰素和γ干扰素表达水平均明显升高,且Ⅰ型干扰素和γ干扰素的激活密切相关[14~15]。DER等[16]研究表明,与健康对照组相比,LN患者肾小管细胞中Ⅰ型干扰素的表达水平显著升高,同时Ⅰ型干扰素在LN患者的肾小球系膜细胞和内皮细胞中也有不同程度的升高。这些研究说明,Ⅰ型干扰素与LN的发生发展密不可分,对Ⅰ型干扰素在LN中的发病机制进行深入研究有重要的意义。
RSAD2是干扰素诱导的铁硫簇结合抗病毒蛋白,有研究发现,在SLE患者幼稚CD4+T细胞中甲基化的RSAD2表达明显升高,RSAD2基因CpG位点甲基化与SLE相关自身抗体的产生相关[17~18]。JOSEPH等[19]最近的研究证实,在SLE患者中CpG位点甲基化的RSAD2基因与SLE疾病活动指数相关。而在一项LN全基因组DNA甲基化的研究中,RSAD2基因CpG位点甲基化被证明与LN有很高的相关性[20]。
OAS1是2'5'寡腺苷酸合成酶(OAS)的一种亚型,已知其功能是抗病毒。有研究表明,与对照组相比,SLE患者外周血中的OAS1和OAS1 mRNA表达水平显著升高[21~22]。LANDOLTMARTICORENA等[23]研究显示,B细胞活化因子和OAS1的表达水平在SLE中均升高,且两者的表达水平紧密相关,与对照组相比,SLE患者外周血中的B细胞增生异常,表明OAS1的高表达可能与B细胞的不同表型相关。FENG等[24]对具有不同疾病活动指数的SLE患者进行了研究,并根据病情的变化情况对患者进行3~12个月的随访,发现与正常对照组相比,SLE患者外周血中OAS1的表达水平显著升高,且OAS1的高表达与SLE疾病活动性、LN均密切相关。
MX1是一种干扰素诱导基因。有学者发现,在LN患者肾小球系膜区中MX1染色呈显著阳性,而在IgA肾病、紫癜性肾炎、直立性蛋白尿患者的肾组织中未能检测到MX1的表达[25]。SHIMIZU等[26]研究发现,在LN患者PBMC中MX1蛋白的表达显著高于IgA肾病患者、ANCA相关性血管炎患者和健康对照组,在肾组织活检中发现,LN患者的肾小球和肾小管区域均可检测到MX1,而在IgA肾病和ANCA相关性血管炎患者中仅观察到非常微弱的MX1染色。有研究表明miR155可能通过靶向STAT1信号通路调节Ⅰ型干扰素的应答[27],而LEISS等[28]在使用miR155敲除小鼠研究SLE发病机制时发现,与诱导的野生型狼疮小鼠相比,miR155缺陷型狼疮小鼠自身抗体的血清水平和肾脏损伤程度明显减少,MX1的表达也减少。这些发现提示MX1可能是LN发生发展中的一个重要的调节因子。
ISG15也是一种干扰素诱导基因。YUAN等[29]研究显示,SLE患者血液中ISG15的表达与正常对照组相比明显升高,ISG15的表达水平与SLE疾病活动相关,同时该研究还发现SLE患者体内淋巴细胞的减少与ISG15的表达水平密切相关,其机制可能是ISG15参与淋巴细胞凋亡的过程。HAN等[14]在研究SLE患者EB病毒感染和ISG15的关系时,发现ISG15与EB病毒基因LMP1的表达呈正相关,表明可能是EB病毒的LMP1基因通过激活Ⅰ型干扰素通路参与SLE的发病机制。此前的研究报道显示,使用姥鲛烷(Pristane)诱导的狼疮小鼠ISG15的表达显著上调,而ISG15在用Pristane诱导的miR155敲除小鼠中表达减少,miR155缺陷型狼疮小鼠的自身抗体血清水平和肾脏损伤程度也明显减少,提示ISG15与LN的腎脏损伤密切相关[28]。
DDX58是RIGI的编码基因。RIGI是诱导Ⅰ型干扰素信号的最常见受体,作为病毒传感器,RIGI可以检测到病毒的入侵并触发抗病毒应答,其持续异常的激活会引起自身免疫性疾病[30~31]。SUZUKI等[8]在活动性LN患者的肾脏标本中发现RIGI有显著的表达,且RIGI的表达水平与肾小球病变的严重程度相关。之前有学者发现,地塞米松能够抑制星形胶质细胞的RIGI表达水平[32],IMAIZUMI等[33]的研究发现,γ干扰素能够诱导肾小球系膜细胞RIGI的表达,而使用地塞米松干预不能够影响γ干扰素诱导的RIGI的表达,表明RIGI可能与肾小球的炎症相关,肾小球系膜细胞产生的RIGI可能参与了LN的发病机制。
IFIH1是一种干扰素诱导基因,可以编码黑色素瘤分化相关蛋白(MDA5)。既往的研究显示,MDA5参与病毒感染后干扰素应答调节的过程,且SLE的疾病活性与MDA5的水平呈负相关[34]。FUNABIKI等[35]的研究显示IFIH1 G821S错义突变的小鼠可出现狼疮样症状,该研究在小鼠血清和肾脏中分别检测到抗核抗体、抗dsDNA抗体的表达和免疫球蛋白、补体的沉积,同时,肾脏中干扰素、IL6、IP10等炎性介质的表达均显著上调,说明错义突变的IFIH1可能通过激活干扰素信号触发机体的自身免疫反应。为了验证FUNABIKI等人的研究结果,MUNROE等[36]对SLE患者血液中IFIH1与炎症介质、自身抗体的相关性进行评估,发现IFIH1基因与炎性介质IL6、IP10、自身抗体的表达密切相关。这些研究表明,IFIH1可能与LN的肾脏损伤有关。
IFI44、IFI44L屬于干扰素诱导蛋白家族,是干扰素诱导基因,参与了Ⅰ型干扰素信号通路。KIROU等[37]研究显示,在SLE的PBMC中IFI44的表达显著增高,提示IFI44参与了α干扰素信号通路介导的SLE发生发展过程,但他们的研究并未揭示IFI44与SLE肾脏损伤的关系。ZHAO等[38]研究发现,SLE患者IFI44L启动子DNA甲基化水平显著低于正常对照组,同时,有肾脏受累的SLE患者IFI44L启动子DNA甲基化水平也显著低于没有肾脏受累的SLE患者,提示IF144L启动子的DNA甲基化水平可能与SLE患者的肾脏损害有关。
IFIT1、IFIT3属于干扰素诱导的四肽重复蛋白家族,它们同样是干扰素诱导基因。有研究发现,IFIT1在SLE患者的外周血中表达升高,而在LN患者的肾脏组织中同样也观察到IFIT1的表达升高[39]。HU等[40]使用MRL/lpr狼疮小鼠模型研究IFIT1的表达在LN肾脏病理变化中的作用,发现IFIT1与MRL/lpr小鼠肾组织中Factin、Nephrin和Podocin三种足细胞蛋白的表达呈负相关,进一步的病理分析提示随着IFIT1表达的升高,MRL/lpr小鼠的肾组织中足细胞大量丢失,说明上调IFIT1的表达可能通过破坏足细胞的细胞结构引起肾脏的损伤。WANG等[41]的研究结果表明,与正常对照组相比,IFIT3在SLE患者PBMC表达明显升高,且IFIT3可以激活cGASsting信号通路。XIAO等[42]利用CRISPR/Cas9技术,构建了可产生自身免疫症状的基因突变Trex1D18N/D18N小鼠,当删除Trex1D18N/D18N小鼠cGAS的一个等位基因时发现肾脏的炎症显著减轻,进一步的研究发现,是cGAS介导了Trex1D18N/D18N自身抗体的产生,而自身抗体的产生是狼疮样自身免疫的标志之一。这些发现表明IFIT3和cGAS信号通路可能参与了狼疮样Trex1D18N/D18N小鼠的肾脏损害。
综上所述,本研究运用生物信息学的方法挖掘了与LN相关的枢纽基因,发现这些枢纽基因在LN的发生发展中起到关键的作用,这为LN的机制研究提供了一种全新的思路。本研究的不足之处在于,我们未能通过实验进一步验证这些枢纽基因的表达水平。因此,未来我们可以通过后续的实验研究,验证这些枢纽基因的功能,这可能是未来研究的热点。
声明:本研究不存在任何利益冲突。
参考文献
[1] KRONBICHLER A,BREZINA B,GAUCKLER P,et al.Refractory lupus nephritis:When,why and how to treat[J].Autoimmun Rev,2019,18(5):510518.
[2] CLOUGH E,BARRETT T.The gene expression omnibus database[J].Methods Mol Biol,2016,1418:93110.
[3] ZANDMANGODDARD G,SOLOMON M,ROSMAN Z,et al.Environment and lupusrelated diseases[J].Lupus,2012,21(3):241250.
[4] KOMOLAFE O O.Rapidly progressive glomerulonephritis:a wild card manifestation of lupus nephritis[J].Saudi J Kidney Dis Transpl,2018,29(2):443451.
[5] YUNG S,YAP D Y,CHAN T M.Recent advances in the understanding of renal inflammation and fibrosis in lupus nephritis[J].F1000Res,2017,6:874.
[6] DRABORG A,IZARZUGAZA J M,HOUEN G.How compelling are the data for EpsteinBarr virus being a trigger for systemic lupus and other autoimmune diseases?[J].Curr Opin Rheumatol,2016,28(4):398404.
[7] YU X X,YAO C W,TAO J L,et al.The expression of renal EpsteinBarr virus markers in patients with lupus nephritis[J].Exp Ther Med,2014,7(5):11351140.
[8] SUZUKI K,IMAIZUMI T,TSUGAWA K,et al.Expression of retinoic acidinducible geneI in lupus nephritis[J].Nephrol Dial Transplant,2007,22(8):24072409.
[9] TSUGAWA K,OKI E,SUZUKI K,et al.Expression of mRNA for functional molecules in urinary sediment in glomerulonephritis[J].Pediatr Nephrol,2008,23(3):395401.
[10] HARLEY J B,HARLEY I T,GUTHRIDGE J M,et al.The curiously suspicious:a role for EpsteinBarr virus in lupus[J].Lupus,2006,15(11):768777.
[11] CROW M K,OLFERIEV M,KIROU K A.Type I interferons in autoimmune disease[J].Annu Rev PatholMech Dis,2019,14(1):369393.
[12] RNNBLOM L,ALM G V,ELORANTA M L.The type I interferon system in the development of lupus[J].Semin Immunol,2011,23(2):113121.
[13] MOHAN C,PUTTERMAN C.Genetics and pathogenesis of systemic lupus erythematosus and lupus nephritis[J].Nat Rev Nephrol,2015,11(6):329341.
[14] HAN L,ZHANG Y,WANG Q,et al.EpsteinBarr virus infection and type I interferon signature in patients with systemic lupus erythematosus[J].Lupus,2018:961203317753069.
[15] LIU M M,LIU J L,HAO S M,et al.Higher activation of the interferonGamma signaling pathway in systemic lupus erythematosus patients with a high type I IFN score:relation to disease activity[J].Clin Rheumatol,2018,37(10):26752684.
[16] DER E,SURYAWANSHI H,MOROZOV P,et al.Tubular cell and keratinocyte singlecell transcriptomics applied to lupus nephritis reveal type I IFN and fibrosis relevant pathways[J].Nat Immunol,2019,20(7):915927.
[17] COIT P,JEFFRIES M,ALTOROK N,et al.Genomewide DNA methylation study suggests epigenetic accessibility and transcriptional poising of interferonregulated genes in naive CD4+T cells from lupus patients[J].J Autoimmun,2013,43:7884.
[18] CHUNG S A,NITITHAM J,ELBOUDWAREJ E,et al.Genomewide assessment of differential DNA methylation associated with autoantibody production in systemic lupus erythematosus[J].PLoS One,2015,10(7):e0129813.
[19] JOSEPH S,GEORGE N I,GREENKNOX B,et al.Epigenomewide association study of peripheral blood mononuclear cells in systemic lupus erythematosus:Identifying DNA methylation signatures associated with interferonrelated genes based on ethnicity and SLEDAI[J].J Autoimmun,2019,96:147157.
[20] MOK A,SOLOMON O,NAYAK R R,et al.Genomewide profiling identifies associations between lupus nephritis and differential methylation of genes regulating tissue hypoxia and type 1 interferon responses[J].Lupus Sci Med,2016,3(1):e000183.
[21] YE S,GUO Q,TANG J P,et al.Could 2'5'oligoadenylate synthetase isoforms be biomarkers to differentiate between disease flare and infection in lupus patients?A pilot study[J].Clin Rheumatol,2007,26(2):186190.
[22] FENG X B,WU H,GROSSMAN J M,et al.Association of increased interferoninducible gene expression with disease activity and lupus nephritis in patients with systemic lupus erythematosus[J].Arthritis Rheum,2006,54(9):29512962.
[23] LANDOLTMARTICORENA C,WITHER R,REICH H,et al.Increased expression of B cell activation factor supports the abnormal expansion of transitional B cells in systemic lupus erythematosus[J].J Rheumatol,2011,38(4):642651.
[24] FENG X B,HUANG J,LIU Y,et al.Identification of interferoninducible genes as diagnostic biomarker for systemic lupus erythematosus[J].Clin Rheumatol,2015,34(1):7179.
[25] WATANABE S,IMAIZUMI T,TSURUGA K,et al.Glomerular expression of myxovirus resistance protein 1 in human mesangial cells:possible activation of innate immunity in the pathogenesis of lupus nephritis[J].Nephrology (Carlton),2013,18(12):833837.
[26] SHIMIZU Y,YASUDA S,KIMURA T,et al.Interferoninducible Mx1 protein is highly expressed in renal tissues from treatmentnaive lupus nephritis,but not in those under immunosuppressive treatment[J].Mod Rheumatol,2018,28(4):661669.
[27] GRACIAS D T,STELEKATI E,HOPE J L,et al.The microRNA miR155 controls CD8(+) T cell responses by regulating interferon signaling[J].Nat Immunol,2013,14(6):593602.
[28] LEISS H,SALZBERGER W,JACOBS B,et al.MicroRNA 155deficiency leads to decreased autoantibody levels and reduced severity of nephritis and pneumonitis in pristaneinduced lupus[J].PLoS One,2017,12(7):e0181015.
[29] YUAN Y,MA H,YE Z,et al.Interferonstimulated gene 15 expression in systemic lupus erythematosus:Diagnostic value and association with lymphocytopenia[J].Z Rheumatol,2018,77(3):256262.
[30] KATO H,FUJITA T.RIGIlike receptors and autoimmune diseases[J].Curr Opin Immunol,2015,37:4045.
[31] BUERS I,NITSCHKE Y,RUTSCH F.Novel interferonopathies associated with mutations in RIGI like receptors[J].Cytokine Growth Factor Rev,2016,29:101107.
[32] YOSHIDA H,IMAIZUMI T,LEE S J,et al.Retinoic acidinducible geneI mediates RANTES/CCL5 expression in U373MG human astrocytoma cells stimulated with doublestranded RNA[J].Neurosci Res,2007,58(2):199206.
[33] IMAIZUMI T,TANAKA H,TAJIMA A,et al.Retinoic acidinducible geneI (RIGI) is induced by IFN{Gamma} in human mesangial cells in culture:possible involvement of RIGI in the inflammation in lupus nephritis[J].Lupus,2010,19(7):830836.
[34] SU Y J,CHIU W C,KUO H C.Inverse association between antiviral immunity and lupus disease activity[J].Viral Immunol,2018,31(10):689694.
[35] FUNABIKI M,KATO H,MIYACHI Y,et al.Autoimmune disorders associated with gain of function of the intracellular sensor MDA5[J].Immunity,2014,40(2):199212.
[36] MUNROE M E,PEZANT N,BROWN M A,et al.Association of IFIH1 and proinflammatory mediators:Potential new clues in SLEassociated pathogenesis[J].PLoS One,2017,12(2):e0171193.
[37] KIROU K A,LEE C,GEORGE S,et al.Coordinate overexpression of interferonalphainduced genes in systemic lupus erythematosus[J].Arthritis Rheum,2004,50(12):39583967.
[38] ZHAO M,ZHOU Y,ZHU B C,et al.IFI44L promoter methylation as a blood biomarker for systemic lupus erythematosus[J].Ann Rheum Dis,2016,75(11):19982006.
[39] WECKERLE C E,FRANEK B S,KELLY J A,et al.Network analysis of associations between serum interferonα activity,autoantibodies,and clinical features in systemic lupus erythematosus[J].Arthritis Rheum,2011,63(4):10441053.
[40] HU W P,NIU G D,LI H B,et al.The association between expression of IFIT1 in podocytes of MRL/lpr mice and the renal pathological changes it causes:an animal study[J].Oncotarget,2016,7(47):7646476470.
[41] WANG J H,DAI M,CUI Y G,et al.Association of abnormal elevations in IFIT3 with overactive cyclic GMPAMP synthase/Stimulator of interferon genes signaling in human systemic lupus erythematosus monocytes[J].Arthritis & Rheumatology(Hoboken,N.J.),2018,70(12):20362045.
[42] XIAO N Y,WEI J J,XU S,et al.cGAS activation causes lupuslike autoimmune disorders in a TREX1 mutant mouse model[J].J Autoimmun,2019,100:8494.
(收稿日期:2020-04-10修回日期:2020-05-08)
(編辑:潘明志)
