桑树ISSR-PCR反应体系建立及优化
2020-07-06刘玲唐仕成郑丹柯皓天吕银
刘玲 唐仕成 郑丹 柯皓天 吕银

摘要:采用正交试验设计L16(45)对桑树ISSR-PCR反应体系中的模板DNA、引物、Mg2+、dNTPs和rTaq酶5个因素及反应程序中变性时间、退火时间、延伸时间和循环数进行优化分析。结果表明,各因素水平变化对反应体系的影响大小依次为DNA模板>rTaq酶>Mg2+>引物>dNTPs。最终确立了最佳反应体系,即在10 μL反应体系中,含25 ng/μL DNA模板1 μL、10×PCR buffer 1 μL、20 μmol/L引物0.2 μL、2.5 mmol/L Mg2+ 0.8 μL、2.5 mmol/L dNTPs 1 μL、5 U/μL rTaq 0.1 μL。优化得到的反应程序为94 ℃预变性5 min;94 ℃变性40 s,合适的退火温度退火45 s,72 ℃ 延伸90 s,40个循环;72 ℃延伸10 min,16 ℃保存。通过梯度PCR,确定引物ID37的退火温度为49.5 ℃。稳定性检测表明该体系能用于桑树ISSR分析。
关键词:桑树;正交试验设计;ISSR-PCR;反应体系优化
中图分类号:S888.2 文献标志码: A 文章编号:1002-1302(2020)10-0080-05
收稿日期:2019-05-15
基金项目:四川省科技计划(编号:2019YJ0291)。
作者简介:刘 玲(1989—),女,山西大同人,硕士,工程师,主要从事桑树分子标记工作。E-mail:liuling1022@yeah.net。
桑树是桑科(Moraceae)桑属(Morus L.)多年生木本植物,是重要的经济植物,桑叶是家蚕的主要饲料。我国地域辽阔,生态环境各异,经长期的自然选择和人工选育,形成了非常丰富的桑树种质资源。研究桑树的遗传多样性及其亲缘关系对桑树的栽培育种、良种选育等有重要意义。
简单重复序列间扩增(inter-simple sequence repeat,简称ISSR)标记是一种基于微卫星序列发展起来的分子标记,以PCR技术为基础,利用真核生物基因组中广泛存在的微卫星序列设计引物,对2个距离较近、方向相反的SSR序列之间的基因组片段进行扩增。ISSR分子标记技术结合了RAPD和SSR分子标记的优点,稳定性和重复性好,多态性高。国内外众多研究者已将ISSR分子標记技术应用于桑树种质资源的遗传多样性研究等方面[1]。
本研究参考以往桑树ISSR研究,采用正交设计和极差分析结合的方法,对桑树ISSR-PCR反应体系中的5个因素:模板DNA、引物、Mg2+、dNTPs和rTaq酶,进行4个水平优化,并对反应程序中的变性时间、退火时间、延伸时间和循环次数进行正交设计优化,以期建立适合于桑树ISSR分析的PCR反应体系,为ISSR分子标记在桑树上更广泛地应用提供试验基础。
1 材料与方法
1.1 材料
试验材料采自四川省丝绸科学研究院桑树种质资源圃。选择品种资源“团桑10号”作为PCR反应体系、反应程序和引物退火温度优化的模板。
扩增引物参考由加拿大英属哥伦比亚大学UBC公司2006年公布的ISSR引物序列和已有的ISSR分子标记在桑树方面的应用研究结果[2],由上海生工生物工程股份有限公司合成。经初步试验,选择扩增片段清晰的引物ID37(5′-GAGGAGGAGGAGGC-3′)作为建立反应体系的固定引物。PCR反应所用的Mg2+、dNTPs、rTaq酶、DNA marker和10×buffer均购自TaKaRa公司。
1.2 基因组DNA提取及质量检测
基因组DNA提取参照CTAB法,从约0.15 g硅胶干燥的嫩桑叶中提取。DNA质量用1%琼脂糖凝胶电泳检测,浓度用微量紫外分光光度计检测确认。
1.3 ISSR-PCR反应体系的建立及优化
PCR反应体系中的5个因素:模板DNA、引物、Mg2+、dNTPs和rTaq酶,每个因素分别设置4个水平(表1)。采用L16(45)正交设计(表2),共16组反应体系。每组还含有1 μL 10×buffer。优化模板为团桑10号,引物为ID37,每个反应体系设2次重复。
对正交设计试验结果进行直观分析和极差分析,确定2种方法各自的最佳反应体系;若结果不同,则分别对这2种体系进行PCR扩增,根据电泳结果确定哪种反应体系效果更好。
PCR反应在Thermal Cycler(上海山富科学仪器有限公司)上进行。初始反应体系(总体积10 μL):25 ng模板1 μL、10×buffer 1 μL、20 μmol/L引物0.1 μL、2.5 mmol/L Mg2+ 0.4 μL、2.5 mmol/L dNTPs 0.5 μL、5 U/μL rTaq 0.1 μL,加ddH2O至 10 μL。初始扩增程序:94 ℃预变性5 min;94 ℃变性60 s,50 ℃退火40 s,72 ℃延伸90 s,40个循环;72 ℃延伸10 min,反应结束控制在12 ℃。PCR反应产物经1.5%琼脂糖电泳检测后,在紫外投射反射仪下观察拍照保存。每个反应重复2次。
1.4 ISSR-PCR反应程序的正交试验
采用优化好的反应体系对PCR扩增程序中的变性时间、退火时间、延伸时间和循环次数进行优化,这4个因素水平设置见表3,扩增程序的优化采用L9(34)正交试验设计(表4),每个组合重复扩增2次。产物用1.5%琼脂糖凝胶电泳检测后,对结果进行直观分析,确定最终的PCR反应程序。
1.5 引物退火温度优化
以确定的最佳反应体系为基础,以提取的“团桑10号”DNA作为模板,对引物ID37的退火温度进行优化筛选,退火温度设定范围为47~53 ℃,PCR仪自动生成12个温度。PCR扩增后产物用 1.5% 的琼脂糖凝胶电泳检测,根据扩增结果确定引物ID37(表5)的最佳退火温度。
1.6 桑树ISSR-PCR最佳反应体系稳定性验证
用8个桑树材料:团桑5号、江油12-3、江油19号、江油22号、江油24号、江油402、江油椹1号、白桑,用引物ID7(表5)对优化出的反应体系做稳定性检测,PCR产物用1.5%琼脂糖凝胶电泳检测。
2 结果与分析
2.1 ISSR-PCR正交反应体系的直观分析
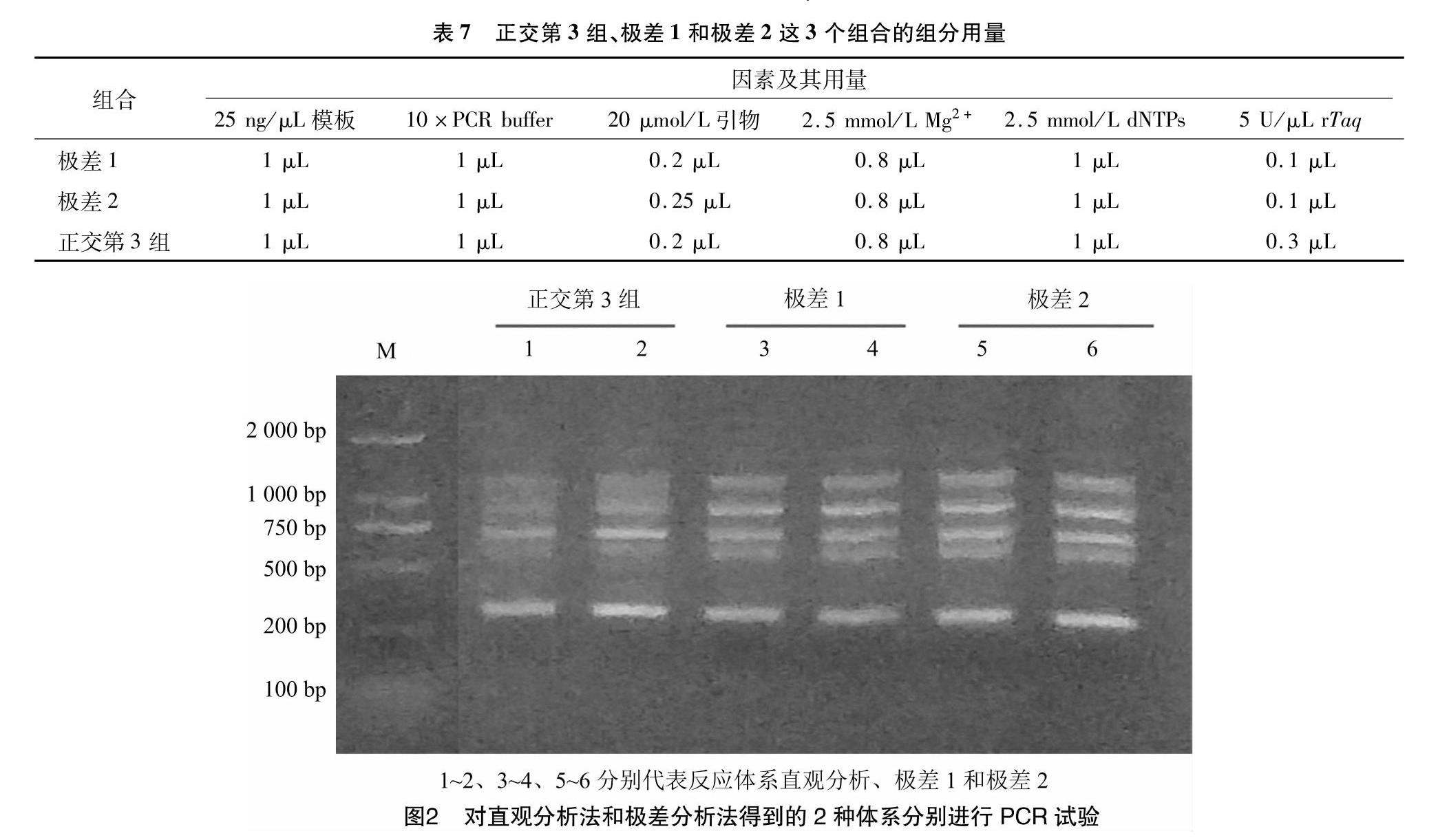
由图1可知,除组合2和组合3外,其余各组扩增条带都存在条带缺失和不清晰的情况。与第3组相比,组合2的扩增条带清晰度弱,因此,电泳结果认为第3组合为最优体系,即DNA模板第1水平、引物第3水平、Mg2+第3水平、dNTPs第3水平、rTaq酶第3水平,即10 μL反应体系中含25 ng模板1 μL、10×PCR buffer 1 μL、20 μmol/L引物 0.2 μL、2.5 mmol/L Mg2+ 0.8 μL、2.5 mmol/L dNTPs 1 μL、5 U/μL rTaq 0.3 μL。
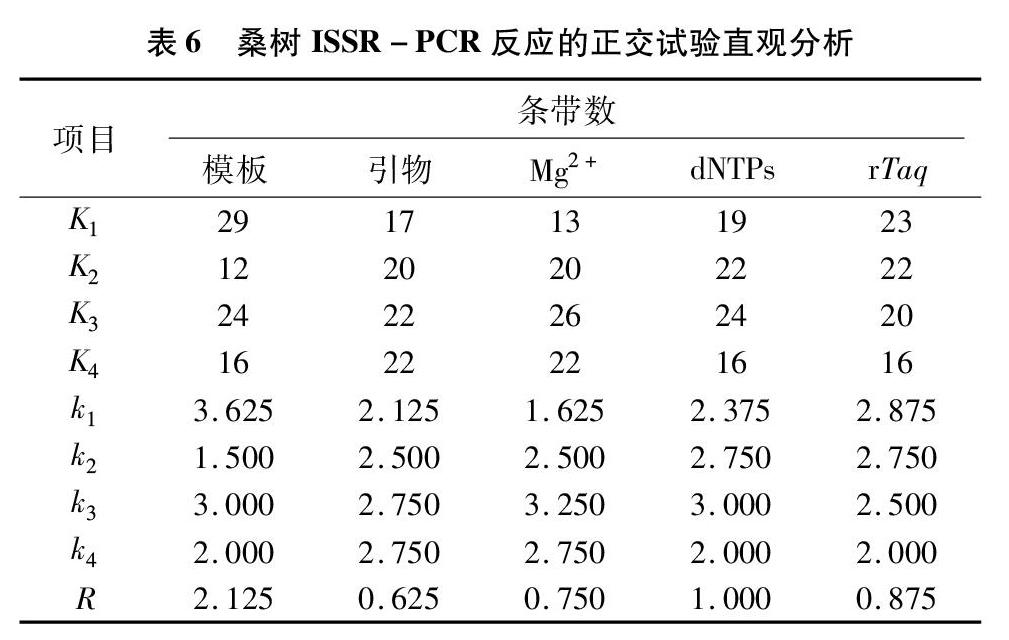
为了进一步证明对试验结果初步判断的准确性,根据李志勇等对正交设计试验中的各组分浓度组合进行K值等分析[3],分析结果见表6。K值代表某水平下某因子参与反应所产生的扩增条带的总和;k代表某因子在某水平下参与反应所产生的扩增条带的平均值;R为某因子的极差,即某因子在不同水平下最大平均值与最小平均值之差。
R值的大小反映了该因子对试验结果影响的大小,R值越大,影响越显著。根据极差R分析可知,本研究中的5个因素在设定的4个水平内对试验结果的影响大小依次为DNA模板>rTaq酶>Mg2+>引物>dNTPs。
k值反映了影响因素各水平对反应体系的影响情况,k值越大,反应水平越好。模板DNA第1水平最好,引物第3、4水平最好,Mg2+第3水平最好,dNTPs第3水平最好,rTaq酶第1水平最好。即极差分析得到2种最佳反应体系(引物量不同),分别命名为极差1和极差2,每个组合的组分用量如表7。
2.2 正交设计直观分析和极差分析结果比较
对正交第3组、极差1和极差2的反应体系作PCR扩增并直观比较。从图2中可以看到,极差1和极差2的扩增效果差不多,谱带亮度强,多态性高且比较稳定,都明显好于正交第3组。反应体系极差1与极差2相比,区别只是引物量少一些。从节约角度考虑,选择极差1为最佳反应体系,即10 μL反应体系中含25 ng/μL模板1 μL、10×buffer 1 μL、20 μmol/L 引物0.2 μL、2.5 mmol/L Mg2+ 08 μL、2.5 mmol/L dNTPs 1 μL、5 U/μL rTaq 01 μL。
2.3 扩增程序优化
采用优化好的反应体系优化桑树ISSR-PCR扩增程序,结果如图3所示。从电泳结果来看,不同组合扩增条带的深浅、优劣不同。第1、2、4、8、9组合条带模糊,不好辨认;第3、5、6、7组合条带相对清晰,其中第3组合条带最好辨认。因此,选择第3组合为桑树ISSR-PCR的扩增程序,即94 ℃预变性 5 min;94 ℃变性40 s,49.5 ℃退火45 s,72 ℃延伸90 s,40个循环;72 ℃延伸10 min,反应结束后 16 ℃保存。
2.4 引物退火温度筛选
由图4可见,退火温度对PCR扩增结果影响明显。当温度高于49.5 ℃时,扩增条带不清晰或者条带不全;温度低于49.5 ℃时,扩增条带清晰、完全;因此,选择49.5 ℃为引物ID37的退火温度。大多数ISSR引物适合较高的退火温度,且能提高 ISSR-PCR反应的特异性及精确度[4],故在47.0~49.5 ℃范围内,选择了略高的49.5 ℃来退火。
2.5 最佳反应体系和程序稳定性检测
采用优化得到的ISSR-PCR反应体系,用引物ID7对8个桑树基因组DNA样品进行扩增反应,以检测优化得到的最佳反应体系和反应程序的稳定性,检测结果如图5所示。
由图5可见,引物ID7从8份种质材料中扩增的条带清晰、稳定、多态性高。表明,经过优化后建立的ISSR-PCR反应体系及程序具有较好的稳定性。
3 讨论与结论
本研究通过直观分析和正交极差分析对比筛选出的最佳体系,结果表明,正交极差分析得到的2组反应体系效果都好于直观分析效果。直观分析带有一定的主观性,极差分析是通过计算将结果转换成数字,能更好地对比出各个因素间的相互作用[5]。
本研究优化得到的桑树ISSR-PCR反应体系为10 μL反应体系中含25 ng模板1 μL、10×PCR buffer 1 μL、20 μmol/L引物0.2 μL、2.5 mmol/L Mg2+ 0.8 μL、2.5 mmol/L dNTPs 1 μL、5 U/μL rTaq 0.1 μL。5个因素在设定的4个水平内对试验结果的影响大小依次为DNA模板>rTaq酶>Mg2+>引物>dNTPs。
桑树ISSR-PCR反应体系仅见思彬彬等在2012年采用正交试验设计和单因素设计结合的方法优化过[6],得到的优化体系为25 μL中10×PCR buffer 2.5 μL、dNTPs 0.35 mmol/L、Mg2+ 2.5 mmol/L、rTaq 0.05 U/μL、引物0.4 μmol/L、DNA 4 ng/μL。本研究与之相比,5个因素的浓度只有rTaq和引物相同,可能是由于试验材料、引物、浓度梯度設置和技术要求不同造成的。
同时,本研究还采用正交试验设计优化了桑树ISSR-PCR反应程序中的变性时间、退火时间、延伸时间和循环数,通过电泳检测PCR产物,得到最适桑树ISSR-PCR反应程序:94 ℃预变性5 min;94 ℃ 变性40 s,49.5 ℃退火45 s,72 ℃延伸90 s,40个循环; 72 ℃延伸10 min, 反应结束后16 ℃保
存。稳定性检验结果表明,得到的反应体系和程序能够扩增出清晰的条带。
本研究得到的反应体系和程序可用于桑树ISSR分析,为桑树系统学和种质资源鉴定及遗传多样性的研究奠定基础。
参考文献:
[1]周延清. DNA分子标记技术在植物研究中的应用[M]. 北京:化学工业出版社,2005:144-149.
[2]陈仁芳,刘 玲,钱永华,等. 北方蚕区89份桑种质资源的ISSR标记遗传多样性分析[J]. 蚕业科学,2015,41(3):410-416.
[3]李志勇,李鸿雁,孙启忠,等. 正交设计优化扁蓿豆ISSR反应体系的研究[J]. 华北农学报,2010,25(6):97-103.
[4]闫 林,黄丽芳,谭乐和,等. 咖啡ISSR与RAPD-PCR反应体系优化[J]. 热带作物学报,2012,33(5):854-859.
[5]张龙进,白成科. 正交设计优化北重楼ISSR-PCR体系[J]. 植物研究,2011,31(1):105-108.
[6]思彬彬,刘明丽. 桑叶ISSR-PCR反应体系的建立与优化[J]. 安徽农业科学,2012,40(3):1329-1331.
