干旱胁迫下玉米开花期雄穗差异表达蛋白质的鉴定与筛选
2020-03-12韩登旭王业建阿布来提阿布拉梁晓玲郗浩江李铭东
杨 杰,韩登旭,王业建,阿布来提·阿布拉,梁晓玲,郗浩江,李铭东,王 仙
(新疆农业科学院粮食作物研究所,乌鲁木齐 830091)
0 引 言
【研究意义】玉米(ZeamaysL.)是中国乃至世界上栽培面积最大的粮食作物,广泛应用于食品、饲料、工业原料、生物质能源等领域,在保障我国粮食和能源安全方面具有重要作用(http://www.fao.org)。干旱胁迫是影响全球作物生产的重要非生物胁迫因子之一,研究植物耐旱机制已成为一个重要领域[1]。我国70%以上的玉米种植区经常遭受干旱的影响,每年造成巨大的损失。玉米是典型的雌雄同株异花作物,雄穗是玉米主要的生殖器官。在雄穗花器官分化期,对干旱胁迫最为敏感,该阶段遭遇干旱胁迫可引起胚子生长发育不正常,导致花粉活力下降,结实不良,造成大幅度的减产[2]。【前人研究进展】有研究认为这种伤害的发生并不是由于生殖器官干旱脱水造成的,是由于营养器官(如叶片)响应干旱胁迫的间接作用引起的[3]。作物耐旱性属于复杂的数量遗传性状,植物对干旱胁迫的应答是一个多基因控制的复合性状,需要多个基因、蛋白和代谢途径共同作用[4]。功能蛋白基因是指在植物逆境抗性中起作用的蛋白,在干旱胁迫下,能在细胞内直接发挥保护功能。调节蛋白是指在信号转导和逆激基因表达过程中起调节作用的蛋白质。主要有磷脂酶、蛋白激酶以及转录因子等,通过这些调节蛋白来提高细胞内信息传递和基因表达能力,提高植物对胁迫环境的适应能力[5-6]。近年来,利用分子技术在转录水平上有研究,并克隆了一些相关基因。但是在蛋白质水平仍然有很多问题尚未解决,干旱胁迫代谢网络、大部分干旱诱导蛋白的功能和表达机理尚不清楚。双向凝胶电泳和质谱技术为研究干旱胁迫诱导产生的蛋白质组变化、寻找耐旱相关基因提供了有利方法[7]。已应用于多种作物的干旱胁迫研究。如使用2-DE和MS对棉花的耐旱性进行研究,检测出个550蛋白点,有16个蛋白上调,6个蛋白下调[8]。有研究通过研究水稻的差异蛋白质组,发现了3种响应干旱胁迫的蛋白质,类胚胎晚期丰富蛋白、叶绿体铜锌SOD蛋白和Rieske铁硫蛋白,其中前2种蛋白上调,后2种蛋白下调。研宄表明,植物蛋白质组在干旱胁迫下是高度动态变化的,蛋白质组学己成为研究不同植物耐旱机制的可行、有效手段[9]。差异蛋白质组学在研究干旱胁迫下玉米的适应性和发掘优异耐旱基因上有着不可忽视的作用。对干早胁迫后4~14 d的2种玉米自交系进行研宄,发现水分亏缺下叶片中有46种蛋白表达量明显增加;部分蛋白表达量的提高具有基因型差异;同时,差异蛋白的变化不同:有些蛋白是恒定增加,有些则先快速增加再稳定增加,而有些只是瞬时增加。这些研究大多是以干旱胁迫下的幼苗期玉米为研究对象,但是,在玉米生育期中,苗期对干旱胁迫的抵抗力相对较强,适度的干旱处理可促进根系发育,增强植株的耐旱性[10]。【本研究切入点】对于干旱胁迫下玉米开花期雄穗差异蛋白质组研究报道较少。分析玉米开花期不同耐旱性玉米自交系的蛋白质表达差异,研究玉米响应干旱胁迫的主要代谢途径并发掘有价值的耐旱基因。【拟解决的关键问题】以强耐旱系PHBA6和弱耐旱系吉63的雄穗小花为材料,采用双向凝胶电泳和质谱分析技术鉴定和筛选差异蛋白,分析不同耐旱性玉米自交系蛋白质组响应干旱的变化规律和内在联系,为研究玉米开花耐旱提供理论依据。
1 材料与方法
1.1 材 料
试验于2015~2016年在新疆农业科学院安宁渠综合试验场进行。采用塑料条盆种植,盆长60 cm,宽30 cm,高cm,花盆内装入草木灰、蛭石和试验田表层土(7∶3∶5)的混合物,每盆重10 kg。每盆种植5株玉米,盆高的3/4埋入土中,自然光照,正常灌水。强耐旱自交系PHBA6(PHZ51×PHG47)和弱耐旱自交系吉63[(127-32×铁84)(W24×W20)辐)],试验材料均由新疆农业科学院粮食作物研究所提供。
样品由南京起肽生物科技有限公司,进行蛋白提取、凝胶电泳、质谱分析和相关数据分析。
1.2 方 法
1.2.1 干旱处理
在玉米雄穗抽出喇叭口之前,控制灌水,进行干旱胁迫。夜间及阴雨天启动防雨棚,晴天白天收起防雨棚,以使玉米植株在自然条件下正常生长。共分干旱胁迫10 d和干旱胁迫0 d(正常灌水~CK)2个处理,每个处理重复3次。分别取各处理植株的雄穗花药,用液氮速冻,于-70℃冻存待用。
1.2.2 总蛋白提取与样品制备(TCA/丙酮法)
将样品放于研钵中,加入液氮研磨粉碎(研磨过程中加入适量 PVP)。将磨好的样品粉末放入 50 mL离心管中,加入-20℃预冷的 10% TCA-丙酮溶液(含 0.1% DTT 和 1 mM PMSF),-20℃静置过夜。15 000 r/min,4℃,离心 20 min,弃上清。在沉淀物中加入-20℃冰箱预冷的丙酮溶液(含 0.1% DTT 和 1 mMPMSF),-20℃静置 2 h。15 000 r/min,4℃,离心 20 min,弃上清。将沉淀物放入冷冻真空干燥机中干燥 30 min。将干燥的蛋白粉末至于-70℃冰箱中保存备用[11]。
1.2.3 蛋白酶解与双向电泳
从胶上切取待鉴定蛋白,分别放入离心管中,写上标记。每管用超纯水清洗胶点3次,弃去水分。每管用50 μL碳酸氢铵(100 mmol/L NH4HCO3)进行脱色。每管用50 μL去离子水处理5 min,弃去多余水分。每管加入50 μL含50% ACN的水溶液处理5 min,弃去多余溶液,加入100% ACN处理5 min。每管加入5μL含10 ng Trypsin酶的碳酸氢铵溶液(50 mmoL/L NH4HCO3)。每管用20 μL碳酸氢铵溶液进行覆盖,37℃酶解16 h。加5 μL5% TFA终止10 min。
蛋白上样量:1 500 μg;胶条24 cm pH 4-7 IPG 预制干胶条,取-20℃冷冻保存的水化上样缓冲液,室温溶解;加入0.01 g DTT,Bio-Lyte 4-7 的电解质 10 L,混匀;取400 L水化上样缓冲液,加入100 L样品,混匀;取出 IPG 预制胶条,将样品与上样缓冲液混合溶液加到聚焦槽中;分清胶条的正负极,将胶条放到聚焦槽中;在每根胶条上覆盖 3 mL 矿物油。盖上盖子,设置等电聚焦程序。
1.2.4 质谱分析与搜库鉴定
吸取酶解后的溶液上清1μL点在样品靶上,待干燥后,同样点1μL的HCCA基质溶液,全部干燥后上机分析。样品靶放入MALDI-TOF-TOF质谱仪,启动控制软件,进行数据采集。
质谱采集参数:正离子反射模式,一级质谱分子量范围为700~3 500 Da,二级质谱分子量范围为40~1 050 Da。合并一级质谱和二级质谱数据,产生peak list文件;利用MASCOT搜索引擎进行蛋白质检索。检索数据库为NCBInr数据库。
1.2.5 电泳凝胶图像
经考马斯亮蓝染色的双向电泳凝胶在UMAX PowerLook 2100XL扫描仪(MaxiumTechnologies, Taipei, China), 300 dpi的分辨率和16位的灰阶,进行图像扫描。
1.2.6 蛋白质质谱分析和数据检索
酶解后的肽段用MALDI-TOF-TOF进行质谱分析。得到的MS/MS数据在MASCOT网站(www.matrixscience.com)(Matrix Science Ltd., London, U.K.)进行搜索,肽质量指纹图谱容许误差为0.3。
1.2.7 蛋白功能分类
使用http://61.50.138.118/gofact上的预测软件GOfac对上述鉴定到的蛋白质进行功能鉴定。
1.2.8 主要农艺性状调查
开花期调查水旱区强耐旱系PHBA6和弱耐旱系吉63植株叶片卷曲度、抽雄期、散粉期、吐丝期等性状。采用SPAD-502Plus便携式叶绿素仪在干旱胁迫期检测叶片叶绿素含量;采用NZ99-TWS-1土壤温度,水分温湿度计对土壤含水量及温湿度进行检测;采用RC-4HC Elitecg仪器对试验小区大气温湿度进行检测。
1.3 数据处理
所有数据均采用Excel2003进行处理,所测定的各生理指标用SPSS 13.0 package for Windows(SPSS Inc.Chicage, IL, USA)中的one-way ANOVA进行方差分析。使用PDQuest软件对图像进行分析,利用 MASCOT 搜索引擎进行蛋白质检索,检索数据库为NCBInr数据库。
2 结果与分析
2.1 差异蛋白质点的质谱鉴定与筛选
2.1.1 双向凝胶电泳
对强耐旱自交系PHBA6和弱耐旱自交系吉63进行干旱胁迫与正常灌溉处理,开花期取其雄穗小花样品,经双向凝胶电泳分离和凝胶图像扫描、分析。以每片胶上样量1 500 μg、上样体积450 μl(不足体积用裂解液补足)的标准,经pH4~7、24 cm非线性IPG预制胶等电聚焦,12.5%聚丙烯酰胺凝胶(SDS-PAGE)作第二向电泳,考马斯亮蓝染色后,获得图谱,经PDQuest 8.0 软件分析得出:考马斯亮蓝染色图谱中共有542个高清晰、重复性强的蛋白质点。图1

注:第一向是PH4-7IPG胶条中进行;第二向在12%de SDS-PAGE胶完成A:吉63干旱胁迫7 d(大图标记)); B:吉63正常灌溉(CK);C:吉63干旱胁迫7 d;C:PHBA6正常灌溉(CK);D: 干旱胁迫7 d
Note:The first is always carried out in PH4-7IPG rubber strip; The second direction is completed at 12% DE sds-page glue.A: J63 drought stress 7 d(large map marker));B: J63 normal irrigation(CK);C: J63 drought stress 7 d;C:PHBA6 normal irrigation(CK);D: drought stress 7 d
图1 PHBA6与吉63雄穗蛋白SDS-PAGE电泳图谱
Fig.1 SDS-PAGE electrophoretogram analysis of PHBA6 and J63 male spike protein
2.1.2 质谱鉴定
对双向凝胶电泳后考马斯亮蓝染色图谱中542个高清晰、重复性强的蛋白质点进行鉴定,筛选出的59个差异表达丰度达2.0倍以上的蛋白质点。对这59个差异表达的蛋白质点再进行MALDI-TOF-TOF质谱分析(质谱采集参数:正离子反射模式,一级质谱分子量范围为700~3 500 Da,二级质谱分子量范围为40~1 050 Da。)、Mascot软件在线搜索。其中,共有57个差异蛋白鉴定成功,2个差异蛋白鉴定失败。对鉴定成功的57个差异蛋白进一步进行信息检索与主要功能分析。
质谱鉴定结果表明,干旱胁迫10 d和0 d(正常灌溉CK)影响PHBA6和吉63花器官分化期雄穗的蛋白质组成变化差异较大。一些功能蛋白质的表达受到干旱胁迫的诱导上调或者抑制下调。鉴定出差异表达丰度达2.0倍以上的蛋白质点共有59个,强耐旱系PHBA6中有26个,弱耐旱系吉63中有37个,在强耐旱系PHBA6与弱耐系吉63中都表达且差异显著的蛋白质点有4个。这些蛋白质分别参与糖代谢、能量代谢、解毒作用、细胞架的构建和细胞程序性死亡等生命活动过程。表1~2
表1 Mascot软件在线搜索参数
Table 1 Online search parameters of Mascot software
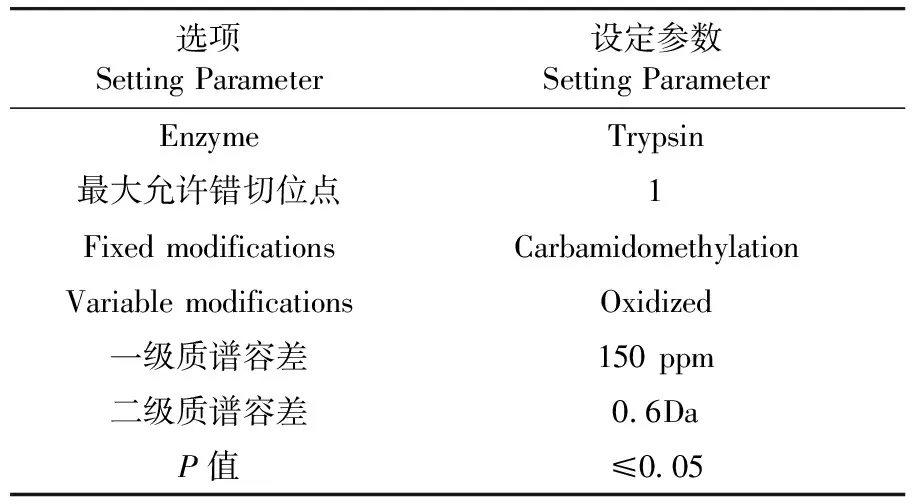
选项SettingParameter设定参数SettingParameterEnzymeTrypsin最大允许错切位点1FixedmodificationsCarbamidomethylationVariablemodificationsOxidized一级质谱容差150ppm二级质谱容差0.6DaP值≤0.05
表2 差异相应蛋白质点信息
Table 2 Statistical table of corresponding protein point information
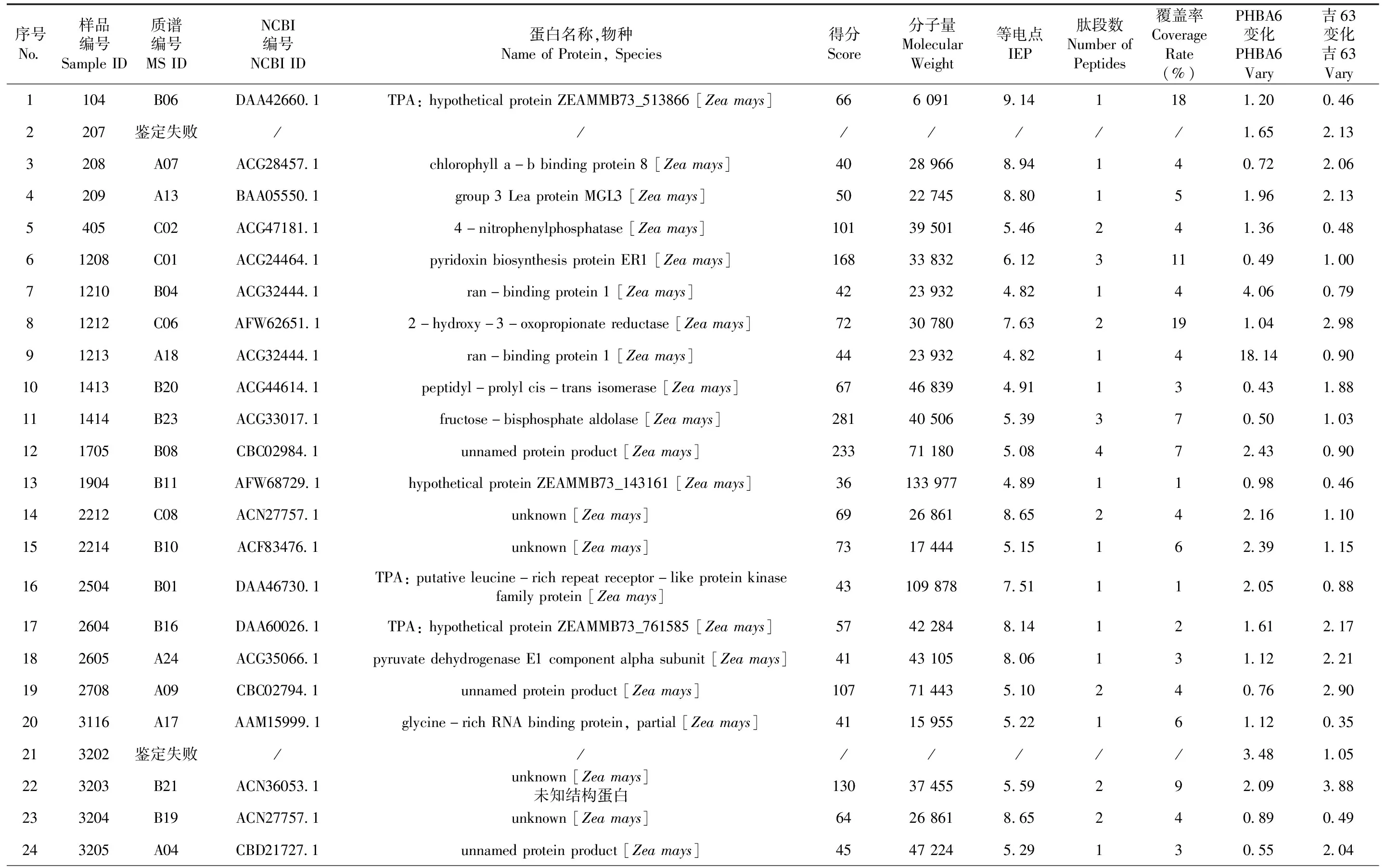
序号No.样品编号SampleID质谱编号MSIDNCBI编号NCBIID蛋白名称,物种NameofProtein,Species得分Score分子量MolecularWeight等电点IEP肽段数NumberofPeptides覆盖率CoverageRate(%)PHBA6变化PHBA6Vary吉63变化吉63Vary1104B06DAA42660.1TPA:hypotheticalproteinZEAMMB73_513866[Zeamays]6660919.141181.200.462207鉴定失败///////1.652.133208A07ACG28457.1chlorophylla-bbindingprotein8[Zeamays]40289668.94140.722.064209A13BAA05550.1group3LeaproteinMGL3[Zeamays]50227458.80151.962.135405C02ACG47181.14-nitrophenylphosphatase[Zeamays]101395015.46241.360.4861208C01ACG24464.1pyridoxinbiosynthesisproteinER1[Zeamays]168338326.123110.491.0071210B04ACG32444.1ran-bindingprotein1[Zeamays]42239324.82144.060.7981212C06AFW62651.12-hydroxy-3-oxopropionatereductase[Zeamays]72307807.632191.042.9891213A18ACG32444.1ran-bindingprotein1[Zeamays]44239324.821418.140.90101413B20ACG44614.1peptidyl-prolylcis-transisomerase[Zeamays]67468394.91130.431.88111414B23ACG33017.1fructose-bisphosphatealdolase[Zeamays]281405065.39370.501.03121705B08CBC02984.1unnamedproteinproduct[Zeamays]233711805.08472.430.90131904B11AFW68729.1hypotheticalproteinZEAMMB73_143161[Zeamays]361339774.89110.980.46142212C08ACN27757.1unknown[Zeamays]69268618.65242.161.10152214B10ACF83476.1unknown[Zeamays]73174445.15162.391.15162504B01DAA46730.1TPA:putativeleucine-richrepeatreceptor-likeproteinkinasefamilyprotein[Zeamays]431098787.51112.050.88172604B16DAA60026.1TPA:hypotheticalproteinZEAMMB73_761585[Zeamays]57422848.14121.612.17182605A24ACG35066.1pyruvatedehydrogenaseE1componentalphasubunit[Zeamays]41431058.06131.122.21192708A09CBC02794.1unnamedproteinproduct[Zeamays]107714435.10240.762.90203116A17AAM15999.1glycine-richRNAbindingprotein,partial[Zeamays]41159555.22161.120.35213202鉴定失败///////3.481.05223203B21ACN36053.1unknown[Zeamays]未知结构蛋白130374555.59292.093.88233204B19ACN27757.1unknown[Zeamays]64268618.65240.890.49243205A04CBD21727.1unnamedproteinproduct[Zeamays]45472245.29130.552.04
续表2 差异相应蛋白质点信息
Table 2 Statistical table of corresponding protein point information
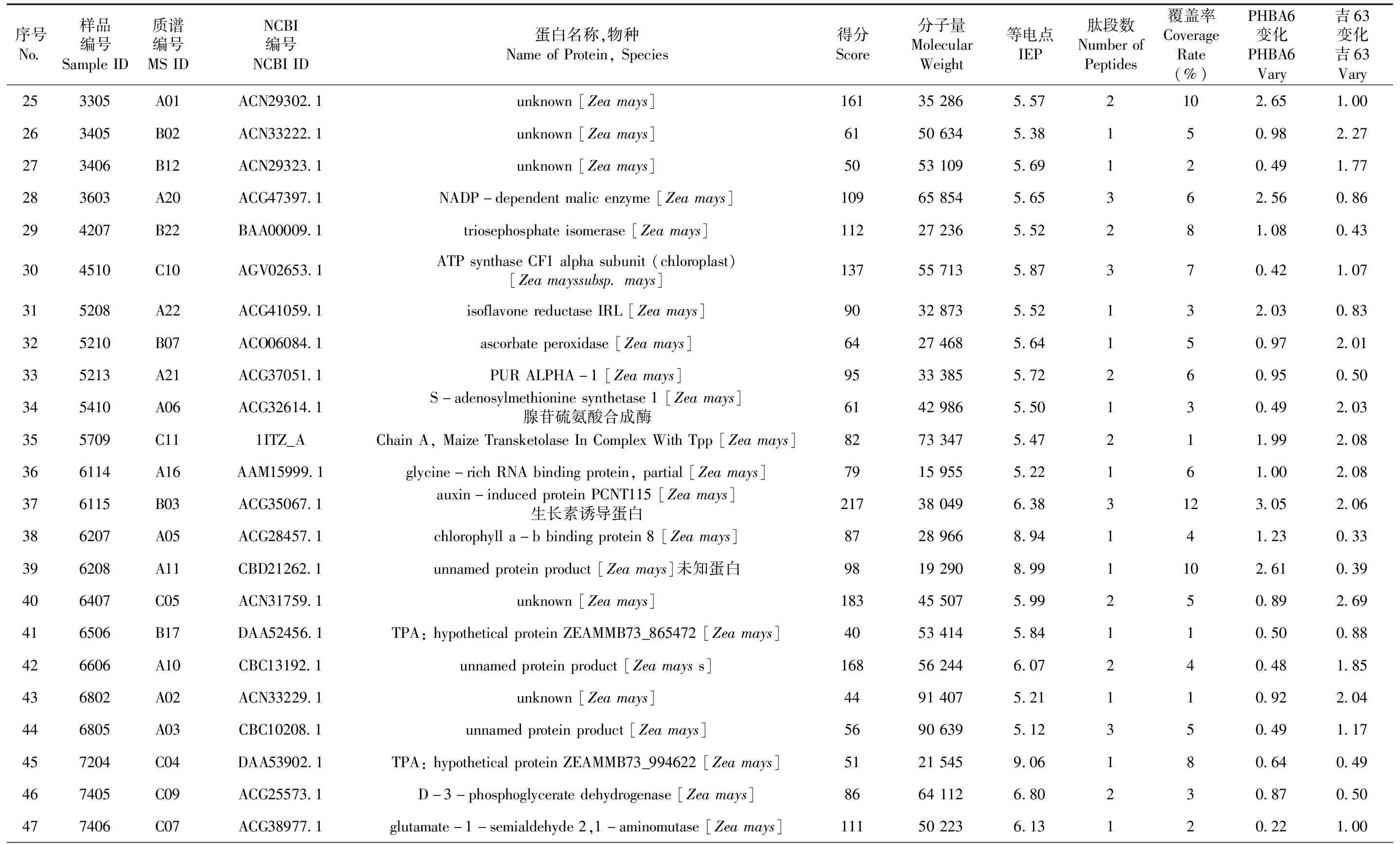
序号No.样品编号SampleID质谱编号MSIDNCBI编号NCBIID蛋白名称,物种NameofProtein,Species得分Score分子量MolecularWeight等电点IEP肽段数NumberofPeptides覆盖率CoverageRate(%)PHBA6变化PHBA6Vary吉63变化吉63Vary253305A01ACN29302.1unknown[Zeamays]161352865.572102.651.00263405B02ACN33222.1unknown[Zeamays]61506345.38150.982.27273406B12ACN29323.1unknown[Zeamays]50531095.69120.491.77283603A20ACG47397.1NADP-dependentmalicenzyme[Zeamays]109658545.65362.560.86294207B22BAA00009.1triosephosphateisomerase[Zeamays]112272365.52281.080.43304510C10AGV02653.1ATPsynthaseCF1alphasubunit(chloroplast)[Zeamayssubsp.mays]137557135.87370.421.07315208A22ACG41059.1isoflavonereductaseIRL[Zeamays]90328735.52132.030.83325210B07ACO06084.1ascorbateperoxidase[Zeamays]64274685.64150.972.01335213A21ACG37051.1PURALPHA-1[Zeamays]95333855.72260.950.50345410A06ACG32614.1S-adenosylmethioninesynthetase1[Zeamays]腺苷硫氨酸合成酶61429865.50130.492.03355709C111ITZ_AChainA,MaizeTransketolaseInComplexWithTpp[Zeamays]82733475.47211.992.08366114A16AAM15999.1glycine-richRNAbindingprotein,partial[Zeamays]79159555.22161.002.08376115B03ACG35067.1auxin-inducedproteinPCNT115[Zeamays]生长素诱导蛋白217380496.383123.052.06386207A05ACG28457.1chlorophylla-bbindingprotein8[Zeamays]87289668.94141.230.33396208A11CBD21262.1unnamedproteinproduct[Zeamays]未知蛋白98192908.991102.610.39406407C05ACN31759.1unknown[Zeamays]183455075.99250.892.69416506B17DAA52456.1TPA:hypotheticalproteinZEAMMB73_865472[Zeamays]40534145.84110.500.88426606A10CBC13192.1unnamedproteinproduct[Zeamayss]168562446.07240.481.85436802A02ACN33229.1unknown[Zeamays]44914075.21110.922.04446805A03CBC10208.1unnamedproteinproduct[Zeamays]56906395.12350.491.17457204C04DAA53902.1TPA:hypotheticalproteinZEAMMB73_994622[Zeamays]51215459.06180.640.49467405C09ACG25573.1D-3-phosphoglyceratedehydrogenase[Zeamays]86641126.80230.870.50477406C07ACG38977.1glutamate-1-semialdehyde2,1-aminomutase[Zeamays]111502236.13120.221.00
续表2 差异相应蛋白质点信息
Table 2 Statistical table of corresponding protein point information

序号No.样品编号SampleID质谱编号MSIDNCBI编号NCBIID蛋白名称,物种NameofProtein,Species得分Score分子量MolecularWeight等电点IEP肽段数NumberofPeptides覆盖率CoverageRate(%)PHBA6变化PHBA6Vary吉63变化吉63Vary487512B24AGV02653.1ATPsynthaseCF1alphasubunit(chloroplast)[Zeamayssubsp.mays]42557135.87251.070.49497603A15AAD10504.1NADP-malicenzyme[Zeamays]61735526.60111.062.90507711B09AAK91502.1NADP-dependentmalicenzyme[Zeamays]104712116.46341.112.03518204A12AAB04687.1histoneH2A[Zeamays]461641710.59150.852.06528310C03ACN32075.1unknown[Zeamays]244384086.374101.142.49538405B13ACN31389.1unknown[Zeamays]108501756.55122.181.21548407A14P24825.1RecName:Full=ChalconesynthaseC2;AltName:Full=Naringenin-chalconesynthaseC267435676.33240.662.05558408A19P24825.1RecName:Full=ChalconesynthaseC2;AltName:Full=Naringenin-chalconesynthaseC296435676.33241.192.17568409B15ACG32271.1GDP-mannose3,5-epimerase1[Zeamays]296433285.944131.103.76379301B18P08440.1RecName:Full=Fructose-bisphosphatealdolase,cytoplasmicisozyme65390367.52142.071.06589303B05ACG32359.1auxin-inducedproteinPCNT115[Zeamays]108378447.62240.471.60599502A08DAA45489.1TPA:hypotheticalproteinZEAMMB73_259316[Zeamays]40469179.79140.652.38
2.2 差异表达蛋白功能分类
2.2.1 GO功能分类
研究表明,强耐旱系PHBA6中差异蛋白参与的生物学过程主要包括代谢物和能量前体合成、核苷酸代谢、丙酮酸代谢过程、含吡啶化合物的复合代谢过程、氧化还原辅酶代谢过程和蛋白的膜定位等;弱耐旱系吉63中差异蛋白参与的生物学过程主要有蛋白翻译调控、细胞蛋白质及氨基酸代谢过程的调控、含硫化合物的合成与代谢过程、半胱氨酸的生物合成及代谢过程和光合作用等。细胞组分分类显示二者中的差异蛋白都与叶绿体及其结构相关。强耐旱系PHBA6中差异蛋白参与的分子功能分类主要包括二磷酸果糖醛酶、乙醛裂解酶、二氨基庚酸转氨酶、葡萄糖磷酸腺苷转移酶及碳酸脱水酶等一些酶的活性;而弱耐旱系吉63中差异蛋白参与的分子功能主要是3-羟基丁酸脱氢酶的活性、poly RNA 的结合、转羟乙醇酶活性和GDP的解离抑制剂活性等。在弱耐系吉63中细胞代谢过程是被促进的,在吉63中基因表达调控、细胞大分子生物合成调控,被抑制的。二者差异蛋白的细胞组分分类一致,而在生物学过程及分子功能分类上相差较大。图2

注:a图为PHBA6品种干旱处理前后差异蛋白的GO功能分析结果;b图为吉63品种干旱处理前后差异蛋白的GO功能分析结果
Note:a shows the GO function analysis results of different proteins of PHBA6 varieties before and after drought treatment.b shows the GO function analysis results of different proteins before and after drought treatment
图2 差异蛋白GO功能
Fig.2 FunctionaLAnalysis of differentiaLProtein GO
2.2.2 KEGG通路
研究表明,PHBA6和吉63 2个自交系的差异蛋白通路基本一致,大部分蛋白由于功能未知,导致信号通路不明确。在强耐旱系PHBA6中的差异蛋白中功能未知蛋白占75%,参与内质网蛋白的加工的蛋白占8%,参与氮代谢和硫胺素代谢的蛋白占4%。在弱耐旱系吉63中的差异蛋白中功能未知蛋白占82%,参与氮代谢、硫胺素代谢、黄酮类化合物合成和光合天线蛋白均占3%。
在KEGG通路分析中,弱耐旱系吉63中的差异蛋白主要参与到下图所示的通路中,最为显著的是Thiamine metabolism(硫胺代谢蛋白)、Photosynesis-antenna proteins(光合作用-天线蛋白质)和Flavonoid biosynthesis(类黄酮生物合成蛋白),这3个代谢过程对植物抵御非生物逆境有重要的作用。图3

注:a图为PHBA6品种干旱处理前后差异蛋白的KEGG通路分析结果;b图为吉63品种干旱处理前后差异蛋白的KEGG通路分析结果。
Note:a shows the KEGG pathway analysis results of different proteins in PHBA6 varieties before and after drought treatment.bshows the KEGG pathway analysis results of different proteins before and after drought treatment.
图3 差异蛋白KEGG通路
Fig.3 Analysis of differentiaLProtein KEGG pathway.
2.2.3 PPI蛋白互作网络
研究表明,PHBA6品种中差异蛋白形成了10个功能组,主要涉及氨基糖和核苷酸糖代谢(3个蛋白)、淀粉和蔗糖的代谢(3个蛋白)、氮素代谢(2个蛋白)、剪接体(6个蛋白)、内质网蛋白加工(6个蛋白)、硫胺素代谢(2个蛋白)、果糖和甘露糖代谢(6个蛋白)、葡聚糖(6个蛋白)、磷酸戊糖途径(6个蛋白)和光合器官的碳固定(6个蛋白)等。吉63品种中差异蛋白也形成了10个功能组,主要涉及内质网蛋白加工(4个蛋白)、内吞作用(4个蛋白)、剪接体(4个蛋白)、硫胺素代谢(2个蛋白)、类黄酮合成(3个蛋白)、生物钟(3个蛋白)、氮素代谢(2个蛋白)、谷胱甘肽代谢(3个蛋白)、抗坏血酸和醛糖代谢(3个蛋白)和光合天线蛋白(4个蛋白)等。图4

注:a图为PHBA6品种差异的蛋白互作网络;b图为吉63品种差异蛋白的互作网络
Note:a shows the protein interaction network of PHBA6 varieties.b shows the interaction network of different proteins of J63
图4 差异蛋白互作网络
Fig.4 DifferentiaLProtein interaction network
3 讨 论
研究结果表明,干旱胁迫10 d和0(正常灌溉CK)影响PHBA6和吉63花器官分化期雄穗的蛋白质组成变化差异较大。一些功能蛋白质的表达受到干旱胁迫的诱导上调或者抑制下调。鉴定出差异表达丰度达2.0倍以上的蛋白质点共有59个,强耐旱系PHBA6中有26个,弱耐旱系吉63中有37个,在强耐旱系PHBA6与弱耐系吉63中都表达且差异显著的蛋白质点有4个。
GO功能分类分析,强耐旱系PHBA6中差异蛋白参与的分子功能分类主要包括二磷酸果糖醛酶、葡萄糖磷酸腺苷转移酶及碳酸脱水酶等一些酶的活性。KEGG通路分析,弱耐旱系吉63中的差异蛋白主要参与到下所示的通路中,最为显著的是Thiamine metabolism(硫胺代谢蛋白)、Photosynesis-antenna proteins(光合作用-天线蛋白质)和Flavonoid biosynthesis(类黄酮生物合成蛋白),这3个代谢过程对植物抵御非生物逆境有重要的作用。
果糖-l,6-二磷酸醛缩酶是生物体内碳代谢及糖代谢途径中的关键酶之一,也是卡尔文循环的重要限速酶之一,它催化3-磷酸甘油醛和磷酸二羟丙酮的醇醛缩合反应可逆的生成果糖-1,6-二磷酸。该酶既参与了糖酵解和糖异生过程,又参与了磷酸戊糖途径和卡尔文循环,直接影响植物体内蔗糖和淀粉的积累,为生物体物质合成代谢提供能量ATP和底物,果糖-1,6-二磷酸醛缩酶对细胞生命活动起到至关重要的作用[12]。硫胺代谢蛋白、光合作用-天线蛋白质和类黄酮生物合成蛋白等能够有效地减弱干旱胁迫下植物蛋白氧化损伤程度,从而增强玉米抵御干旱的能力,以上研究结果与刘振斌等研究结果一致[12-15]。
4 结 论
以强耐旱系PHBA6和旱敏感系吉63开花期干旱胁迫处理下的小花蛋白质组为研究对象,采用质谱分析和生物信息学分析,在强耐旱系PHBA6中二磷酸果糖醛酶、葡萄糖磷酸腺苷转移酶及碳酸脱水酶等一些酶的活性增强,所控制的蛋白上调表达,增强了玉米抵御干旱的能力;而在旱敏感系吉63中,硫胺代谢蛋白、光合作用-天线蛋白质和类黄酮生物合成蛋白等能够有效地减弱干旱胁迫下植物蛋白氧化损伤程度,增强玉米抵御干旱的能力。
