基于纳米孔测序技术的PNA/λDNA测序精确度研究
2020-02-07熊珊珊
罗 帆,熊珊珊
(东南大学机械工程学院,江苏 南京 211189)
随着科学技术的发展,人们对DNA结构及其携带的遗传信息的深入研究,促使DNA测序技术不断地变化,成为生命科学的重要研究领域,极大地推动了生物工程、医疗技术以及其他学科的发展。第一代基因测序方法是以Sanger的双脱氧链终止法[1]为核心的化学降解法,是人类基因组技术的核心测序方法,其测序精度高达99.999%,但是高成本和低通量的缺点使其无法实现大规模的商业应用。为了降低测序成本以及提高测序基因的通量,人类进入了第二代基因测序时代。第二代基因测序方法以Roche公司454技术、illumina公司的Solexa和Hiseq技术以及ABI公司的Solid技术为代表,其主要特点是高通量和读取速度快,一次可以对几十万条乃至几百万条DNA分子的碱基进行测序,它促进了人类对物种转录组测序以及基因组深度测序方法的发展,但是其试剂价格昂贵,使得第二代基因测序的成本高达几十万美元。近年来,基于单分子纳米孔技术的第三代测序方法应运而生,其主要是通过分辨4种碱基结构的细微差异而导致不同碱基在通过纳米孔时产生不同的离子阻塞电流来进行测序,测序过程不需要试剂,且测序速度比一、二代测序方法快,有望进一步降低测序成本,从而改进人类由基因缺陷引起疾病的治疗方法[2-5]。为了证明基于单分子纳米孔的第三代测序技术的精准性[6-7],本文利用字符串匹配算法模拟单分子探针PNA(肽核酸)与λDNA通过双层纳米孔芯片匹配实验来计算单分子探针读取λDNA的精确度。
1 匹配算法
匹配算法的基本原理:把λDNA的碱基信息看成一个长字符串,PNA探针看成一个短字符串,把PNA探针与λDNA的匹配变成短字符串与长字符串的匹配。
图1所示为PNA/λDNA模拟实验的流程图,图中V为PNA探针字符串,B为λDNA的字符串,C为探针读取λDNA碱基位置数组,K为探针首个碱基读取的λDNA碱基有效位置数组,Q为所有PNA探针读取有效位置数组,P为各类探针匹配个数数组,lb为λDNA的长度(单位:b),lc为探针读取的位置长度(单位:b),lq为探针读取的有效位置长度(单位:b),per为探针库所有探针读取λDNA的精确度。
在进行字符串匹配时,首先要进行的是探针的自动输入。由于探针库S的大小与探针组成碱基个数n成幂次方(S=4n)关系,故当n值较大时,探针库的容量就会很大,自动输入探针就十分必要。在MATLAB中,使用函数comb,只要输入组成PNA探针的碱基个数及其碱基种类,就能直接生成一个包含探针所有类型的字符数组,只需要对字符数组中的单个字符串进行调用,就可实现PNA探针的自动输入,如图1中Ⅰ所示。其次在探针输入之后,进行短字符串PNA探针与长字符串λDNA的匹配。使用字符串处理函数strncmp比较PNA探针碱基信息与λ DNA上的碱基片段是否相等,如图1中Ⅱ所示。其匹配过程见表1,表中序号为λDNA中碱基的排列顺序, A,C,G,T分别代表腺嘌呤、胞嘧啶、鸟嘌呤、胸腺嘧啶。
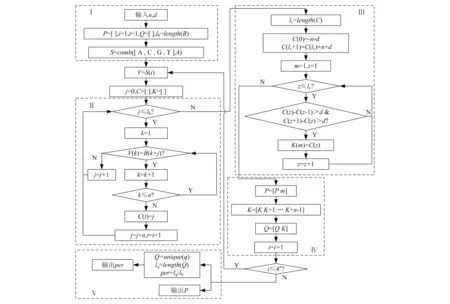
图1 PNA/λDNA模拟实验的流程图

表1 字符串匹配过程
如果PNA探针碱基信息与λDNA上读取的碱基信息相同,则输入当前PNA探针首个碱基在λDNA上的位置,然后PNA探针往后移动其碱基个数大小的位置再继续与λDNA上的碱基片段进行匹配;如果不同,PNA探针往后移动一个位置继续与λDNA上的碱基片段进行匹配,直至PNA探针最后一个碱基对应λDNA的最末端位置或超出最末端时,该种探针匹配过程结束,接着进行下一种探针与λDNA的匹配。以此类推,直至探针库中所有的探针与λDNA匹配完,PNA/λDNA模拟实验结束。
由于芯片具有一定的厚度,当前一个PNA探针未完全通过纳米孔通道而后一个PNA探针进入纳米孔通道时,碱基过孔电流信号就会互相干扰,从而无法准确读取出λDNA上相应的碱基片段信息,因此PNA探针之间必须有一定的间隔,满足间距要求的PNA探针的位置为有效位置。在有效位置筛选阶段,可以对PNA探针输出首个碱基位置与相邻的PNA探针输出的位置进行比较,即通过芯片的厚度算出对应的碱基数目间距d,当PNA探针与其相邻PNA探针的间距大于d时,PNA探针读取的位置才为有效位置,如图1中Ⅲ所示。有效位置筛选的具体流程见表2,表中序号为λDNA中碱基的排列顺序。

表2 有效位置筛选过程
在表2中,取有效间距d=15 b(b,碱基),PNA探针的碱基序列为GGG,由于与λDNA匹配的PNA探针序号②和③的间距为12 b,两者通过芯片时产生的信号会重叠而无法区分,故只有序号为①、④和⑤的PNA探针所读取的λDNA的位置为有效位置。
得到单种PNA探针首个碱基读取λDNA的位置之后,生成PNA探针匹配λDNA的个数数组P,如图1中Ⅳ所示。
将所有种类的PNA探针读取的有效位置存放在一个向量中,然后进行数据处理,剔除掉重复的数据,向量中剩下的数据即为PNA探针所有碱基读取的λDNA碱基信息,再与λDNA的碱基总数进行对比,结果即为PNA探针读取λDNA的精确度,如图1中V所示。
2 实验结果及分析
分别对3merPNA探针和5merPNA探针与λDNA进行匹配实验,测试其匹配λDNA的精确度。
由于PNA探针由腺嘌呤(A)、胞嘧啶(C)、鸟嘌呤(G)、胸腺嘧啶(T)4种碱基组成,为了区别不同探针,采用四进制对其进行编码,顺序依次为A,C,G,T。例如图2中横坐标2代表PNA探针的碱基序列AAC,图3中横坐标10代表PNA探针的碱基序列AAAGC。图2和图3分别为间隔d为12 b时,3merPNA探针和5merPNA探针与λDNA匹配数量直方图。
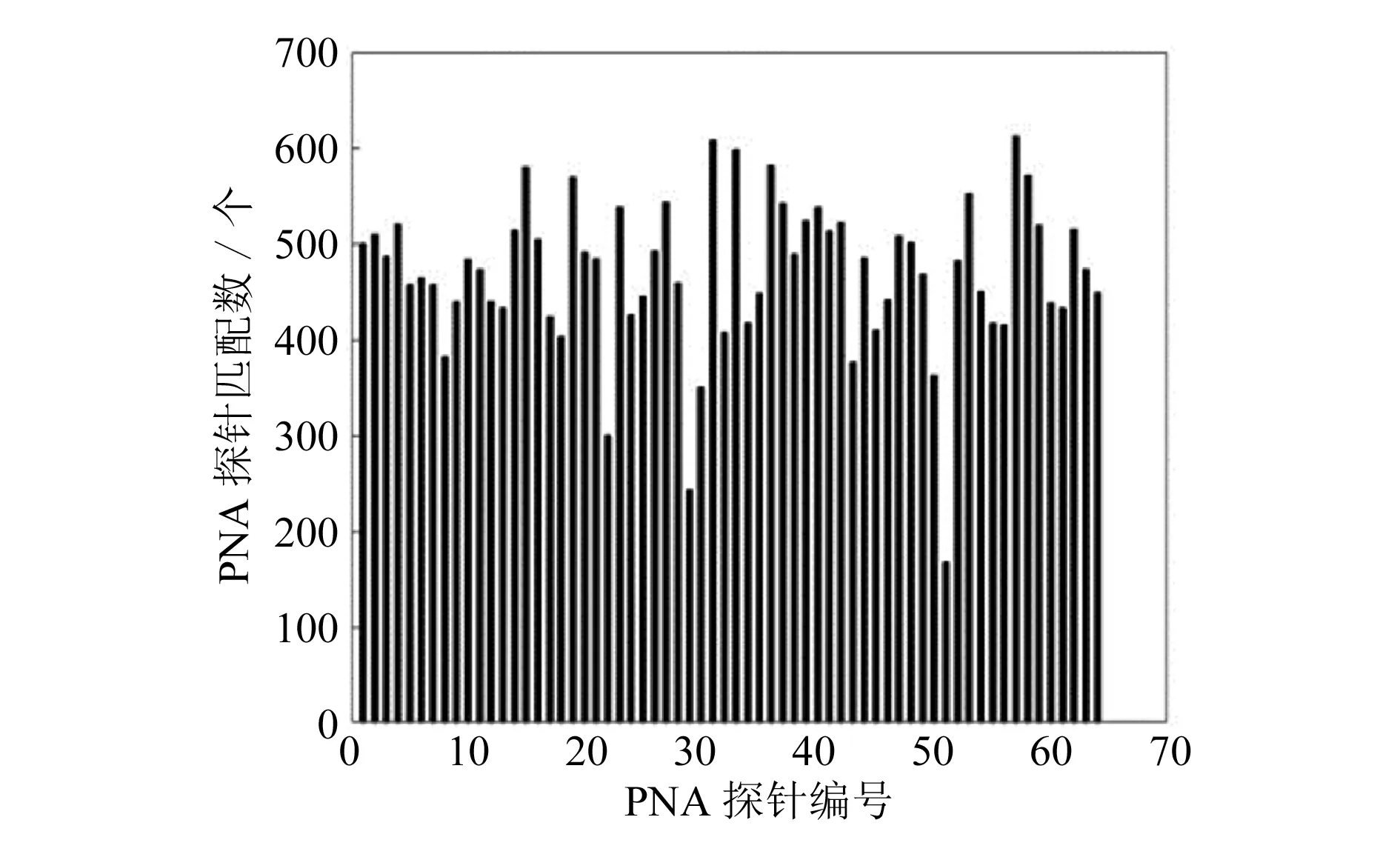
图2 3merPNA与λDNA匹配数量直方图

图3 5merPNA与λDNA匹配数量直方
从图2和图3可以得知,当PNA探针碱基个数相同时,不同种类的PNA探针匹配的λDNA的个数相差很多,PNA探针碱基个数越多,能够与λDNA匹配的数目就越少。为了了解PNA探针匹配λDNA的精确度per与PNA探针间距d之间的关系,取d=0~50进行计算,结果如图4所示。
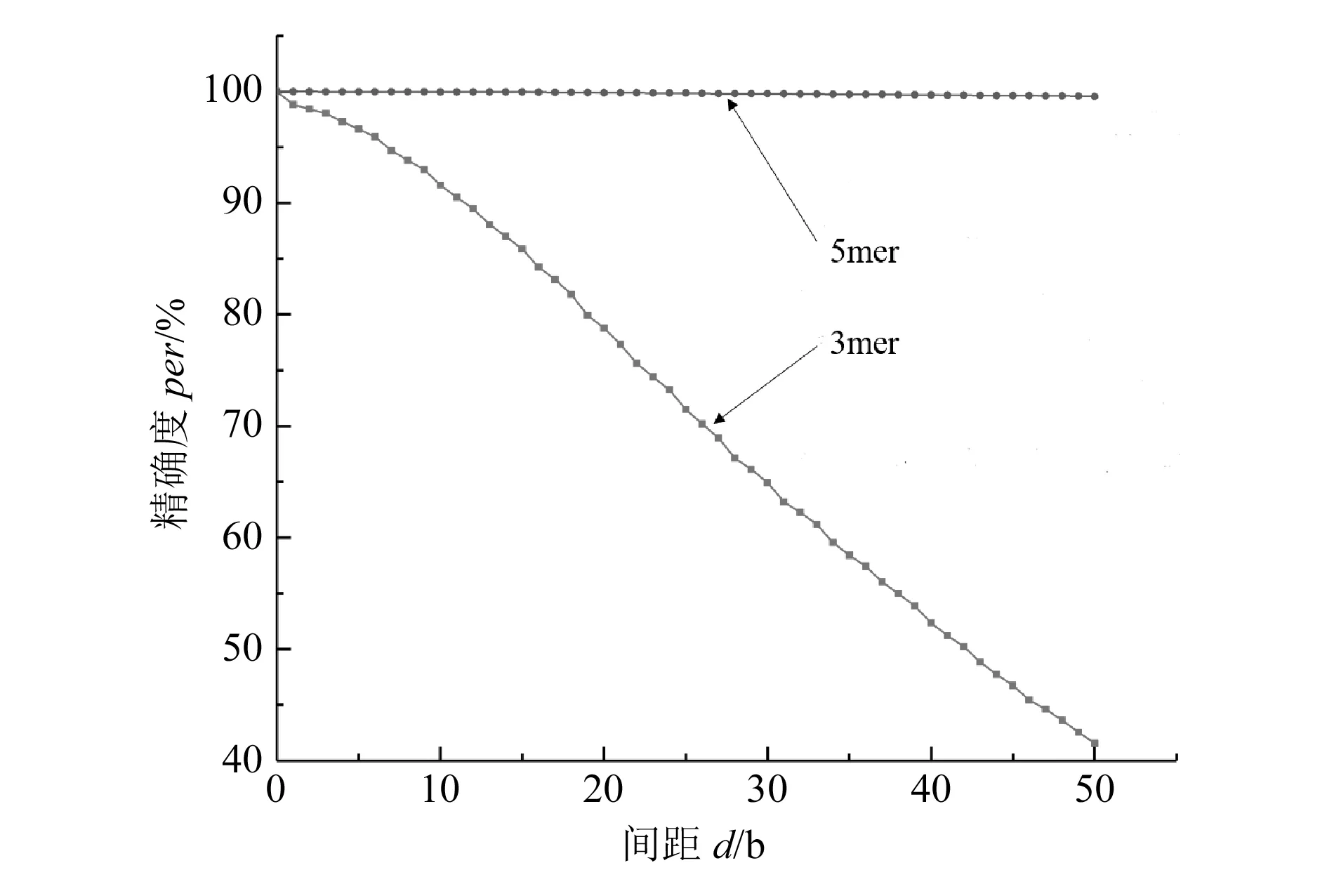
图4 精确度per与间距d关系图
由图4可知,相邻2个3merPNA探针的间距d在0~50时,精确度per随着间距d的增大而减少,从98.75%下降到42.13%;而5merPNA探针的精确度受d的变化影响不大,仅从99.99%下降到了99.50%。
3 结束语
本文通过字符串匹配算法来模拟PNA探针与λDNA匹配过程,模拟实验结果证明了基于单分子纳米孔的第三代测序技术能精确地读取DNA的碱基信息,其精确度可高达99.99%,为第三代基因测序技术提供了一定的理论依据。
