羊传染性脓疱病毒NZ2毒株CBP 蛋白结构分析与表位预测
2019-12-06成伟伟陈伯祥常攀峰赵子惠
成伟伟,陈伯祥,常攀峰,杨 明,赵子惠
(甘肃省畜牧兽医研究所,甘肃平凉 744000)
羊传染性脓疱是由羊传染性脓疱病毒(ORFV)感染山羊和绵羊,在其嘴唇部皮肤形成脓疱或结成厚痂的一种人兽共患传染病[1]。该病主要在3月龄左右的羔羊和部分免疫力下降的成年羊中流行,可导致感染羔羊死亡、成年羊生产性能下降[2]。该病也可感染人,使人的手部皮肤出现溃疡甚至坏死[3-4]。因而该病既影响养羊业健康发展,也威胁人们身体健康。
ORFV 的ORF112基因编码的CBP 蛋白是趋化因子结合蛋白,含有286个氨基酸残基[5]。ORF112基因长约861 bp。CBP 蛋白能够竞争性地结合趋化因子,阻止G 蛋白偶联受体与趋化因子结合,从而抑制宿主的免疫反应。有研究[6]表明,敲除ORF112基因,对ORFV 的致病性有明显影响。然而其具体机制并不清楚,CBP 蛋白是否还具有其他功能也不清楚。所以,本研究以ORFV NZ2毒株的CBP 蛋白氨基酸序列为研究对象,应用生物信息学方法,对CBP 蛋白的理化特性、亲水性、跨膜区、信号肽、二级结构、三级结构、B 细胞表位和T 细胞表位进行预测分析,以期为CBP 蛋白的功能研究和ORFV 的致病机制研究提供理论依据。
1 材料与方法
1.1 氨基酸序列
选 取GenBank 中ORFV NZ2毒株的CBP 基因序列(序列号为ABA00630),运用Editseq 软件翻译成CBP 蛋白的氨基酸序列,序列如下:


1.2 方法
运用在线工具ProtParam,分析CBP 蛋白的理化特性[7];利用DNAstar 软件中的KyteDolittle方法,预测CBP 蛋白的亲水性和疏水性;利用在线软件TMHMM 和SingnalIP,预测CBP 蛋白的跨膜区和信号肽[8];利用DNAstar 软件,预测分析CBP 蛋白的二级结构[9-10];将CBP 蛋白的氨基酸序列提交Swiss-Model,使用RasMol 分析软件构建OVIFNR 蛋白的三级结构模型[11];运用在线ABCpred 工具和DNAstar 软件,预测CBP 蛋白B细胞表位[12-13],并综合分析单参数预测结果,筛选出优势B 细胞表位;分别运用在线服务器nHLAPred 和在线软件SYFPEITHII,预测CBP 蛋白所含的HLA-A*0201、HLA-A*0202、HLA-A*0203三种MHC-I 结合肽段,并结合两种方法预测出的结果,筛选出优势CTL 表位[14];分别使用在线服务器ProPred 和在线软件SYFPEITHI,预测CBP 蛋白所含的HLA-DRB1*0101、HLADRB1*0102、HLA-DRB1*0301三种MHC-II 结合肽段,然后结合两种方法预测出的结果,筛选出优势Th 表位[15]。
2 结果
2.1 CBP 蛋白的理化特性预测分析
CBP 蛋白的理化特性预测分析结果显示,CBP 蛋白的分子式为C1357H2155N367O451S11,相对分子质量为31.179 89 kDa,理论等电点为4.68,半衰期为30 h,稳定系数为43.06,脂肪族指数为81.78,总平均亲水性为-0.383。
2.2 CBP 蛋白的亲疏水性预测分析
CBP 蛋白的亲水性和疏水性预测结果(图1)显示,CBP 为亲水性蛋白,其亲水性区域主要位于19~62、71~90、119~125、132~158、165~184、188~220、242~290位氨基酸区域。
2.3 CBP 蛋白的跨膜区和信号肽预测分析
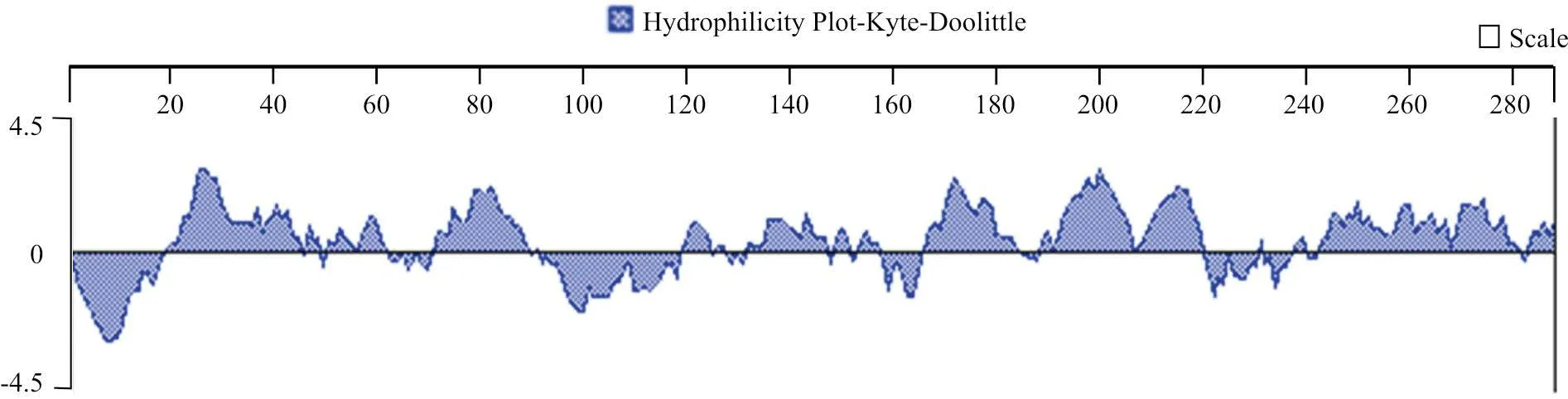
图1 CBP 蛋白亲水性预测结果
CBP 蛋白的跨膜区预测结果(图2)显示,CBP 蛋白无跨膜区,其1~286个氨基酸残基全部在膜外。CBP 蛋白信号肽预测结果(图3)显示,CBP 蛋白存在潜在的信号肽,位于1~15位氨基酸残基处,在16~17位处存在剪切位点。
2.4 CBP 蛋白的二三级结构预测分析

图2 CBP 蛋白跨膜区预测结果
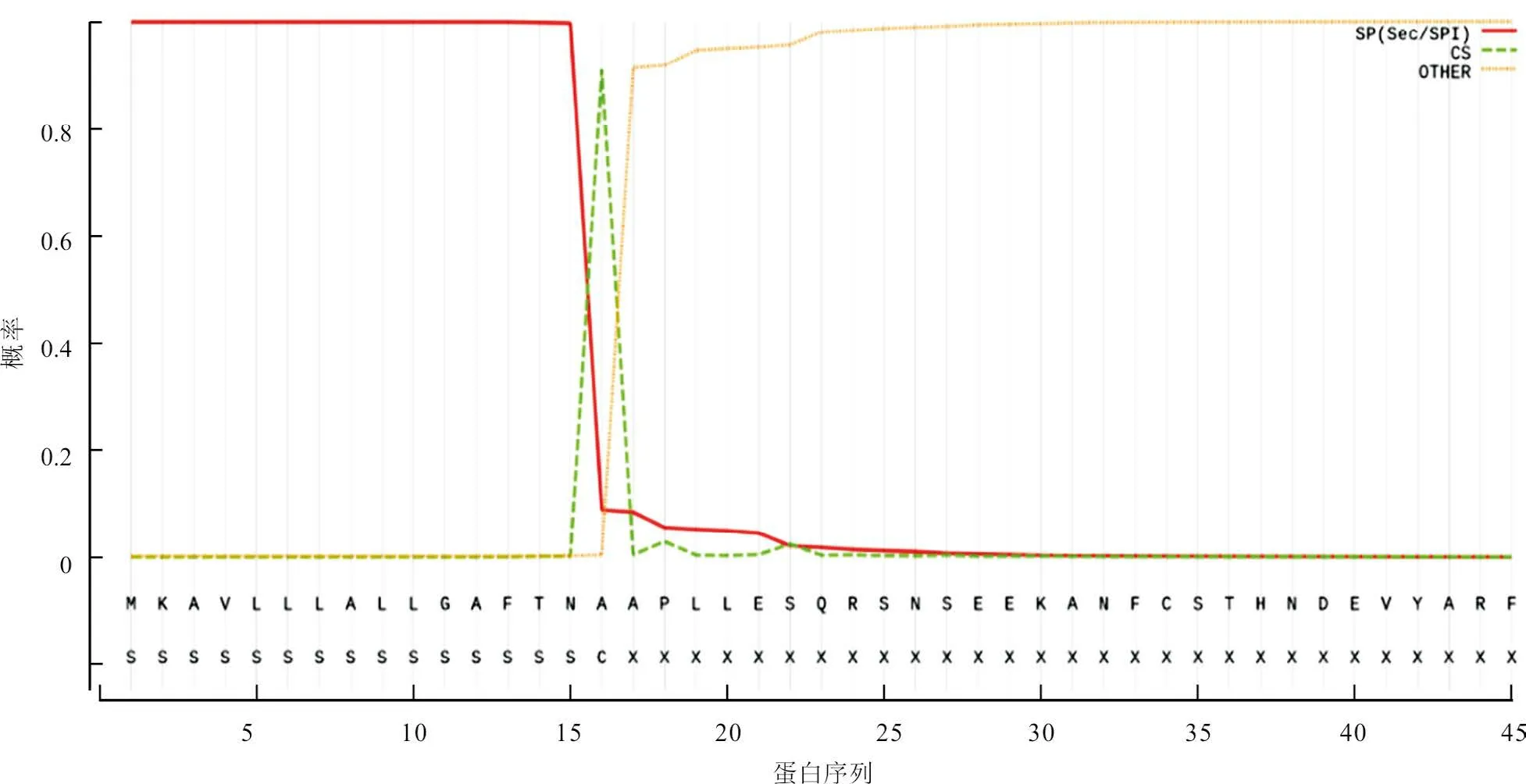
图3 CBP 蛋白信号肽预测结果
CBP 蛋白的二级结构的预测结果(图4)显示:CBP 蛋白的二级结构中主要包括α 螺旋、β 折叠、β 转角和无规卷曲4种类型,所占比重分别为α 螺旋38.11%、β 折叠33.91%、β 转角5.94%、无规卷曲10.48%;CBP 蛋白的α 螺旋主要位于1~24、27~35、39~48、67~95、155~163、235~245、251~253、260~273,β 折叠主要位于49~57、63~66、96~114、122~131、136~143、151~154、182~189、206~212、217~227、229~234、246~250、276~281,β 转角主要位于58~61、146~148、166~169、175~177、198~200,无规卷曲主要位 于36~38、117~121、132~134、170~172、178~181、190~195、201~203、282~284。使用在线软件Swiss-Model 构建的CBP 蛋白三级结构模型如图5所示。
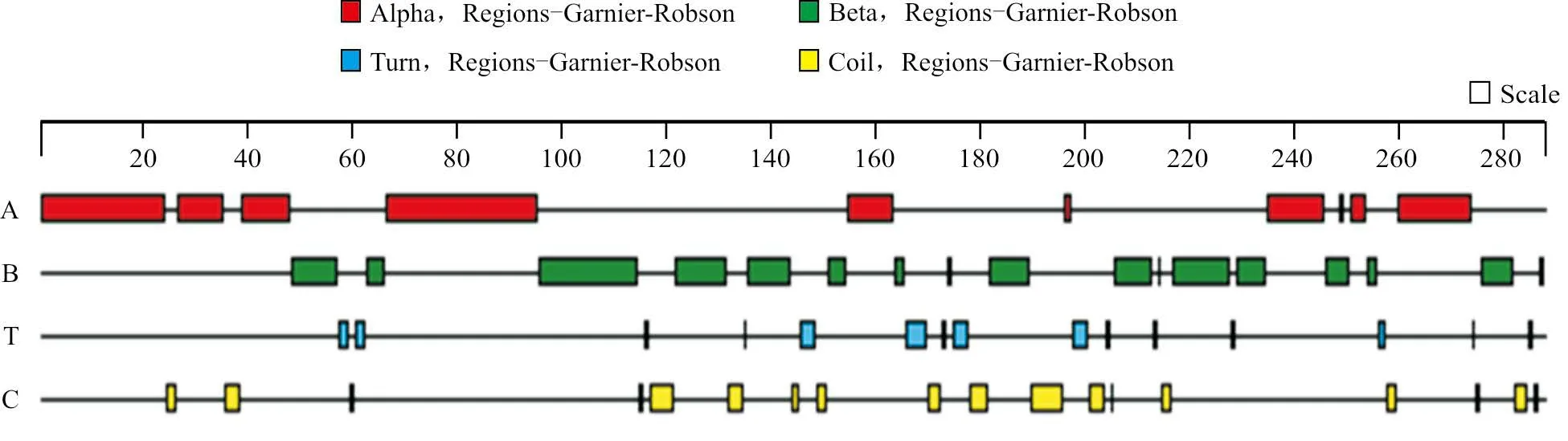
图4 CBP 蛋白二级结构预测结果
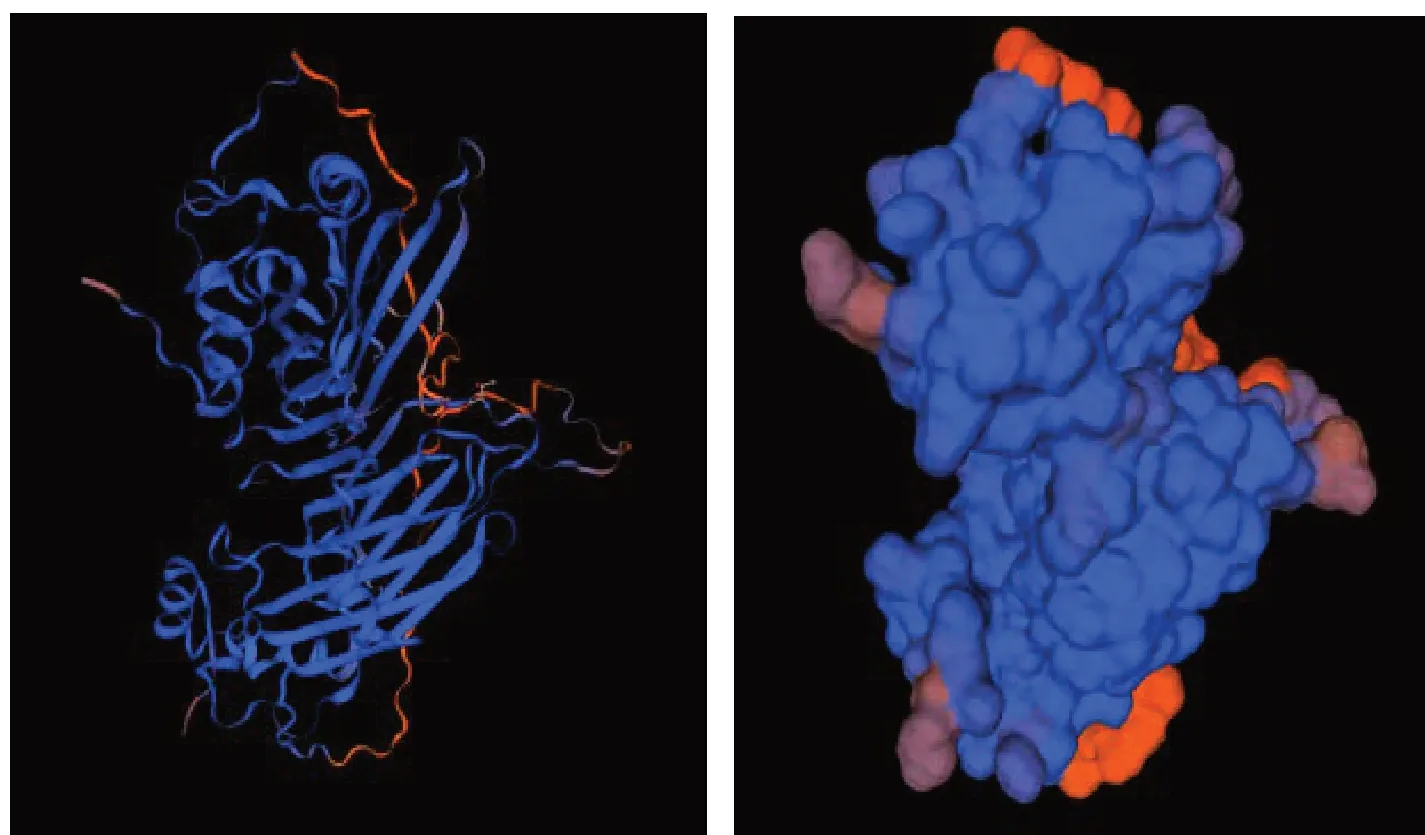
图5 CBP 蛋白三级结构同源建模
2.5 CBP 蛋白的B 细胞表位预测分析
利用DNAstar 软件对CBP 蛋白的柔性区域、抗原指数和表面可及性进行预测,结果如图6所示;运用在线软件ABCpred 预测CBP 蛋白B 细胞表位,结果如表1所示。将β 转角、无规卷曲、亲水性区域、柔韧性区域、抗原指数和表面可及性区域的预测结果作为CBP 蛋白B 细胞表位预测的参数,结合ABCpred 预测结果,综合筛选出CBP 蛋白优势B 细胞表位,结果筛选出CBP 蛋白优势B 细胞表位6个,分别是17~27、21~31、74~84、81~91、118~128、132~142(表2)。
2.6 CBP 蛋白的T 细胞表位预测分析
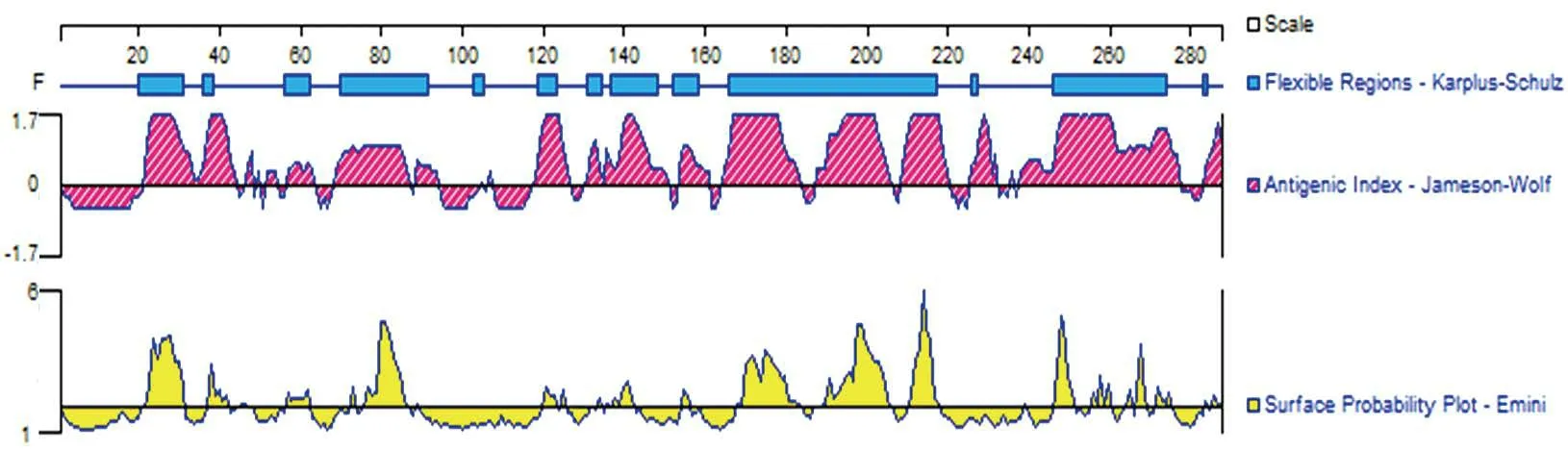
图6 CBP 蛋白的柔韧性区域、抗原指数和表面可及性预测结果
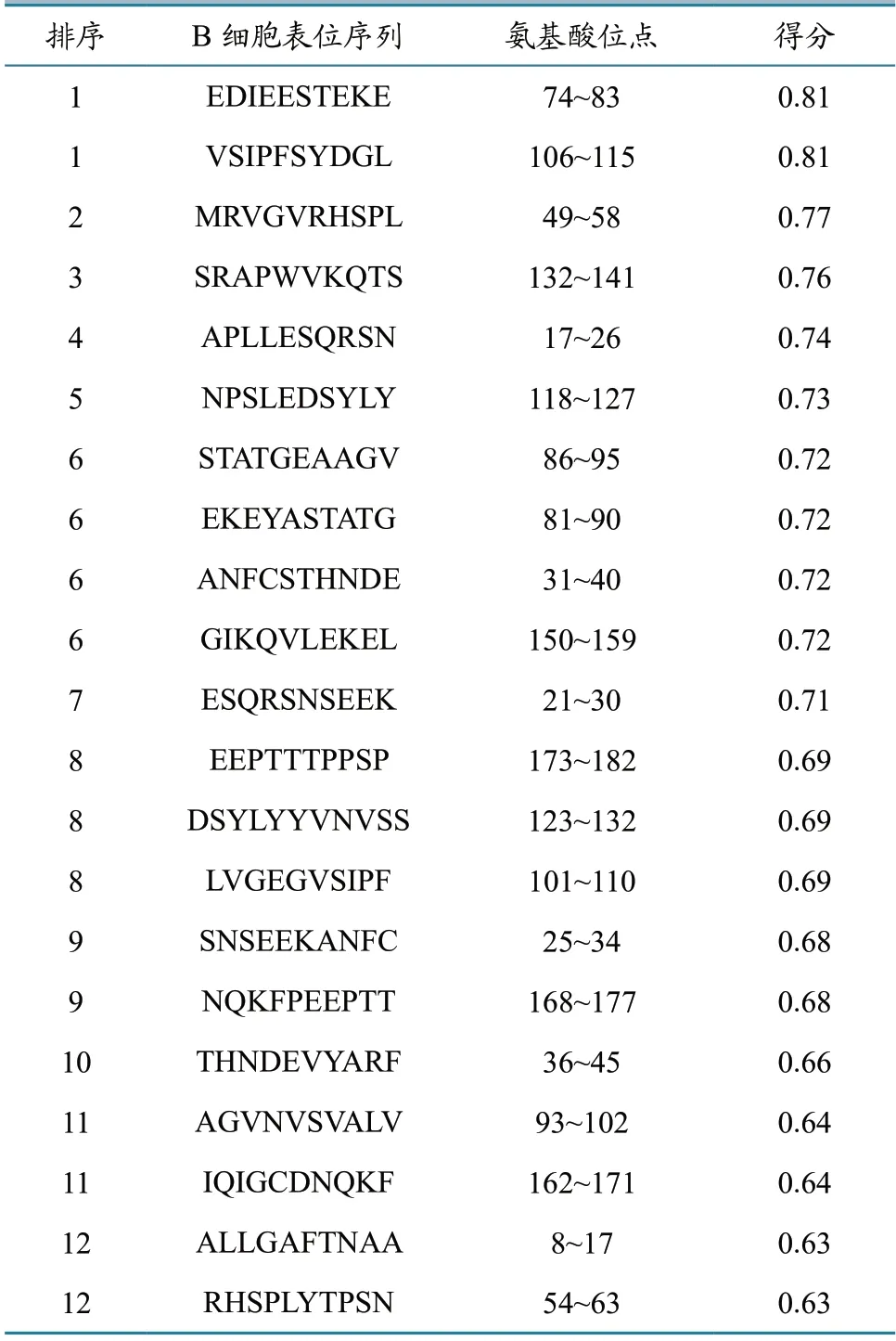
表1 ABCpred 预测CBP 蛋白B 细胞表位
运用在线服务器nHLA-Pred 对CBP 蛋白所含的HLA-A*0201、HLA-A*0202、HLA-A*0203三种MHC-I 结合肽段的预测结果见表3,利用在线软件SYFPEITHII 对CBP 蛋白CTL 表位进行预测,预测结果见表4。结合两种方法的预测结果,筛选出CBP 蛋白的3个优势CTL 表 位,分别为4~13位 的VLLLALLGA、65~74位 的CMLDIEDSV 和120~129位 的SLEDSYLYV。
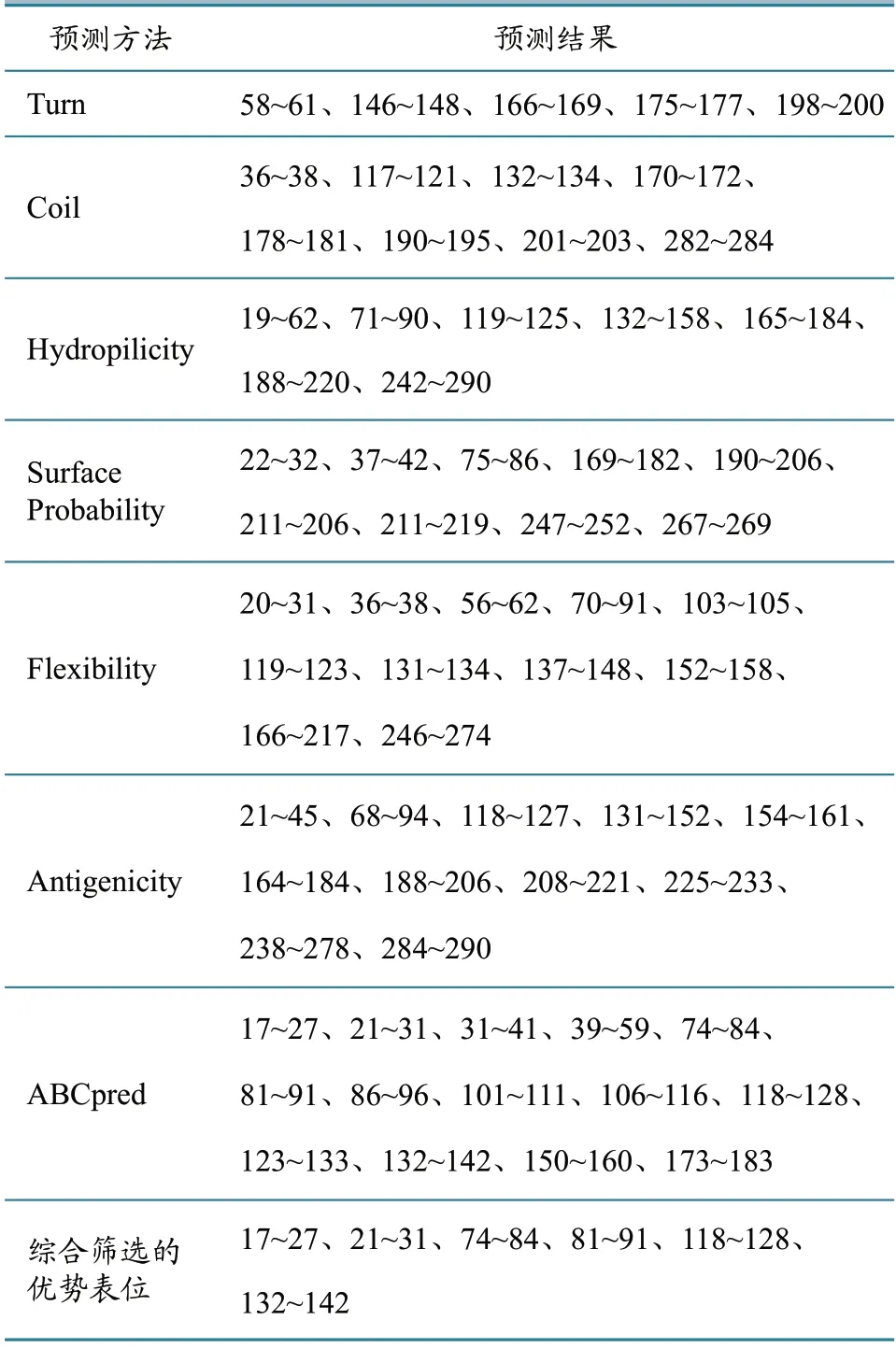
表2 综合筛选CBP 蛋白优势B 细胞表位
使用在线服务器ProPred 预测CBP 蛋白HLA-DRB1*0101、HLA-DRB1*0102和HLADRB1*0301三种MHC-II 结合肽段,未预测出MHC-II 结合肽段;运用在线软件SYFPEITHI 预测CBP 蛋白的MHC-II 结合肽段,也未预测出MHC-II结合肽段。两种预测的结果表明,CBP 蛋白可能不含Th 细胞表位。
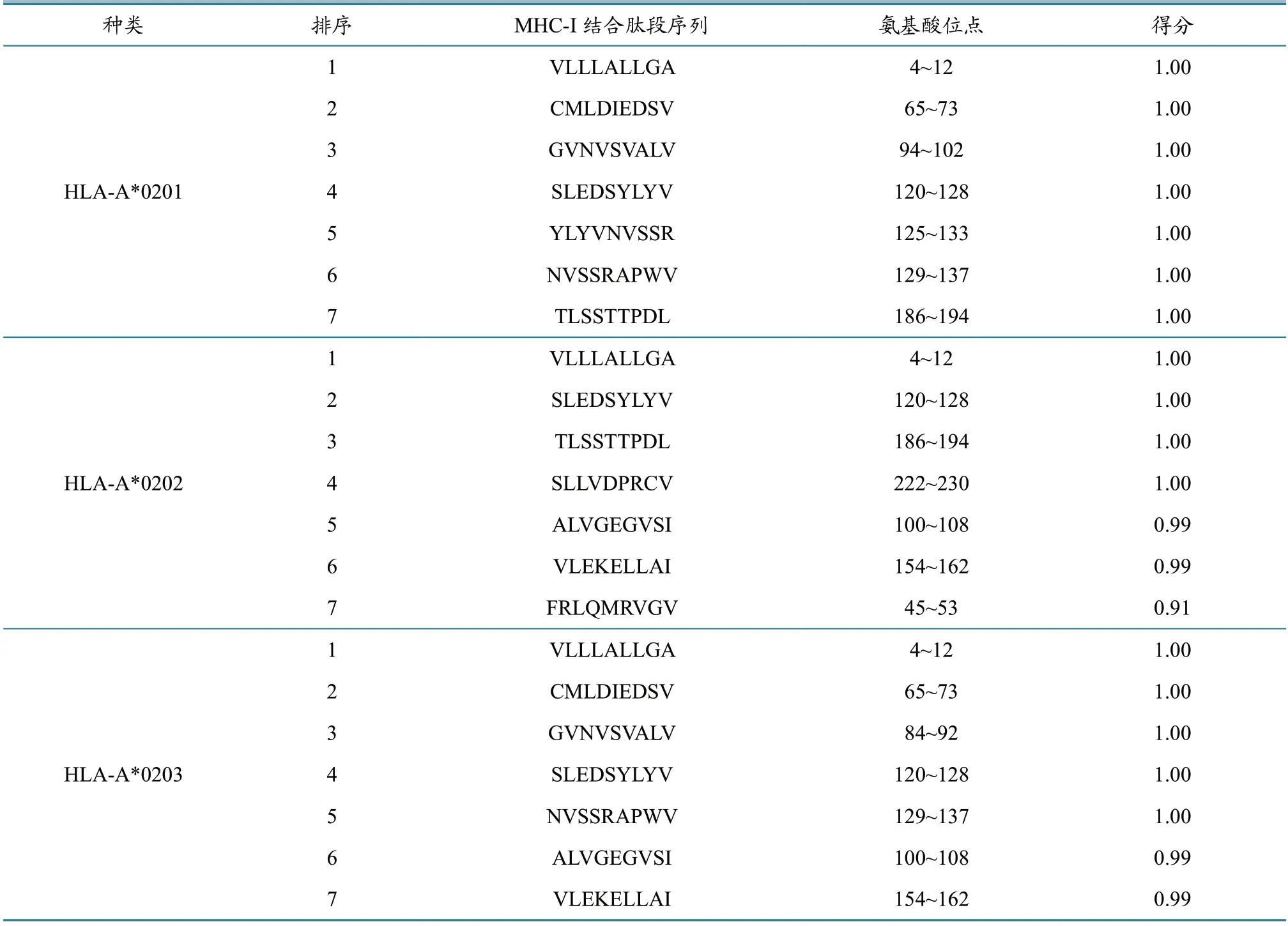
表3 nHLA-Pred 方法对CBP 蛋白CTL 表位的预测结果
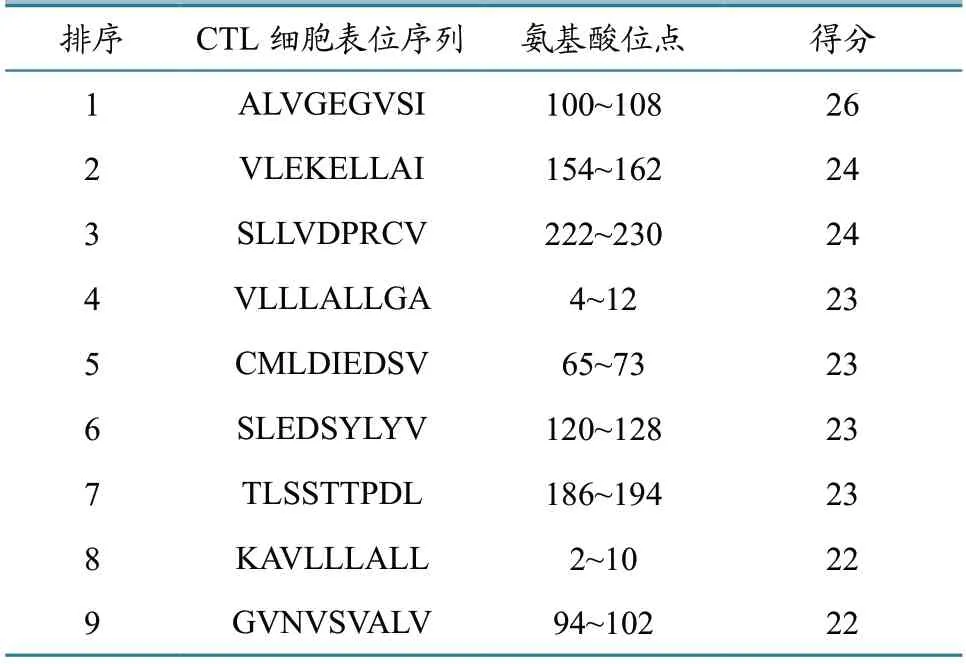
表4 SYFPEITHI 方法对CBP 蛋白CTL 表位预测
3 讨论
通过生物信息学软件预测出CBP 蛋白的理论等电点为4.68。蛋白质所处环境的pH 大于理论等电点时,蛋白质带负电,小于理论等电点时,蛋白质带正电[16-17]。而CBP 蛋白在ORFV 感染宿主的早期表达中,宿主体内的pH 约为7.0左右,所以该蛋白在宿主体内带负电;该蛋白的稳定系数为43.06,说明该蛋白不太稳定。蛋白质的稳定性与其特定的氨基酸序列、剪切加工及空间结构都有密切的关系。蛋白质是否为亲水性蛋白主要取决于其分子表面的电荷量。CBP 蛋白虽为亲水性蛋白,但较强的亲水性区域较少,说明该蛋白形成抗原表位方面的能力不足。在线软件SingnalIP 预测结果表明,CBP 蛋白存在潜在的信号肽,有信号肽的蛋白大多被分泌到膜外发挥作用;而在线软件TMHMM 预测结果表明,CBP 蛋白无跨膜区,1~286个氨基酸残基全部在膜外。这两种预测结果充分说明,CBP 蛋白被运输到膜外发挥作用。
CBP 蛋白的二级结构预测表明,β 转角和无规卷曲所占比重较小,说明该蛋白不易形成抗原表位;而CBP 蛋白的表位预测结果也说明了这一点,CBP 蛋白预测出的细胞表位较少。Swiss-Model 是基于同源建模的方法构建蛋白质三级结构,是目前三级结构预测中最好的预测工具之一,构建出的三级结构具有更高的准确性。蛋白质三维结构的构建有助于对蛋白质功能的认识。本研究构建的CBP蛋白的三维结构形似倒立的哑铃状,上下两部分比较对称。本研究在CBP 蛋白B 细胞表位预测方面,将β 转角、无规卷曲、亲水性区域、柔韧性区域、抗原指数和表面可及性区域的预测结果作为参数,并结合 ABCpred 预测结果筛选CBP 蛋白的B 细胞表位,这样筛选出的CBP 蛋白B 细胞表位具有更高的可信度。在CTL 细胞表位预测方面,结合了nHLA-Pred 和SYFPEITHII 两种方法预测的结果,在Th 细胞表位预测方面,结合了ProPred 和SYFPEITHII 两种方法预测的结果,所以预测出的CBP 蛋白T 细胞表位具有更高的可靠性。本研究筛选出CBP 蛋白优势B 细胞表位6个、CTL 细胞表位3个,未预测出Th 细胞表位。这样CBP 蛋白较少的细胞表位与CBP 蛋白二级结构中β 转角和无规卷曲所占比重较小相互印证。本研究在B 细胞表位预测上与黎摄儿等[18]对ORFV 湖北分离株的CBP 蛋白B 细胞表位预测结果基本一致,但在T 细胞表位预测上差异较大。这可能与ORFV 各毒株间的差异有关。CBP 蛋白未预测出Th 细胞表位,说明该蛋白可能在ORFV 逃避宿主免疫反应过程中发挥了作用。
本研究仅运用生物信息学方法对ORFV NZ2毒株CBP蛋白的结构与细胞表位进行了分析预测,因受条件限制,尚未对得到的结果和结论进行实验验证,但该仍可为CBP 蛋白的功能研究与ORFV的致病机制研究提供一定的参考。
