基于RAPD标记对71个柞蚕品种的聚类分析
2019-07-22刘丹梅李文利王丹丹
刘丹梅 李文利 王丹丹


摘要:采用随机引物扩增多态性DNA(RAPD)的方法,在DNA分子水平上对我国不同地区、不同化性的71个柞蚕品种进行聚类分析。从140条RAPD随机引物中筛选出28条可稳定扩增条带的多态性引物,共扩增出92条多态性条带,占总扩增带数的29%;采用非加权组平均法(unweighted pair-group method with arithmetic means,简称UPGMA)聚类法对试验结果进行分析,构建了71个柞蚕品种的亲缘关系,结果显示,各个地区与不同化性的柞蚕品种之间亲缘关系没有明显区别,遗传类型相互混杂。
关键词:RAPD标记;柞蚕;UPGMA;聚类分析;亲缘关系
中图分类号: S885.1 文献标志码: A 文章编号:1002-1302(2019)11-0061-05
柞蚕属于鳞翅目(Lepidoptera)天蚕蛾科(Saturniidae)的绢丝类昆虫,我国境内的柞蚕在分类学上属于柞蚕(Antheraea pernyi),主要分布在10余个省份。目前保存着上百个地理种群或生态类型,如山东的客岭庄种、河南的鲁山种、辽宁的瑷阳种、贵州的湄潭种以及用颜色命名的青皮蚕、黄皮蚕、青黄蚕、银白蚕、蓝靛蚕等[1]。这些品种都是经过长期的人工选择培育以及自然突变逐渐形成的具有不同生理、遗传特性和区域适应性的柞蚕品种,其经济性状各具特征[2]。
随着分子遗传学的发展,可以通过DNA大分子本身来分析遗传差异,为物种的起源、亲缘关系和进化研究开辟了一个新的途径。这些研究所提供的大量客观的分子水平上的信息,非常有力地推动了物种起源、亲缘关系和进化的研究。20世纪90年代发明的分子标记技术,特别是随机扩增多态性DNA(random amplified polymorphic DNA,简称RAPD)、选择性扩增DNA片段(selective amplification DNA fragments,簡称SADF)等技术[3-7],以其快速方便的特点,在核酸分子水平上为物种的多态性提供了丰富的信息,从而被广泛应用于物种遗传变异、进化和亲缘关系等研究领域。这些技术的应用已经揭示了许多物种种内或种间的遗传进化与亲缘关系,如亚洲玉米螟地理种群分化的研究[8]、蚱属种间亲缘关系研究[9]、我国野桑蚕和家蚕的系统发育关系研究等[10]。目前已利用这些标记技术成功地构建了家蚕的连锁图[11-14]。在柞蚕上,此方面的研究也已经开展起来[15],刘彦群等利用RAPD标记分析4种体色柞蚕品种的遗传关系,表明品种的遗传聚类划分与体色之间的相关性不大,柞蚕体色并不能真实反映品种间的遗传关系[16];靳向东等作了广适性大型多丝量柞蚕新品种选育报告,将柞蚕品种资源研究运用于新品种选育中,育成2个多丝量柞蚕新品种[17]。在技术上,RAPD技术已被应用在多种动植物上,如农志欢等在十大功劳属6个种上[18]、李言等在海带上[19]、石洪玥等在中华绒螯蟹上[20]、陈德西等在蝗虫上[21]都利用RAPD技术开展了种质资源的研究。目前,国内的研究主要集中在家蚕、玉米、水稻等生物的种质资源方面,有关柞蚕种质资源遗传进化、分类以及亲缘关系的研究在深度和广度上有待完善。因此,本研究采用RAPD技术对71个柞蚕品种的分类和亲缘关系进行了聚类分析,以确立品种间的亲缘关系,以期对我国这一特有生物资源开展进一步的分子生物学研究。
1 材料与方法
1.1 试验材料
收集我国辽宁、河南、吉林、山东与内蒙古等地的不同地理种群和不同化性的柞蚕品种(表1)[22-26]。本研究在大连理工大学生命科学实验室完成。
参照美国Operon公司设计的含有10个寡核苷酸随机引物,由宝生物工程(大连)有限公司合成。RAPD反应系统购自北京鼎国生物工程有限公司。Protease K(蛋白酶K)、RNaseA购自美国Promega公司,琼脂糖为西班牙产,其他常用试剂均为国产分析纯。
1.2 方法
1.2.1 柞蚕基因组DNA的提取 取液氮中冷冻的单个柞蚕蛹组织0.5 g研磨[27],加入2 mL提取缓冲液[10 mmol/L 三(羟甲基)氨基甲烷(Tris)-HCl pH值8.0,0.1 mmol/L 乙二胺四乙酸(EDTA),0.5% 十二烷基硫酸钠(SDS),50 μg/mL 蛋白酶K],在50~60 ℃保温3 h[28-31]。加入等体积的酚混匀,4 ℃、12 000 r/min离心10 min;取上层液加入等体积的酚/三氯甲烷抽提1次。最后用2/3体积的异丙醇沉淀,用75%乙醇洗沉淀1次。溶于400 μL TE中,加RNase于37 ℃消化30 min,-20 ℃保存备用。
1.2.2 RAPD反应体系及扩增程序 参照Williams等的方法[3]对随机扩增具有影响的反应参数, 如镁离子、引物、模板浓度、dNTP和Taq酶用量等进行优化[32-34]。25 μL反应液中,含2 U Taq酶、2.5 μL 10×缓冲液(1.5 mmol/L MgCl2+10 mmol/L Tris-HCl+50 mmol/L KCl+0.01%明胶)、dNTP(各200 umol/L)和Primer(10 pmol/L)各1.0 μL、DNA模板1.0 μL(100 ng)。反应程序如下:94 ℃预变性3 min;94 ℃变性30 s,37 ℃退火40 s,72 ℃延伸2 min,35个循环; 72 ℃延伸10 min。
1.2.3 RAPD多态性分析 每个柞蚕品种取6个个体,分别提取DNA后混合作为模板进行PCR反应。将RAPD扩增产物在1.4%琼脂糖凝胶中恒压电泳2 h,经溴化乙锭(EB)染色后在紫外分析仪下观察拍照,选取重复性好且亮度较强的条带进行统计分析[35]。在电泳图谱上同一RAPD位点上有电泳带者记为1,无电泳带者记为0。根据Nei的公式:I=2NXY/(NX+NY)[36],计算出任意2个个体(群体或物种)间的遗传距离D和遗传相似系数I。式中:NXY为两者共同拥有谱带(RAPD标记)的数目;NX和NY分别为X和Y个体(或群体、物种)各自拥有的全部谱带数。遗传距离的计算和系统聚类采用NTSYS-PC 2.10e[37]。
2 结果与分析
2.1 引物筛选及PCR扩增
采用已经确定的反应体系和条件,以小黄皮、鲁黄、宽青和抗病2号的DNA作为模板,对7组共140条随机引物进行随机扩增,结果共筛选出有效引物60条,再从中筛选出较好的引物28条(表2)。对71个柞蚕品种的DNA进行扩增,共得到92条清晰的具有多态性的谱带,占总带数的29%,片段大小介于500~3 000 bp间,平均每条引物可扩增3条具有多态性的谱带。多态性谱带最多的引物为OPM10(5条),最少的为OPG3(2条)。可见不同引物的扩增产物均有显著差异,其中引物OPH19的扩增结果见图1。此外,不同品种扩增的具有多态性的带数亦不相同,其中多态性谱带最多的品种为印度柞蚕,共有35条,最少的为青6号,只获得16条。
2.2 柞蚕品种间的親缘关系分析
将每个DNA片段看作1个可区分的RAPD标记,根据Nei的公式[36]计算出71份柞蚕品种间的遗传相似系数(I)和遗传距离(D)。结果显示:2个河南一化性品种731与云白的相似系数最高,达到0.886 7,可见两者间的遗传背景比较接近;二化品种黄安东与一化品种豫5号的相似系数为 0.867 1,表明两者之间也有较近的亲缘关系。一化性品种河41同白一化、豫早1号同201、豫早2号同鲁松、鲁黄同松黄等4对品种的相似系数都在0.785 7以上,表明这些品种间的亲缘关系较近。而河41、白一化与其他河南一化性品种的相似系数均低于0.486 2,表明这2个品种与其他一化性品种的亲缘关系较远。
用非加权组平均法(UPGMA)对不同化性和不同地理区域的71个柞蚕品种根据遗传距离(D)进行聚类分析。由图2可以看出,大部分的河南一化品种分布在不同的类群中,并且在各个类群中首先聚到一起。但是,也有不同地区与化性的品种能够较早地聚在一起,如黄安东与豫5号,它们仅次于河南一化性品种731与云白,第2次就聚在一起。图2还说明,各个地区与不同化性的柞蚕品种之间亲缘关系没有明显区别,遗传类型相互混杂。
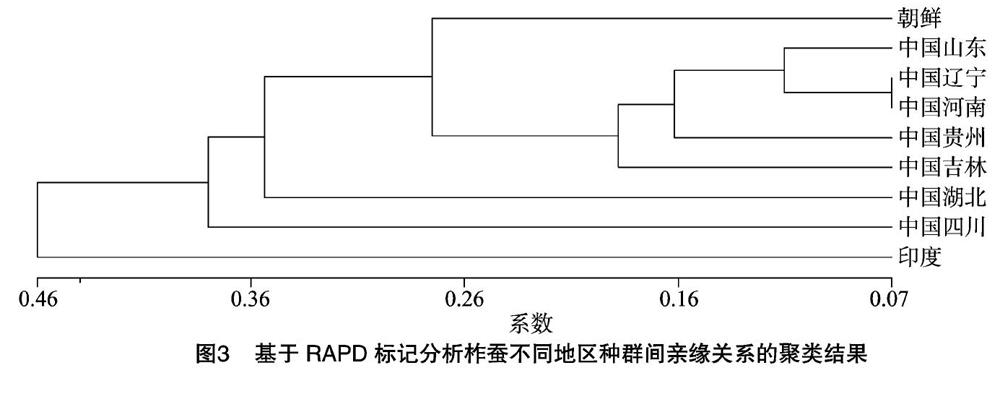
2.3 不同地区柞蚕种群的DNA多态性聚类分析
为了进一步揭示不同地理区域间柞蚕品种的遗传进化关系,根据每个柞蚕品种的最初来源,选择具有代表性的、背景清楚的品种作为该地区的代表性品种(表3)。
将表2中来自同一个地区的所有代表性品种的标记作为该地区种群的RAPD标记,进行不同地区柞蚕种群的遗传距
离(D)分析(表4)。
利用表3数据,用UPGMA法构建聚类树状图。从图3可以看出,来自河南地区的种群与辽宁地区的种群首先聚类到一起, 两者之间的遗传距离只有0.066 3。山东与辽宁、山东与河南种群的遗传距离分别为0.104 2、0.126 1。除了四川与湖北、四川与印度的遗传距离分别为0.693 1、0.665 0(>0.5)外,其他地区之间的遗传距离都<0.5。
3 结论与讨论
柞蚕为鳞翅目(Lepidoptera)天蚕蛾科(Saturniidae)的绢丝类昆虫,已记载的共有35个种,其中31个种分布在印沃区,3个在古北区,1个在北美区。为人们所熟知并用于商业性生产的有中国柞蚕(Antheraea pernyi)、2种印度柞蚕(Antheraea assamensis和Antheraea mylitta)和日本天蚕(Antheraea yamamai)。
我国现存130多个品种都是中国柞蚕的变种和地理种或生态型。各个品种之间的血缘关系非常复杂,各个品种之间既有高度变异的一面,又有亲缘很近的一面。刘彦群等通过对河41、四青、青黄1号和杏黄4个品种的研究发现,在DNA水平上的分类并没有按体色进行[38],对现行的体色分类系统提出了质疑。
本研究表明,不同的品种,即使分布相距很远,有的竟表现出惊人的相似性和紧密的亲缘关系;而同一个品种的不同生态型,有的却相差悬殊。如在对71个柞蚕品种进行RAPD多态性聚类分析时发现,整个系统聚类图分为2支,每个分支中各地区的品种相互掺杂。但是在第一集团的40个品种中,一化性品种有15个,占37.5%;在第二集团31个品种中,一化性品种只有8个,占25.8%。从整个系统聚类图上可以看出,大部分的一化品种分布在不同的类群中,并且在各个类群中首先分别聚到一起。河南的一化性品种731与云白的相似系数最高,达到了0.886 7,可见两者间的遗传背景非常接近;一化性品种河41同白一化、豫早1号同201、豫早2号同鲁
松、鲁黄同松黄4对品种的相似系数也都在0.785 7以上,表明这些品种间的亲缘关系较近。但是,也有不同地区与化性的品种能够较早地聚在一起,如黄安东与豫5号,它们仅次于2个河南一化性品种731与云白,相似系数为0.867 1。
有关柞蚕的分布与分化,印度学者乔利认为,生长在热带的印度柞蚕(A.assamensis)是柞蚕的原始类型,其他品种都起源于它,我国境内的柞蚕是由印度东北向我国南部传播的[39]。本研究结果表明,辽宁地区的品种首先与河南地区的品种聚在一起,然后又与山东地区的品种聚在一起。以0.728 1为分界,除了贵州以外,包括朝鲜在内的长江以北的地区品种首先聚类在一起,这一集团中包括我国2个古代柞蚕传播中心——山东和河南与现代柞蚕的传播中心辽宁。印度柞蚕最早从整个系统中分离出来,然后是四川、湖北。虽然在古文献中没有找到有关这些地区柞蚕品种起源的记载,但是从系统聚类图中可以推测,在远古的时候柞蚕由印度传到我国后,由四川经湖北向长江以北地区传播,到达我国古代柞蚕传播中心山东、河南与现代柞蚕的传播中心辽宁。然后又从这些地区向吉林、贵州以及朝鲜等地传播,所以发现吉林、贵州以及朝鲜的品种又从这些品种中分化出来。在最后剩下的辽宁、河南、山东的品种中,辽宁与河南分化得最晚;长江以北的地区品种的分化情况基本上与历史资料的记载相吻合[1]。
现代遗传学的观点认为物种的遗传性状是由基因决定的,各种外部形态及内部性状的变异都是由于基因中DNA分子碱基序列发生改变。本研究在分子水平上对我国柞蚕品种资源的遗传分化和亲缘关系进行了探讨,可能与已有的研究不完全相符,期望有助于对我国这一特有生物资源的保护和深入研究。
参考文献:
[1]辽宁省蚕业科学研究所. 中国柞蚕品种志[M]. 沈阳:辽宁科学技术出版社,1994.
[2]郭奕生,陆 俊. 微卫星标记在杂交稻种子纯度鉴定上的应用[J]. 广东农业科学,2014,41(16):138-141.
[3]Williams J G,Kubelik A R,Livak K J,et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Research,1990,18(22):6531-6535.
[4]Welsh J,McClelland M. Fingerprinting genomes using arbitraryprimers[J]. Nucleic Acids Research,1990,18(24):7213-7218.
[5]van der Voort J R,Wolters P,Folkertsma R,et al.Mapping of the cyst nematode resistance locus Gpa2 in potato using a strategy based on co migrating AFLP markers[J]. Theoretical and Applied Genetics,1997,95(5/6):874-880.
[6]Keim P,Schupp J M,Travis S E,et al. A high-density soybean genetic map based on AFLP markers[J]. Crop Science,1997,37(2):537-543.
[7]陳 洪,王振山,朱立煌. SRFA法构建水稻DNA指纹图谱[J]. 生物工程学报,1996,12(3):266-269.
[8]孙 姗,徐茂磊,王戎疆,等. RAPD方法用于亚洲玉米螟地理种群分化的研究[J]. 昆虫学报,2000,43(1):103-106.
[9]蒋国芳,陆 敢,黄 琨,等. 用RAPD标记研究蚱属五个种间的亲缘关系[J]. 昆虫学报,2002,45(4):499-502.
[10]鲁 成,余红仕,向仲怀. 基于RAPD分析的中国野桑蚕和家蚕遗传多样性和系统发育关系研究[J]. 昆虫学报,2002,45(2):198-203.
[11]夏庆友,周泽扬,鲁 成,等. 家蚕不同地理品种分子系统学研究[J]. 昆虫学报,1998,41(1):32.
[12]李 斌,鲁 成,周泽扬,等. RAPD标记构建家蚕分子连锁图[J]. 遗传学报,2000,27(2):127-132.
[13]何宁佳,鲁 成,李 斌,等. 结合SADF与RAPD标记构建家蚕连锁图[J]. 昆虫学报,2001,44(4):476-482.
[14]朱玉芳,谭远德,万春玲,等. 家蚕AFLP连锁框架图谱的构建[J]. 昆虫学报,2001,44(4):483-493.
[15]桂慕燕,左正宏,王学民,等. RAPD分析在绢丝昆虫亲缘关系研究中的应用Ⅱ.柞蚕品种间的遗传差异[J]. 遗传,2001,23(5):452-454.
[16]刘彦群,鲁 成,向仲怀. 4种体色柞蚕品种遗传关系的RAPD分析[J]. 蚕业科学,2006,32(1):20-24.
[17]靳向东,朱兴友,任晓义,等. 广适性大型多丝量柞蚕新品种选育报告[J]. 北方蚕业,2016,37(1):1-6.
[18]农志欢,张启伟,杨 平,等. 十大功劳属6个种的遗传多样性及亲缘关系RAPD分析[J]. 南方农业学报,2016,47(7):1077-1082.
[19]李 言,刘延岭,崔翠菊,等. 海带种质资源遗传多样性的RAPD分析[J]. 生物技术通报,2016,32(4):151-158.
[20]石洪玥,刘 阳,王晓梅,等. 七里海中华绒螯蟹遗传多样性的RAPD和ISSR分析[J]. 水产科学,2016,35(3):252-260.
[21]陈德西,封传红,何忠全,等. 四川省甘孜州9个不同地理蝗虫种群遗传多样性的RAPD分析[J]. 西南农业学报,2016,29(6):1336-1342.
[22]宋娜娜,柴志欣,何世明,等. 三江黄牛RAPD遗传多样性研究[J]. 畜牧与兽医,2016,48(6):30-35.
[23]Imazio S,Labra M,Grassi F,et al. Chloroplast microsatellites to investigate the origin of grapevine[J]. Genetic Resources and Crop Evolution,2006,53(5):1003-1011.
[24]王梦亮,任晓琳,崔晋龙,等. 野生红景天的RAPD和ISSR遗传多样性分析[J]. 中草药,2016,47(3):469-473.
[25]宋宪军,聂 磊,张 涛,等. 柞蚕部分品种及杂交种的RAPD分析[J]. 蚕业科学,2004,30(4):428-431.
[26]赵菲佚,焦成瑾,李志明,等. RAPD法鉴定紫花苜蓿品种的条件优化[J]. 湖南农业科学,2016(1):1-5.
[27]曹兰娟,杨宝山,李 俊,等. 不同地区栗蚕(Dictyoploca japonica)的RAPD的分析[J]. 蚕业科学,2007,33(2):293-296.
[28]Wang X L,Wang X L,Chang C R. RAPD analysis of Rosa laevigata Michx. from different origins[J]. Agricultural Biotechnology,2015,4(5):33-36.
[29]王 卓,曹兰娟,刘振林,等. 不同地区及不同季节的柞蚕寄生蝇RAPD分析[J]. 蚕业科学,2008,34(2):354-358.
[30]王小武,付 开,丁新华,等. 基于RAPD标记的新疆荒漠稻区稻水象甲遗传多样性分析[J]. 新疆农业科学,2016,53(11):2069-2076.
[31]王凤成,刘丹梅,仝振祥,等. 用RAPD标记分析部分柞蚕二化性品种资源的遗传多样性[J]. 蚕业科学,2009,35(1):148-153.
[32]郭晓丽,白丽荣. 10个小黑麦品种(系)的遗传多样性分析[J]. 河北农业科学,2016,45(2):26-28.
[33]程道军,鲁 成,周泽扬,等. 几种绢丝昆虫遗传多样性的RAPD研究[J]. 蠶业科学,2002,28(4):277-282.
[34]Gui M Y,Zuo Z H,Wang X M,et al. Application of RAPD technique in genetic relationship of silk insect. Ⅱ.Genetic variance in Antheraea pernyi[J]. Heredity,2001,23(5):452-454.
[35]钟伯雄,张金卫,丁 农,等. RAPD技术在家蚕杂交率检验中的应用[J]. 浙江大学学报(农业与生命科学版),2003,29(3):321-324.
[36]Nei M.Estimation of average heterozygosity and genetic distance from a small number of individuals[J]. Genetics,1978,89(3):583-590.
[37]陈莹玉,袁思思,吴春兰,等. 广西金秀野生茶遗传多样性及分子指纹图谱研究[J]. 广东农业科学,2016,43(3):21-28.
[38]刘彦群,鲁 成,向仲怀. 中国柞蚕DNA多态性的RAPD分析[J]. 蚕业科学,2002,28(4):283-288.
[39]乔利MS,林华生. 柞蚕属种的分布与分化[J]. 国外农学-蚕业,1983(4):3-9.
