饲用型小黑麦遗传图谱构建及草产量相关性状QTLs初步定位
2019-04-16赵方媛李冬梅田新会杜文华
刘 晶, 赵方媛, 李冬梅, 李 雪, 田新会, 杜文华
(甘肃农业大学草业学院/草业生态系统教育部重点实验室/甘肃省草业工程实验室/中-美草地畜牧业可持续 发展研究中心, 甘肃 兰州 730070)
小黑麦(×TriticosecaleWittmack)是由小麦属(Triticum)和黑麦属(Secale)经属间有性杂交和染色体数加倍培育而成的一种新异源多倍体物种[1]。其不但结合了小麦高产优质和黑麦抗病、抗寒、抗旱和适应性强的优点,而且还具有杂种优势强和营养品质好等特点[1]。饲用型小黑麦单株生物量高,饲草的粗蛋白含量高,植株鲜嫩多汁、消化率高、适口性好,为家畜所喜食[2]。由于其抗寒性强,在青藏高原高寒牧区具有一定应用前景[3]。
饲用型小黑麦的草产量与株高、单株分蘖数以及单株生物量密切相关。通过聚合上述优异性状,就可以培育高产小黑麦新品种(系)。目前我国饲用型小黑麦新品种的培育大多采用常规育种技术,传统育种周期长,自然突变率低,有益突变少,不利于大规模和多方向育种[4],同时也不能满足畜牧业快速发展对优良草品种的需求。如果在常规育种基础上,引进和运用现代育种技术,就可以使品种选育向快速、高效、定向的方向发展[5-6]。
借助DNA分子标记技术构建小黑麦遗传连锁图谱,并对控制草产量相关数量性状(Quantitative Trait Locus,QTLs)的基因进行跟踪和克隆,充分利用,就能提高育种效率和准确性。目前,研究者已将ISSR分子标记(Inter-simple sequence repeat)运用到多个植物的遗传图谱构建中。潘玉玲[7]利用利用3种分子标记技术(SSR、SRAP和ISSR)对丹参(SalviamiltiorrhizaBge)F1代单株标记进行了遗传连锁分析,并构建了一张遗传连锁图谱。汪斌等[8]利用筛选出的20条ISSR多态性引物对84份国内外红麻(Hibiscuscannabinus)种质资源进行PCR扩增,选择5条扩增结果较好的引物构建了国内外红麻种质资源的ISSR指纹图谱构建。殷丽琴等[9]利用ISSR分子标记技术绘制了10份彩色马铃薯(SolanumtuberosumL.)品种的指纹图谱。赵雅姣等[10]筛选出12条ISSR多态性引物对30份饲草型小黑麦种质进行了遗传多样性分析,以了解不同种质间的遗传差异。赵方媛等[11]利用ISSR分子标记构建了1张小黑麦遗传连锁图谱,并对抗条锈QTL进行了初步定位分析。
国内外对牧草相关性状QTL的定位研究主要集中在黑麦草属(Lolium)[12]、赖草属(Leymus)[13]、苜蓿属(Medicago)[14]、三叶草属(Trifolium)[15]、鸭茅(Dactylis)[16]、高丹草(Hupozhongzi)[17]、结缕草(Zoysia)[18]和二倍体冰草(Agropyron)[19]等牧草上。Studer等[20]以306个多花黑麦草(LoliummultiflorumLam.)F1代单株为作图群体,利用AFLP和SSR分子标记构建了多花黑麦草分子遗传连锁图谱,在第1、2连锁群上发现了控制冠锈病相关的QTLs,其遗传贡献率高达56%。Barrett等[21]利用2个高度杂合的白三叶(TrifoliumrepensL.)种质杂交得到的182个F1代为作图群体,构建了白三叶遗传连锁图谱,并对种子产量、花序密度、千粒重和花序收益率进行了定位研究,得到23个与种子产量相关的QTLs,为提高白三叶种子产量和分子标记辅助选择育种奠定了基础。Espinoza和Julier[22]利用蒺藜苜蓿(Medicagotruncatula)的作图群体检测了与牧草品质(粗蛋白、消化率、茎叶比等)相关的QTL定位。李小雷等[23]构建了二倍体冰草的分子遗传连锁图谱,并对冰草10个重要农艺性状进行检测,在6个连锁群上共检测到13个与农艺性状相关的QTLs。国内外小黑麦QTL定位方面的研究较少,主要集中在八倍体粮用型小黑麦上。Niedziela等[24]对粮用型小黑麦耐土壤铝离子的相关基因进行了定位,Würschum等[25]以4个双单倍体群体为作图群体,通过研究粮用型小黑麦孕穗期、盛花期和乳熟期控制株高QTL和抗倒伏基因的关系,旨在降低小黑麦株高,提高种子产量。Alheit等[26]利用多系杂交的4个双单倍体群体对控制饲用型小黑麦乳熟期生物量和株高的QTL进行了定位。截至目前,国内外对六倍体饲用型小黑麦遗传图谱构建研究较少[11],并且尚未有饲用型小黑麦草产量相关性状QTL定位方面的研究报道。为此,本研究拟利用饲用型小黑麦F2代群体构建遗传连锁图谱,并结合田间表型数据,对饲用型小黑麦草产量相关性状(株高,分蘖数,单株生物量)进行QTL定位分析,获取与之紧密连锁的分子标记,为利用分子标记辅助育种培育饲用型小黑麦新品种,以及农艺性状的遗传改良提供理论依据。
1 材料与方法
1.1 试验地概况
试验地位于甘肃省临洮县农校农场,位于甘肃省中部,定西市西部(103°87′ E,35°37′ N)。海拔1 892 m,属温带大陆性气候,多年气象数据资料显示,临洮县年降水量562 mm,无霜期153 d,年平均气温7.0℃。有灌溉条件,土壤为黑麻土。
1.2 试验材料
参试材料为甘农2号小黑麦(父本)和甘农1号小黑麦(母本)杂交形成的521个F2代单株及亲本材料。父本和母本均是甘肃农业大学选育而形成的基因纯合、性状稳定性的六倍体饲用型小黑麦品种,两个亲本生态类型差异较大:母本株高较低,分蘖数较多,单株生物量较高;父本株高较高,分蘖数低,单株生物量低。
1.3 遗传作图群体构建
将收获的F1代单株的主穗种子按编号顺序种植于试验地,并种植父母本材料各1行。点播,行长1 m,株距0.1 m,行距0.2 m,试验期间按照常规田间管理技术进行管理。出苗后20 d给形成的521个单株做好标记,分别采集每个单株的幼嫩叶片,用锡箔纸包装后置于液氮中速冻,储存于-80℃,用于遗传图谱构建。开花期田间测定每个单株的株高,成熟期按单株连根拔出,田间晾干后带回室内测定每个单株的分蘖数(包括有效分蘖数和无效分蘖数),之后剪掉根系,测定单株生物量。参照文献[25-26]的方法,根据521个单株的株高、分蘖数和单株生物量数据,每个指标中分别选取较大值和较小值单株各30株用于草产量相关性状QTLs定位。
1.4 遗传作图群体基因组DNA提取及ISSR-PCR扩增
用改进的CTAB法[27]提取521个单株的全基因组DNA,利用1.0%琼脂糖凝胶电泳和紫外分光光度计检测DNA浓度和纯度,合格的样品于-20℃冰箱保存备用。饲用型小黑麦最优(20 μl) ISSR-PCR反应体系采用项目组前期研究结果[28]。所用ISSR引物为项目组前期从适于禾本科植物ISSR-PCR反应体系的64条引物中筛选出的、适宜于小黑麦多样性分析的谱带清晰稳定、重复性好、多态性高的14条引物[28],一共扩增出清晰可辨的条带共110条,其中多态性条带68条。
1.5 基因型数据统计方法
将ISSR标记所获得的作图群体的多态性条带进行统计。条带统计方法:各个单株如果在该位点的带型与母本相同记为“A”,与父本相同记为“B”,同时具有父母本双亲带型的记为“H”,缺失的带型记为“-”。
1.6 遗传连锁图谱构建和QTL定位分析
本研究采用JoinMap3.0软件[29]绘制完成饲用型小黑麦遗传连锁图谱。参考文献[26],设置LOD≥2,步长为1.0,在2.0~10.0的LOD值范围内对标记进行分组,其余参数值为软件默认参数。利用区间作图法(interval mapping,IM)[30]进行QTL定位分析。
QTL的命名规则方法为“Q+X+定位连锁群及个数”,其中“Q”为QTL的英文缩写,“X”为性状名称的英文缩写(如:株高,plant height,PH),定位连锁群用数字表示,若同一个性状在同一连锁群中检测到多个位点,则在该连锁群后面依次标1、2、3等,QTL的名称一般用斜体[30]。
1.7 田间表型数据分析
使用Microsoft Excel 2007软件对F2代群体的株高、分蘖数和单株生物量的田间表型数据进行统计分析,绘制频率分布柱形图,并对亲本的测定数据进行差异显著性分析。
2 结果与分析
2.1 饲用型小黑麦杂交F2代群体的田间表型鉴定
2.1.1饲用型小黑麦F2代群体草产量相关性状分析 饲用型小黑麦F2代521个单株株高、分蘖数和单株生物量的田间表型结果(表1,图1)表明,F2代群体具有超亲现象且呈现出数量性状的遗传特点。就F2群体各性状的平均值而言,株高、分蘖数和单株生物量均高于中亲值,体现出超亲优势,即后代趋向于分蘖数增多,单株生物量增加。3个性状在F2代中均表现出广泛变异,其中分蘖数和单株生物量的变异系数和变异幅度较大,株高的变异系数和变异幅度较小。变异的连续性是各性状的共同特征,在该群体中株高呈正态分布(峰度=3,偏度<1),分蘖数和单株生物量呈正偏态分布(峰度>3,偏度>1)。说明与草产量相关的3个重要表型性状基本上呈连续正态分布,能够作为遗传图谱作图群体[26]。
2.1.2饲用型小黑麦F2代群体草产量相关性状的相关性分析 饲用型小黑麦F2代群体草产量相关性状的相关性分析(表2)表明,株高和分蘖数、单株生物量极显著正相关,分蘖数和单株生物量极显著正相关(P<0.01)。
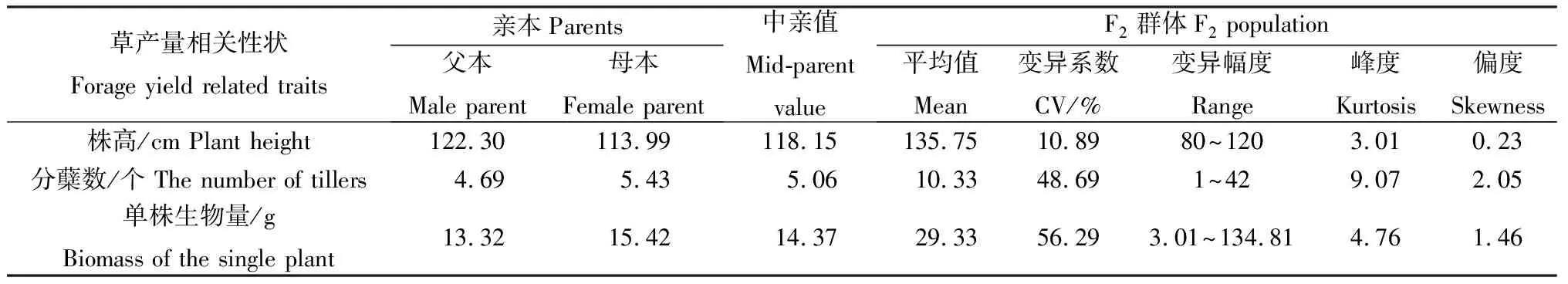
表1 饲用型小黑麦杂交F2群体和父母本草产量相关性状的比较Table 1 Comparison of the forage yield related traits between the F2 population and their parents in triticale
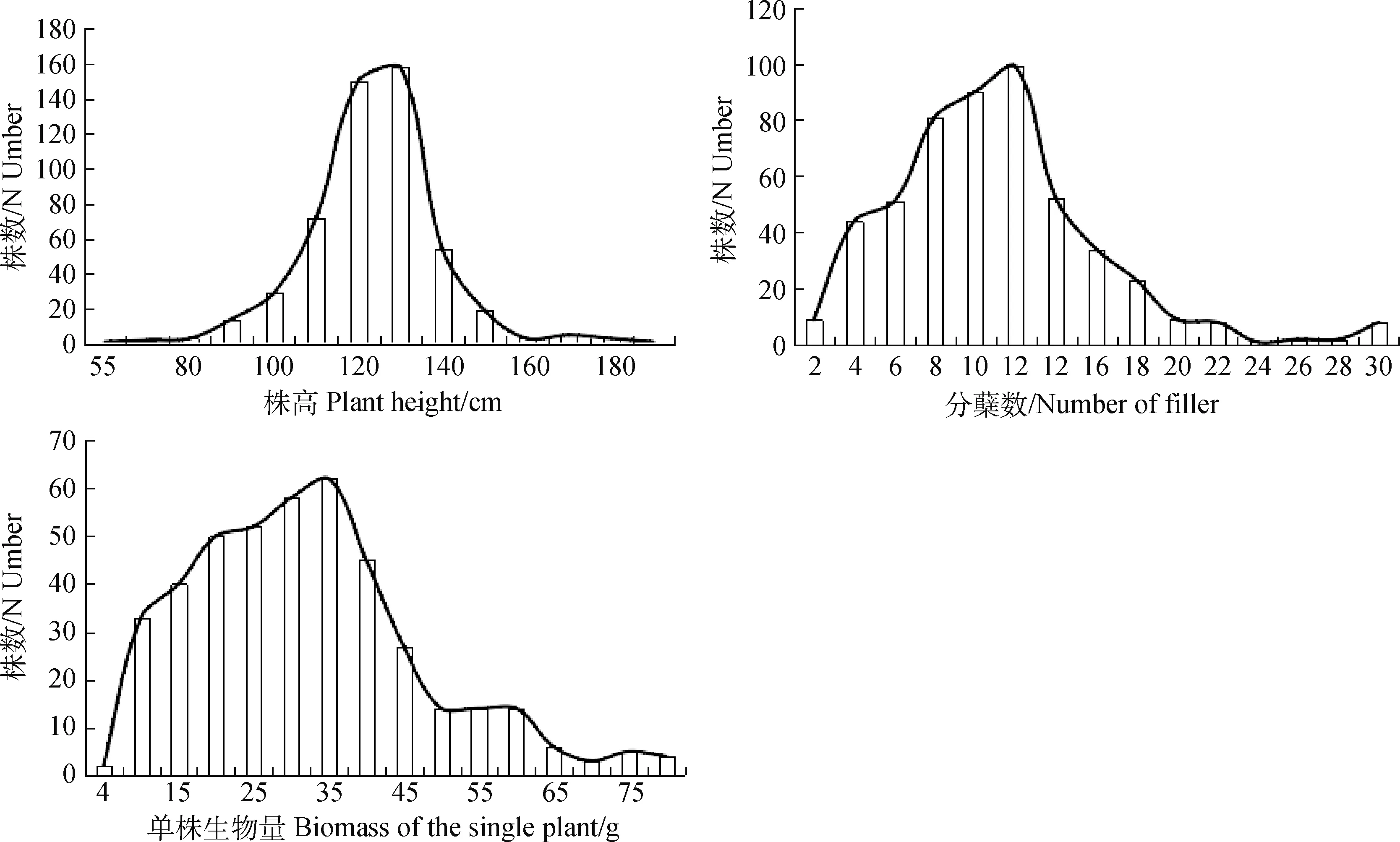
图1 小黑麦杂交F2群体株高、分蘖数和单株生物量的次数分布Fig.1 Distribution frequency of plant height,the number of tillers and biomass of the single plant in the F2 populations of triticale
表2 饲用型小黑麦F2代群体草产量性相关性状的相关系数Table 2 Correlation coefficients among forage yield related traits in F2 populations of triticale

性状Trait株高Plant height分蘖数The number of tillers单株生物量Biomass of the single plant株高Plant height1.0000.354∗∗0.545∗∗分蘖数The number of tillers1.0000.772∗∗单株生物量Biomass of the single plant1.000
注:**表示极显著相关(P<0.01)
Note:**indicate significant correlated at the 0.01 level
2.2 饲用型小黑麦F2群体遗传连锁图谱构建与分析
以F2群体521个单株的田间表型数据为基础,利用Joinmap 3.0作图软件,进行遗传连锁分析作图,构建了1张遗传连锁图谱[11](图2)。本连锁图谱包含7个连锁群(分别命名为LG1~LG7),ISSR分子标记共92个,标记间平均距离为5.90 cM,图谱总长度为542.9 cM。各连锁群上的分子标记数存在差异,变化范围为9~18个;各连锁群对应的空隙数目也各不相同,变化范围为8~17个;各连锁群上标记间隔在9.6~15.0 cM之间;连锁群上标记间平均距离变异范围在4.93~7.80 cM之间;各连锁群的长度在54.7~124.8cM之间(表3)。

图2 小黑麦F2群体ISSR遗传连锁图谱构建 及草产量相关性状QTL定位Fig.2 Genetic linkage map and QTL for forage yield related traits in F2population of triticale based on ISSR marker注:QPH表示控制株高的QTLs;QNT表示控制分蘖数的QTLs;QBS表示控单株生物量的QTLsNote:QPH means QTLs controlling the plant height,QNT means QTLs controlling the number of tillers,and QBS means QTLs to controlling the biomass of single plant
2.3 饲用型小黑麦杂交F2代群体草产量相关性状的QTL定位
运用MapQTL6.0软件,以LOD≥2.0为阈值,对饲用型小黑麦F2群体草产量相关性状(株高,分蘖数,单株生物量)进行QTL定位分析。共检测到17个相关QTLs,分布在6个连锁群(LG1,LG2,LG3,LG4,LG5,LG6)上,平均每个连锁群2.8个,QTLs在6个连锁群上分布不均匀,LG2上分布最多,有5个QTLs;连锁群LG3上分布最少,只有1个QTL(图2)。
控制株高的5个QTLs分布在LG2、LG3和LG7连锁群上,分别为QPH2-1、QPH2-2、QPH3、QPH7-1和QPH7-2,所在区间分别为UBC873-1~UBC873-5、UBC826-1~UBC847-5、UBC849-3~UBC834-2、UBC815-3~UBC826-5和UBC826-5~UBC857-1,临近标记分别为UBC873-3、UBC847-5、UBC857-4、UBC808-9和UBC857-5。
7个控制分蘖数的QTLs分布在LG1、LG2、LG4和LG5连锁群上,分别为QNT1-1、QNT1-2、QNT2-1、QNT2-2、QNT4-1、QNT4-2和QNT5,所在区间分别为UBC849-4~UBC834-5、UBC826-4~UBC822-3、UBC873-3~UBC860-1、UBC860-1~UBC826-1、UBC860-3~UBC857-2、UBC815-1~UBC815-7和UBC835-4~UBC815-4,临近标记分别为UBC825-1、UBC822-3、UBC873-5、UBC873-6、UBC808-8、UBC815-5和UBC849-5,对应的遗传距离(cM)分别为21.13,74.5,39.90,53.33,21.24,92.50和43.76。
5个控制单株生物量的QTLs分布在LG1、LG2、LG4和LG5连锁群上,分别为QBS1、QBS2、QBS4-1、QBS4-2和QBS5,所在区间分别为UBC849-4~UBC825-1、UBC822-1~UBC826-1、UBC860-3~UBC857-2、UBC815-1~UBC815-7和UBC835-4~UBC815-4,临近标记分别为UBC825-1、UBC822-3、UBC873-5、UBC873-6、UBC808-8、UBC815-5和UBC849-5,对应的遗传距离分别为16.23,53.34,21.24,92.50和43.76。
2.3.1控制株高 QTL分析5个控制株高的QTLs(QPH2-1,QPH2-2,QPH3,QPH7-1,QPH7-2),对应的LOD值分别为4.37,2.78,4.82,4.52和5.65,单个QTL的贡献率分别为10.4%,6.7%,11.4%,10.7%和13.2%(表4),其中贡献率为13.2%的QPH7-2为主效QTL,加性效应的变化范围为-23.22~17.91。
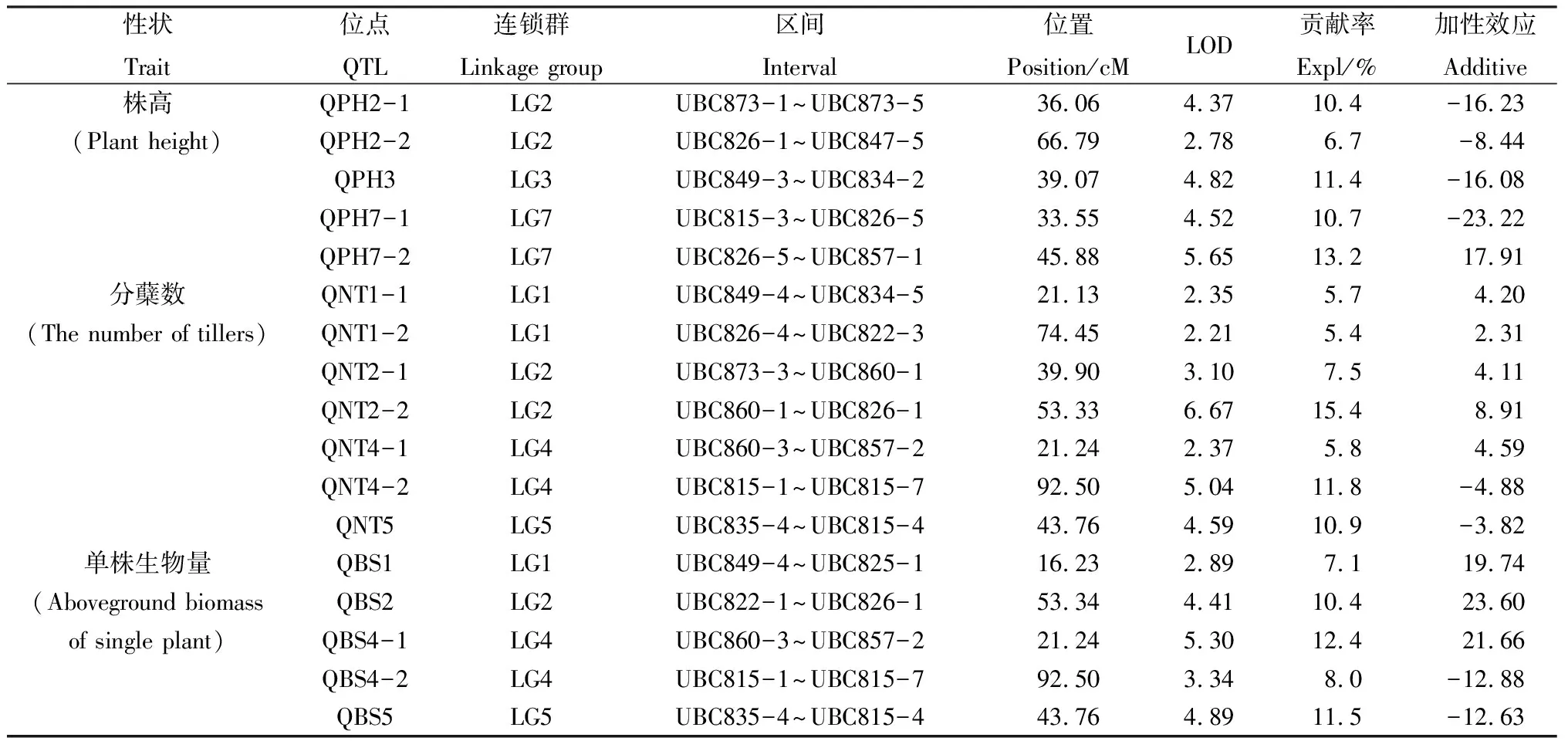
表4 饲用型小黑麦F2代群体株高、分蘖数和单株生物量的QTLs定位结果Table 4 QTLs mapping on plant height,the number of tillers and biomass of the single plant in F2 population of triticale
2.3.2控制分蘖数 QTLs分析控制饲用型小黑麦分蘖数的7个QTLs标记QNT1-1,QNT1-2,QNT2-1,QNT2-2,QNT4-1,QNT4-2和QNT5,对应的LOD值分别为2.35,2.21,3.10,6.67,2.37,5.04和4.59,单个QTL的贡献率分别为5.7%,5.4%,7.5%,15.4%,5.8%,11.8%和10.9%(表4),其中贡献率为15.4%的QNT2-2为主效QTL,加性效应的变化范围在-4.88~8.91之间。
2.3.3控制单株生物量 QTLs分析以LOD≥2.0为阈值,5个控制单株生物量的QTLs(QBS1,QBS2,QBS4-1,QBS4-2,QBS5)对应的LOD值分别为2.89,4.41,5.30,3.34和4.89,单个QTL的贡献率为7.1%,10.4%,12.4%,8.0%和11.5%(表4),其中贡献率为12.4%的QBS2为主效QTL,加性效应的变化范围在-12.88~23.60之间。
3 讨论
小黑麦作为新型饲料作物,其抗寒性强、草产量和粗蛋白含量高,在青藏高原高寒牧区和华北、西北、西南地区具有极其广阔的应用前景。要进一步提高其草产量,培育高产品种是关键。利用QTL技术可以将草产量相关性状进行定位,可为饲用型小黑麦草产量相关主效QTL的挖掘、功能基因定位以及分子标记辅助育种奠定基础,有利于提高育种效率,培育高产饲用型小黑麦品种。但目前国外QTL研究主要集中在粮用型小黑麦[24-25]方面,饲用型小黑麦QTL研究较少[26]。郭建文等[31]研究表明,饲用型小黑麦杂交F1代具有丰富的遗传变异,46个真杂种单株的株高、分蘖数和单株生物量等表现出明显的杂种优势。本研究以饲用型小黑麦F2代为试验材料,研究发现株高、分蘖数和单株生物量均具有超亲现象,有利于选育高产饲用型小黑麦品种(系)。饲用型小黑麦分蘖数和单株生物量测定应在开花期进行[3],本研究考虑到育种需要,要收获种子,所以推迟到完熟期进行。测定分蘖数时也测定了无效分蘖,虽然该指标对种子产量无贡献,但其对饲用型小黑麦草产量却具有重要贡献[3]。QTL定位时,构建作图群体所需株系一般为100~400个[32-33]。本研究F2代521个单株的株高、分蘖数和单株生物量呈连续变异,分布频率大致接近正态分布,体现出数量基因控制分离群体的特点[34]。从而说明可用于QTL定位。
理想的遗传连锁图谱一般含有较多分子标记,并且标记分布均匀、距离较近,连锁群的数目和染色体数目一致等[26]。Würschum等[25]认为,构建一个基本的连锁图谱框架需要分子标记间的平均距离为10~20 cM,用于QTL定位的遗传连锁图谱则要求平均间隔小于10 cM。本研究利用前期研究[28]筛选出的14对ISSR多态性引物对小黑麦F2代群体进行遗传连锁分析,初步构建了1张连锁图,标记间平均距离为5.90 cM,说明该遗传连锁图谱适合于进行QTL定位。但该图谱只包含92个ISSR标记,标记位点较少,有待于利用AFLP、SSR、SNP等其他分子标记,以挖掘更多标记,来增加遗传图谱密度。本研究采用F2代群体并非最理想群体,因此还需在此基础上,进一步采用单粒传法获得高代稳定系(RIL群体),然后通过测定草产量相关性状,以提高QTL结果定位的准确性。
株高、分蘖数和单株生物量是决定饲用型小黑麦草产量高低的重要因素[2]。Würschum等[25]利用DArT标记在粮用小黑麦孕穗期、盛花期和乳熟期分别检测到15、18和8个控制株高的QTLs。本研究利用ISSR标记构建饲用型小黑麦遗传图谱,并对开花期饲用型小黑麦株高、成熟期分蘖数和单株生物量进行QTL定位,共检测到17个QTLs,其中控制株高的QTLs有5个,控制分蘖数的QTLs有7个,控制单株生物量的QTLs有5个。虽然本研究检测到控制开花期株高的QTLs数量较少,且只有1个(QPH7-1)和Würschum等[25]检测到的乳熟期QTL相同,但本研究检测到的、控制饲用型小黑麦开花期株高的QTLs可为前述研究[25]的补充。本研究表明,控制饲用型小黑麦株高、分蘖数和单株生物量的QTLs在连锁群上分布不均匀。这主要是因为控制不同性状的QTLs所处位置不同[26],也与本研究涉及的数量性状较少有关[35-36]。
本研究发现,饲用型小黑麦QTL分布有聚集趋势,表现出一因多效现象,遗传图谱中LG1连锁群上21.23 cM处的标记同时影响分蘖数和单株生物量;LG2连锁群上39.90 cM处的标记同时影响株高和分蘖数,53.34 cM处的标记同时影响分蘖数和单株生物量;LG4连锁群上21.24 cM和92.50 cM处的标记同时影响分蘖数和单株生物量;LG5连锁群上43.76 cM处的标记同时影响分蘖数和单株生物量。这一现象表明遗传连锁图谱上同一个位点的QTL可同时影响株高、分蘖数和单株生物量等不同性状,而且这些性状的相关性较高。这种现象也出现在黄瓜(CucumissativusLinn.)[37]、小麦[38]、水稻(OryzasativaL.)[39]和玉米(Zeamays)[33]等作物中。经典数量遗传学认为,基因的多效性或者连锁性是2个性状具有相关性的主要原因[37]。许多QTL研究[40-42]也试图从分子水平上证明这个问题。因此,需要通过增加标记数量来提高饲用型小黑麦图谱密度以及饱和度,为进一步挖掘主效基因,研究多因一效和一因多效的遗传机制,以及精细定位优良基因奠定基础。
QTL容易受遗传背景和环境条件影响。这可能与数量性状间基因与基因的互作和改变有关,这种现象称为上位性[20],基因与环境的互作效应对QTL也有一定影响。在数量性状遗传中上位效应的作用不可忽视[43-44]。要确保QTL的正确性和真实性,必须严格控制环境对植物的影响[45]。本研究是在一个试验地环境下开展的研究,缺少不同试验地环境因素影响,因此,要构建饲用型小黑麦遗传图谱并进行QTL精细定位,就需要在不同环境条件下对多个基因型的QTL进行分析,以获得更加准确的QTL位点。
4 结论
饲用型小黑麦F2代的521个单株的株高、分蘖数和单株生物量呈连续变异,分布频率大致接近正态分布,可用于QTL定位。饲用型小黑麦遗传图谱包含7个连锁群(LG1~LG7),图谱总长度为542.9 cM,标记间平均距离为5.90 cM,总共有92个位点。LOD≥2.0时,共检测到17个草产量相关QTLs,其中控制株高QTLs 5个,分蘖数QTLs 7个,单株生物量QTL是5个,这些QTLs分布在6个连锁群上,平均每个连锁群2.8个;QTLs在6个连锁群上的分布不均匀,LG2上分布最多(5个),LG3上分布最少(1个)。本研究通过对与饲用型小黑麦草产量紧密相关的主要农艺性状(株高、分蘖数、单株生物量)进行QTLs定位分析,可为相关主效基因的克隆、转化、利用以及农艺性状的遗传改良提供理论依据。
