桑树光合作用转录组研究
2019-01-11李勇于翠莫荣利邓文熊超庄楚雄胡兴明
李勇 于翠 莫荣利 邓文 熊超 庄楚雄 胡兴明

摘要:研究高产桑品种鄂桑1号(E1)和对照桑品种湖桑32号(H32)光合特性及光合相关基因表达变化,为桑树高光效育种以及种质资源筛选提供理论依据。利用LI-6400XT型便携式光合测定仪测定桑树叶片气体交换和叶绿素荧光参数,同时测定叶绿素含量和1,5二磷酸核酮糖羧化酶(RUBP)活性,在桑树光合日变化曲线峰值和谷底取样进行转录组测序,比较不同桑品种光合相关基因表达的差异。对E1和H32的光合日变化测定结果显示,桑品种E1叶片的Pn峰值显著大于H32峰值(P<0.05),H32叶片Tr峰值显著大于E1峰值(P<0.05);不同品种桑树叶片Pn-PAR和Pn-Ci的响应中,CE、CSP为E大于H32,AQY、LCP、LSP、CCP为H32大于E1。E1在较弱光强下实现光合产物积累的能力强于H32,E1对CO2低浓度的利用效率优于H32;不同桑品种叶片叶绿素荧光参数中,E1叶片ΦPSⅡ和qP显著大于H32(P<0.05),H32的NPQ显著大于E1(P<0.05),表明H32叶片吸收的光能较多的以热能的形式进行耗散;H32的叶绿素a、叶绿素b和叶绿素含量均显著小于E1(P<0.05),E1的RUBP活性大于H32(P>0.05),表明E1光合能力优于H32。不同品种不同时段发现3 356个差异表达基因(Differentially expressed genes,DEGs),代谢通路分析显示共有1 136个DEGs注释到KEEG数据库,DEGs显著富集的光合作用相关代谢通路主要是光合作用固碳、碳代谢、卟啉和叶绿素代谢等3条光合相关代谢通路。在FC≥2且FDR<0.01的高差异表达基因中筛选功能描述与光合作用相关的新基因共10个。其中Mulberry_newGene_3646、Mulberry_newGene_1405、Mulberry_newGene_2419和Mulberry_newGene_320共4個DEGs可能是造成H32和E1净光合速率和产量差异的关键基因,可重点研究。
关键词:桑树(Morus alba L.);光合特性;叶绿素荧光特性;转录组
中图分类号:S888 文献标识码:A
文章编号:0439-8114(2019)23-0101-11
DOI:10.14088/j.cnki.issn0439-8114.2019.23.023 开放科学(资源服务)标识码(OSID):
Research on mulberry photosynthesis transcriptome
LI Yong1,2,YU Cui2,MO Rong-li2,DENG Wen2,XIONG Chao2,ZHUANG Chu-xiong1,HU Xing-ming2
(1.College of Life Sciences,South China Agricultural University,Guangzhou 510642,China;
2.Economic Crops Research Institute,Hubei Academy of Agricultural Sciences,Wuhan 430064,China)
Abstract: The photosynthesis characteristics and photosynthetic related gene expression of high-yield mulberry varieties Esang 1 (E1) and control mulberry variety Husang 32(H32) were studied to provide a theoretical basis for high-efficiency breeding of mulberry trees and screening of germplasm resources. Using LI-6400XT mulberry leaf gas exchange and chlorophyll fluorescence parameters, chlorophyll content and ribulose carboxylase(RUBP) activity were measured simultaneously, transcriptome sequencing was performed on the peaks and valleys of mulberry photosynthesis curves, and the differences in photosynthetic related gene expression among different mulberry varieties were compared. The results of photosynthetic variation of E1 and H32 showed that the Pn peak of E1 leaves was significantly larger than that of H32(P<0.05), and the peak of Tr of H32 leaves was significantly larger than that of E1 (P<0.05). In the Pn-PAR and Pn-Ci responses of different mulberry leaves, CE and CSP were E1>H32, AQY, LCP, LSP and CCP were H32>E1. The ability of E1 to achieve photosynthetic product accumulation under weaker light intensity is stronger than that of H32, and the utilization efficiency of E1 to low concentration CO2 is better than that of H32; among the chlorophyll fluorescence parameters of different mulberry varieties, ΦPSII and qP of E1 leaves were significantly higher than H32 (P<0.05), and NPQ of H32 was significantly larger than E1 (P<0.05), indicating that the light energy absorbed by H32 leaves was mainly dissipated in the form of heat energy; the chlorophyll a, chlorophyll b and chlorophyll content of H32 were significantly lower than E1(P<0.05), and the RUBP activity of E1 was higher than that of H32(P>0.05), indicating that the photosynthetic capacity of E1 was better than that of H32. 3 356 DEGs were found in different varieties and different time periods. The metabolic pathway analysis showed that there were 1 136 DEGs annotated to the KEEG database. The photosynthesis-related metabolic pathways significantly enriched by DEGs were mainly photosynthetic carbon sequestration, carbon metabolism, porphyrin and chlorophyll metabolism. Photosynthetically related metabolic pathways. A total of 10 new genes related to photosynthesis were screened for highly differentially expressed genes with FC≥2 and FDR<0.01. The four DEGs, Mulberry_newGene_3646, Mulberry_newGene_1405, Mulberry_newGene_2419 and Mulberry_newGene_320, may be the key genes causing the difference in net photosynthetic rate and yield between H32 and E1, which can be studied intensively.
Key words: mulberry; photosynthetic characteristics; chlorophyll fluorescence characteristics; transcriptome
高等植物光合作用是其物质生产的基础[1],过程复杂,受诸多因子影响,其中,植物自身的光合特性起主要作用。研究植物光合作用特性,探讨高光效机制,在理论和实践上都具有重大意义。
桑树(Morus alba L.)桑属桑科落叶乔木或灌木。桑叶作为家蚕(Bonbyx mori L.)的饲料,是养蚕业的重要基础。桑叶还具有较高的营养和药用价值,被卫生部列为药食同源品种。已有桑树光合作用研究多集中于逆境胁迫和栽培对光合特性的影响方面,鲜见自然条件下不同品种桑树光合特性比较及其关键基因表达调控研究报道。为系统和深入地探究自然条件下不同品种桑树光合作用及产量差异机制,以主推高产桑品种鄂桑1号(E1)和对照桑品种湖桑32号(H32)为材料,比较光合作用上的主要差異,通过转录组分析差异涉及的基因及基因表达特征,探究自然条件下不同桑品种光合特性及其产量差异机制,为桑树高光效育种及种质资源筛选提供理论依据。
1 材料与方法
1.1 材料
鄂桑1号(E1)和湖桑32号(H32)1996年栽植于湖北省桑树种质资源圃,典型黄棕壤性土,肥力适中,pH 5.6~6.5,有机质含量中等偏上,株行距133 cm×67 cm,中干养成。小区地面平整、土地肥力均一,每小区选3株干周、冠径、树势基本一致的桑树为试材,重复3次,按高产桑园进行肥水管理,夏伐,强化病虫害防治和疏芽,确保良好的产叶群体结构。2014—2017年进行调查。
1.2 测定方法
1.2.1 光合生理参数测定 选样株中3个光照良好的顶部新梢,每新梢选1片功能正常叶片(5~7位叶片),用便携式光合测定仪(美国LI-COR公司,LI-6400XT型)测定桑叶净光合速率(Pn)、气孔导度(Gs)、胞间二氧化碳浓度(Ci)、蒸腾速率(Tr)[2]。5月12—13日6:00—18:00每隔2 h测定1次。光合有效辐射(PAR)从0~1 800 μmol/(m2·s)设定14个梯度,大气CO2浓度为400 μmol/mol。以直线回归(0~200 μmol/(m2·s))求得Pn-PAR响应曲线的初始斜率dPn/dPAR,即为表观光合量子效率(AQY),同时计算光补偿点(LCP),饱和光强(LSP)利用拟合曲线方程(y=ax2+bx+c)计算[3]。Ci设定12个浓度梯度(0~1 500 μmol/mol),PAR为1 200 μmol/(m2·s)。以直线回归(0~200 μmol/mol)求得Pn-Ci响应曲线的初始斜率dPn/dCi,即为羧化效率(CE)。CO2补偿点(CCP)与饱和点(CSP)以Pn-Ci响应曲线方程计算[4,5]。
1.2.2 叶绿素荧光参数测定 5月15—16日用便携式光合测定仪(美国LI-COR公司,LI-6400XT型)配备的荧光叶室测定叶绿素荧光参数。叶位、叶片选择同“1.2.1”。试验前用锡箔纸将待测叶片包裹进行12~24 h遮光处理,5:30—7:00准确测定,重复6次。测定最小初始荧光(Fo)、暗适应最大荧光(Fm)、最大荧光(Fm′)、初始荧光(Fo′)。PSⅡ最大光化学效率(Fv/Fm)、PSⅡ的实际光化学效率(ΦPSⅡ)、光化学猝灭系数(qP)、非光化学猝灭(NPQ)以及PSⅡ的电子传递速率(ETR)由仪器自动计算给出。
PSⅡ最大量子效率Fv/Fm=(Fm-Fo)/Fm[6];PSⅡ实际量子效率ΦPSⅡ=△F/Fm′=(Fm′-Fs)/Fm′[6];表观电子传递速率ETR=0.5×0.84×ΦPSⅡ×PPFD[7];光化学淬灭系数qP=(Fm′-Fs)/(Fm′-F0′);非光化学淬灭NPQ=(Fm-Fm′)/Fm′。
1.2.3 理化指标测定 叶绿素含量测定采用丙酮提取法[8];1,5二磷酸核酮糖羧化酶(RUBP)活性测定采用分光光度计法[9]。
1.2.4 光合转录组测序分析
1)RNA提取及转录组测序。分别在桑树光合日变化曲线峰值和谷底取样,液氮冻存,干冰冻存送北京百迈客生物科技有限公司进行RNA提取及转录组测序。
2)测序数据分析。用To-pHat2软件将过滤Raw
reads后得到的Clean reads与桑树基因组(https://www.ncbi.nlm.nih.gov/genome/17692)进行序列比对。用Cufflinks软件拼接Mapped Reads,并与已有基因组注释信息比较,用BLAST软件[10]进行基因NR[11],Swiss-Prot[12],GO[13],COG[14],KOG[15],Pfam[16],KEGG[17]数据库序列比对,用KOBAS2.0[18]得基因KEGG Orthology结果,用HMMER[19]软件与Pfam数据库比对氨基酸序列,获得基因注释信息。将表达差异倍数Fold Change≥2且FDR<0.01作为筛选标准,使用Cufflinks对基因表达水平进行定量,以FPKM表示。以Fold Change≥2且错误发现率FDR<0.01为标准,筛选出2个光合相关差异表达基因,进行GO分类和功能统计,KEGG注释Unigene的Pathway。
1.3 实时荧光定量PCR
实时荧光定量PCR验证内参基因为桑树ACTIN,使用在线软件设计引物(http://www.ncbi.nlm.
nih.gov/tools/primer-blast/),要求PCR产物应尽可能跨越外显子-外显子连接,以忽略DNA污染和选择性剪接。用DNaseI处理总RNA,用Oligo(dT)(Takara,Japan)翻转录第一链cDNA。qRT-PC使用SYBR Green Supermix iTaq试剂盒。20 μL反应体系,反应条件为95 ℃ 3 min,94 ℃ 10 s,55 ℃10 s,72 ℃ 30 s,40个循环。相对定量采用2-ΔΔCt方法[20]计算。
1.4 数据分析
所有数据均取平均值,用Microsoft Office Excel 2007和SPSS 19.0软件进行统计分析。
2 结果与分析
2.1 不同桑树品种叶片光合日变化
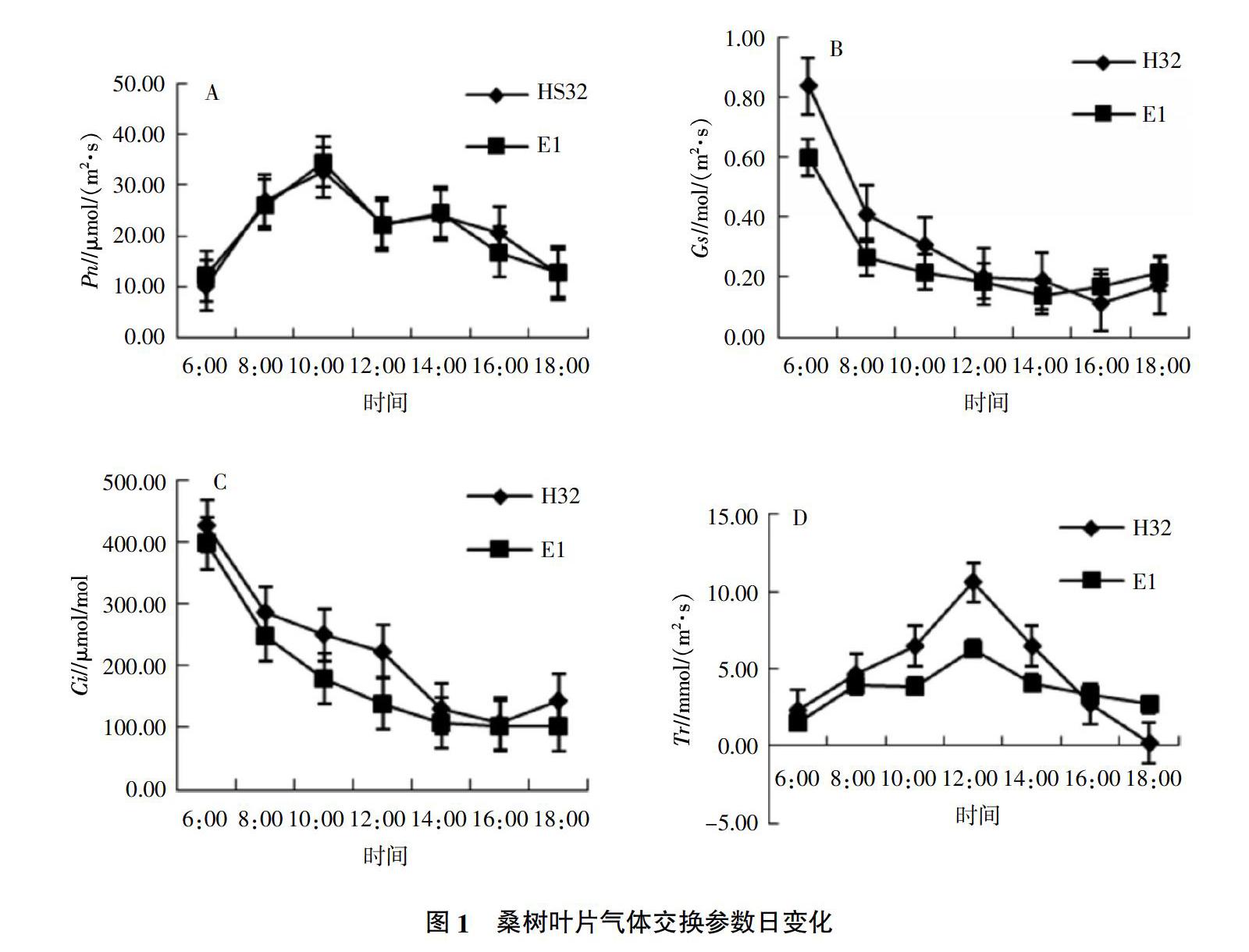
6:00—18:00 Pn均呈典型的双峰曲线(图1A),第一峰值出现在10:00,第二峰值出现在14:00,E1和H32 Pn峰值分别为34.56 μmol/(m2·s)和32.65 μmol/(m2·s),E1叶片Pn峰值显著大于H32峰值(P<0.05);Gs和Ci均呈逐渐下降趋势(图1B,图1C),Tr为单峰曲线(图1D),从6:00开始上升,12:00达最大值,之后开始下降,18:00降至最低,其中E1和H32叶片Tr峰值分别为6.30 mmol/(m2·s)和10.56 mmol/(m2·s),H32叶片Tr峰值显著大于E1峰值(P<0.05)。
2.2 不同品种桑树叶片Pn-PAR和Pn-Ci的响应
CE、CSP E1大于H32,AQY、LCP、LSP、CCP H32大于E1(表1)。H32和E1 LSP分别为1 500、 1 400 μmol/(m2·s),LCP则分别为47.930、31.182 μmol/(m2·s),E1在较弱光强下光合产物积累能力强于H32。H32、E1的CCP分别为74.618、68.724 μmol/mol,表明E1对低浓度CO2利用效率优于H32。2个品种CSP均在1 100 μmol/mol以上,且 E1大于H32,分别为1 212.5、1 162.5 μmol/mol,表明E1利用CO2浓度范围大于H32[21]。
2.3 不同桑树品种叶片叶绿素荧光参数
初始荧光Fo的变化可以反映PSⅡ反应中心的状况或者热能耗散的状况,Fm为黑暗中的最大荧光,指已经过完全暗适应后的光合机构全部PSⅡ反应中心都关闭时的荧光强度,这时所有的非光化学过程均达到最小[22]。由图2A、图2B知,2个桑品种的Fo和Fm无显著差异(P>0.05)。Fv/Fm是PSⅡ最大的光化学量子产量,其大小反映了PSⅡ反应中心原初光能的转化效率,是反映光合机构生理状态的重要荧光参数[23]。由图2C可以看出,E1和H32叶片的初始荧光均没有显著变化。ΦPSⅡ是PSⅡ实际的光化学量子产量,反映被用于光化学途径的激发能占进入到PSⅡ总激发能的占比,是植物光合能力的重要指标,而光化学淬灭系数qP反映的是PSⅡ天线色素所吸收的光能被用于光化学反应的光能,要使光化学淬灭系数保持较高的状态,就要使PSⅡ反应中心处于“开放”,因此光化学淬灭系数在一定程度上也反映了PSⅡ反应中心的开放程度[24,25]。由图2D、图2E可知,不同品种桑树ΦPSⅡ和qP各异,E1叶片ΦPSⅡ和qP显著大于H32(P<0.05)。非光化学淬灭系数NPQ反映的是PSⅡ天线色素所吸收的光能没有被用于光合电子传递而以热的形式耗散掉的部分[26],而ETR为表观光合电子传递速率[27]。由图2F、图2G知,不同品种桑树NPQ和ETR各异,H32叶片ΦPSⅡ和qP显著大于E1(P<0.05)。
2.4 不同桑树品种叶片叶绿素含量及RUBP活性
叶绿素是植物进行光合作用时吸收和传递光能的重要功能物质,其含量的高低反映植物光合作用强弱[28];1,5二磷酸核酮糖羧化酶(RUBP)是光合作用关键酶,在卡尔文循环中催化二氧化碳固定,生成2分子3-磷酸甘油酸(3-PGA),还可催化将氧加在RUBP的C-2位置下生成1分子的磷酸乙醇和1分子的3-磷酸甘油酸,反应速率由氧气和二氧化碳的浓度比调节,即调节光合作用和光呼吸平衡,决定净光合速率,影响光合作用的强弱[29]。叶绿素含量和RUBP活性情况见图3。由图3A可知H32的叶绿素a、叶绿素b和叶绿素含量均显著小于E1(P<0.05),图3B显示,E1的RUBP活性大于H32,但差异不显著(P>0.05)。
2.5 不同桑树品种光合转录组测序分析
2.5.1 不同品种桑树差异表达基因统计 H32样品2个时段测序得到的Clean reads数分别为23 136 096条和27 147 665條,E1样品2个时段测序得到的Clean reads数分别为27 641 855条和30 242 866条,Q30碱基占比均超过94%,测序质量较好。对2个桑品种10:00(TE)和12:00(TW)(H32-10vsH32-12)样品的基因进行差异表达分析(图4),H32 10:00和12:00相比,共有507个差异表达基因,其中表达上调的有297个,下调的有210个;对E1 10:00和12:00(E1-10vsE1-12)样品的基因进行差异表达分析,10:00和12:00相比,共有585个差异表达基因,其中表达上调的有339个,下调的有246个。对10:00 H32和E1样品的基因(H32-10vsE1-10)进行差异表达分析。H32和E1相比,共有1 179个差异表达基因,其中表达上调的有781个,下调的有398个。对12:00 H32和E1样品的基因(H32-12vsE1-12)进行差异表达分析。H32和E1相比,共有1 085个差异表达基因,其中表达上调的有612个,下调的有473个。
对2个品种2个时段共有差异表达基因进行GO功能分析,如图5。图5展示的是在差异表达基因背景和全部基因背景下GO各二级功能的基因富集情况,体现两个背景下各二级功能的地位,具有明显比例差异的二级功能说明差异表达基因与全部基因的富集趋势不同。差异基因在支链氨基酸合成过程、花青素生物合成过程、细胞器组装、跨膜运输、防御反应信号通路、甘氨酸合成过程等有显著富集。
2.5.2 不同品种桑树差异表达基因KEEG分类注释及光合作用相关基因差异表达 对2个品种2个时段差异表达基因代谢通路分析结果见图6。H32-10vsH32-12共有232个DEGs注释到KEEG数据库,E1-10vsE1-12共有190个DEGs注释到KEEG数据库,H32-10vsE1-10共有380个DEGs注释到KEEG数据库,H32-12vsE1-12共有334个DEGs注释到KEEG数据库。但不同桑品种不同时段注释到光合作用碳固定、碳代谢、卟啉和叶绿素代谢等3条光合相关代谢通路的DEGs各异(表2)。生化过程代谢通路光合作用相关基因表达情况见表3,E1-10vsE1-12的光合作用碳固定中,描述为1,4-双羟基-2-环烷酸聚丙基转移酶的DEGs上调,而H32-10vsH32-12没有;E1-10vsE1-12的卟啉和叶绿素代谢中,描述为叶绿素酶的DEGs下调,而H32-10vsH32-12没有;品种间H32-12vsE1-12的光合作用碳固定中,描述为磷酸丙酮酸二激酶的DEGs下调,描述为依赖NADP的苹果酶上调,但H32-10vsE1-10的光合作用碳固定中,该2个DEGs与H32-12vsE1-12的相同,但描述为光系统q(b)蛋白和磷烯醇丙酮酸羧化酶的DEGs分别呈下调和上调;H32-12vsE1-12的卟啉和叶绿素代谢中,描述为叶绿素-1的DEGs上调,而H32-10vsE1-10中则无DEGs出现。
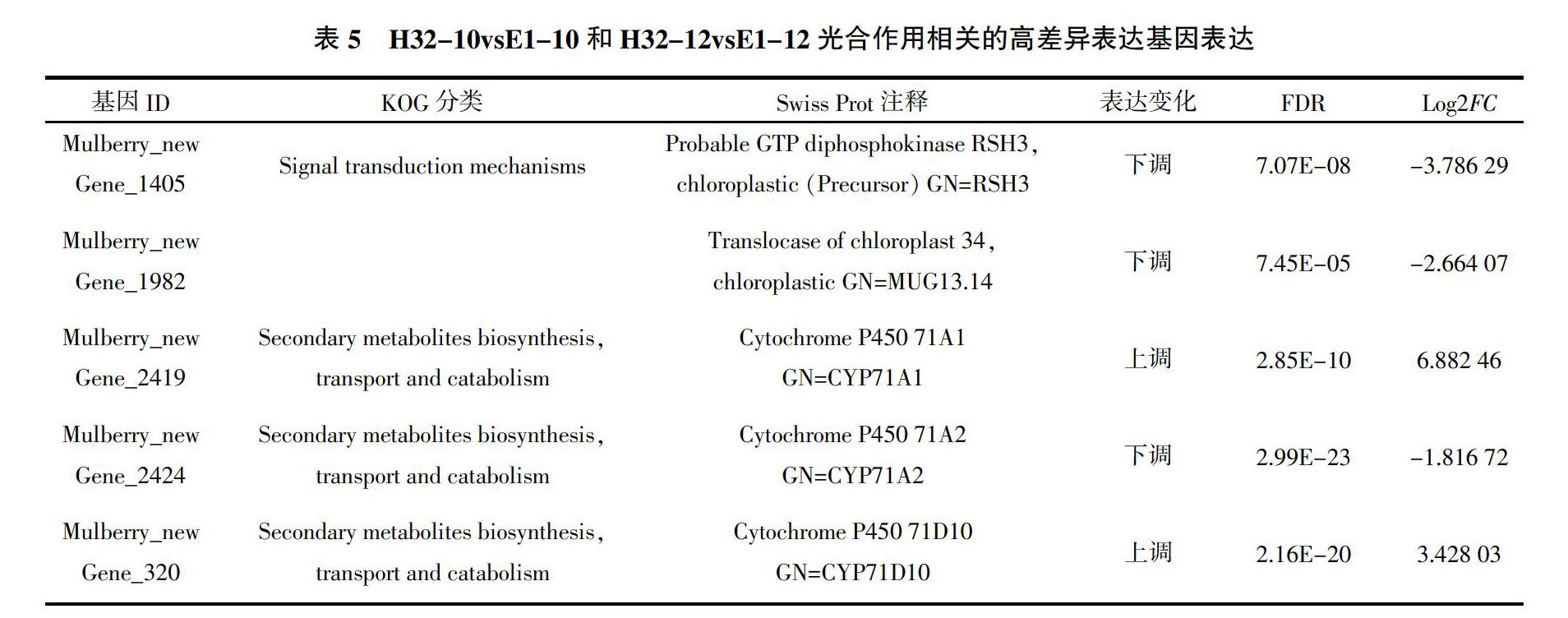
2.5.3 光合相关基因筛选 对H32-10vsH32-12和E1-10vsE1-12、H32-10vsE1-10和H32-12vsE1-12 GO数据库功能注释的1 092个和2 264个DEGs进行分析,在FC≥2且FDR<0.01的高差异表达基因中筛选功能描述与光合作用相关的基因共10个。其中H32-10vsH32-12和E1-10vsE1-12、H32-10vsE1-10和H32-12vsE1-12中均包括3个下调基因和2个上调基因。10个DEGs在KOG class和Swiss Prot annotation的功能描述统计见表4、表5。从表4可以看出,H32-10vsH32-12和E1-10vsE1-12中的5个DEGs,KOG class为碳水化合物的运输和代谢、防御机制、代谢产物的生物合成、运输和分解代谢的3个基因表达下调,而Swiss Prot注释为Glucose-6-phosphate 1-dehydrogenase,chloroplastic (Precursor)和Glucose-6-phosphate 1-dehydrogenase,chloroplastic(Precursor)表达上调;从表5可以看出 H32-10vsE1-10和H32-12vsE1-12中,Swiss Prot annotation注释为Probable GTP diphosphokinase RSH3, chloroplastic(Precursor) GN=RSH3和Translocase of chloroplast 34,chloroplastic GN=MUG13.14以及Cytochrome P450 71A2 GN=CYP71A2的表达下调,而注释为Cytochrome P450 71A1 GN=CYP71A1和Cytochrome P450 71D10 GN=CYP71D10的2个DEGs则表达上调。推测上述10个基因是造成同一品种不同时段、同一时段不同品种间净光合速率差异的原因。
2.5.4 荧光定量PCR验证 选择上述10个同一品种桑树不同时段、同一时段不同品种桑树的DEGs进行荧光定量PCR,结果见图7。同一品种桑树不同时段(图7A、7B、7C、7D、7E)的5个DEGs除Mulberry_newGene_3423外,其他均呈显著性差异(P<0.05),而同一时段不同桑品种(图7F、7G、7H、7J)5个DEGs均呈显著性差异(P<0.05),与测序结果分析基本一致。推测上述9个基因是造成同一品种不同时段、同一时段不同品种间净光合速率差异的原因。其中同一品种不同时段中的Mulberry_newGene_3646,H32在10:00和12:00时的表达量均显著小于E1,而同一时段不同桑品种中的Mulberry_newGene_1405、Mulberry_newGene_2419和
Mulberry_newGene_320,E1在10:00的表达量显著大于H32,上述4個DEGs可能是造成H32和E1净光合速率和产量差异的关键基因,可重点研究。
3 讨论
对E1和H32的光合日变化测定结果显示,2个桑品种的气体交换参数各异,但高产桑品种E1叶片的Pn峰值显著大于H32的Pn峰值(P<0.05),H32叶片Tr峰值显著大于E1的Tr峰值(P<0.05);不同品种桑树叶片Pn-PAR和Pn-Ci的响应中,除CE、CSP为E1大于H32外,AQY、LCP、LSP、CCP均为H32大于E1。说明E1在较弱光强下实现光合产物积累的能力强于H32,E1对CO2低浓度的利用效率优于H32。不同桑树品种叶片叶绿素荧光参数中,E1叶片ΦPSⅡ和qP显著大于H32(P<0.05),而H32的NPQ显著大于E1(P<0.05),表明H32叶片吸收的光能较多地以热能的形式进行耗散;且H32的叶绿素a、叶绿素b和叶绿素含量均显著小于E1(P<0.05),而E1的RUBP活性大于H32,但差异不显著(P>0.05)。表明E1光合能力优于H32。
對2个桑品种 10:00和12:00样品基因进行差异表达分析,发现3 356个DEGs,代谢通路分析显示共有1 136个DEGs注释到KEEG数据库,DEGs显著富集的光合作用相关代谢通路主要是光合作用固碳、碳代谢、卟啉和叶绿素代谢等3条光合相关代谢通路。根据对H32-10vsH32-12和E1-10vsE1-12、H32-10vsE1-10和H32-12vsE1-12 GO(Gene Ontology)数据库功能注释结果中的1 092个和2 264个DEGs进行分析,在FC≥2且FDR<0.01的高差异表达基因中筛选功能描述与光合作用相关的基因10个。其中H32-10vsH32-12和E1-10vsE1-12、H32-10vsE1-10和H32-12vsE1-12中均包括3个下调基因和2个上调基因。荧光定量PCR验证结果显示,同一桑品种不同时段中的Mulberry_newGene_3646,H32在10:00和12:00时的表达量均显著小于E1,而同一时段不同桑品种中的Mulberry_newGene_1405、Mulberry_newGene_2419
和Mulberry_newGene_320,E1在10:00的表达量显著大于H32,上述4个DEGs可能是造成H32和E1净光合速率和产量差异的关键基因,可重点研究。
参考文献:
[1] 许大全.光合作用效率[M].上海:上海科学技术出版社,2002.
[2] CHEN F G,CHEN L G,ZHAO H X,et al.Sex-specific responses and tolerances of Populus cathayana to salinity[J].Physiol plant 2010,140:163-173.
[3] PENUELAS J,FILELLA I,LLUSIA J,et al. Comparative field study of spring and summer leaf gas exchange and photobiology of the Mediterranean trees Quercusilex and Phillyrea latifolia[J].Journal of experiment botany,1998,49(319):229-238.
[4] BERRY J A,DOWNTON W J S. Environmental regulation of photosynthesis[A].Govindjee photosynthesis[M].New York:Academic Press,1982.
[5] LONG S P,BAKER N R,RAINS C A. Analyzing the responses of photosynthetic CO2 assimilation to long-term elevation of atmospheric CO2 concentration[J].Vegetatio,1993,104:33-45.
[6] GENTY B,BRIANTIS J M,BAKER N R.The relationship between the quantum yield of photosynthetic electron transport and quenching of chlorophyll fluorescence[J].Biochim Biophys Acta,1989,894:183-192.
[7] DEMMIG B,WINTER K,KRUGER A,et al.Photoinhibition and zeaxanthin formation in intact leaves:A possible role of the xanthophyllcycle in the dissipation of excess light[J].Plant physiology,1987,84:218-224.
[8] PORRA RJ,THOMPSON WA,KRIEDEMANN PE. Determination of accurate extinction coefficients and simultaneous equations for assaying chlorophylls a and b extracted with four different solvents:verification of the concentration of chlorophyll standards by atomic absorption spectroscopy[J].Biochimica et Biophysica Acta Bioenergetics,1989,975:384-394.
[9] BROUWER B,ZIOLKOWSKA A,BAGARD M, KEECH O,et al.The impact of light intensity on shade-induced leaf senescence[J].Plant cell and environment,2012,35:1084-1098.
[10] ALTSCHUL SF,MADDEN TL,ZHANG J,et al.Gapped BLAST and PIBLASTS:A new gene ration of protein database search programs[J].Nucleic acids rsearche ,1997,25(17):3389-3402
[11] DENG Y Y,LI J Q,WU S F,etal.Integrated nr database in protein annotation system and its localization[J].Computer engineering,2006,32(5):71-74.
[12] APWEILER R,BAIROCH A,WU CH,etal.The universal protein knowledge base[J].Nucleic aids rsearchce,2004,32:D115-D119.
[13] ASHBURNER M,BALL C A,BLAKE JA,et al.Gene ontology:tool for the unification of biology[J].Nature genetics,2000,25(1):25-29.
[14] TATUSOV R L,GALPERIN M Y,NATALE D A.The COG database:A tool for genome scale analysis of protein functions and evolution[J].Nucleic aids research,2000,28(1):33-36.
[15] KOONIN E V,FEDOROVA N D,JACKSON JD,et al.A comprehensive evolutionary classification of proteins encoded in complete eukaryotic genomes[J].Genomebiology,2004,5(2):R7.
[16] FINN R D,BATEMAN A,CLEMENTS J,et al.Pfam:The protein families database[J].Nucleic aids research,2014,42:D222.
[17] KANEHISA M,GOTO S,KAWASHIMa S,et al.The KEGG resource for deciphering the genome[J].Nucleic acids research,2004,32:D277-D280.
[18] XIE C,MAO X,HUANG J,et al.KOBAS 2.0:A web server for annotation and identification of enriched pathways and diseases[J].Nucleic aids research,2011,39:W316-W322.
[19] EDDY S R.Profile hidden Markov models[J].Bioinformatics,1998,14(9):755-763.
[20] LiVAK KJ,SCHMITTGEN TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method[J].Methods,2001,25:402-408.
[21] CALNEL M G R,THOMLEY J H M.Temperature and CO2 responses of leaf and canopy photosynthesis:A clarification using the non-rectangularHyperbola model of photosynthesis.[J].Annals of botany,1998,82:883-892.
[22] MORALES M,ABAD?魱A A,ABAD?魱A J.Photoinhibition and photoprotection under nutrient deficiencies,drought and salinity[A].DEMMIG A B,ADAMS W W,MATTOO A K. Photoprotection,gene regulation,and environment[M].Berlin:Springer-Verlag,2008.
[23] LU C M,ZHANG J H. Heat-induced multiple effects on PS II in wheat plants[J].J Plant Physiol,2000,156:259-265.
[24] EFEO■LU B,EKMEK?覶I Y,?覶I?覶EK N. Physiological responses of three maize cultivars to drought stress and recovery[J].J S Afr Bot,2009,75:34-42.
[25] SCHRADER S M,WISE R R,WACHOLTZ W F,et,al.Thylakoid membrane responses to moderately high leaf temperature in Pima cotton[J].Plant Cell Environ,2004,27:725-735.
[26] SHARKEY T D. Effects of moderate heat stress on photosynthesis:Importance of thylakoid reactions, rubisco deactivation, reactive oxygen species,and thermotolerance provided by isoprene[J].Plant Cell Environ,2005,28:269-277.
[27] FERREIRA S,HJERN?覫 K,LARSEN M,et al.Proteome profiling of Populus euphratica Oliv. upon heat stress[J].Ann Bot,2006, 98:361-77.
[28] MASCLAUX-DAUBRESSE C,REISDORF-CREN M,ORSEL M. Leaf nitrogen remobilisation for plant development and grain filling[J].Plant biology,2008,10,23-36.
[29] Parry M A. Rubisco activity and regulation as targets for crop improvement[J].J Exp Bot,2013,64(3):717-730.
