植物microRNA靶基因的预测与验证 技术研究进展
2018-08-20崔俊霞魏康宁谢伊源李用芳
崔俊霞,魏康宁,谢伊源,王 丽,李用芳
(河南师范大学 生命科学学院,河南 新乡 453007)
MicroRNA(miRNA)是一类真核生物体内普遍存在的具有重要调控功能的内源非编码小分子RNA (sRNA),长度在20~24 nt[1]。植物miRNA与植物的生长、发育、代谢以及胁迫存在着密切的联系[2],预测并验证miRNA调控的靶基因是阐明miRNA在生物体内功能的前提。植物miRNA主要通过剪切机制导致靶基因被降解,也可通过抑制翻译对靶基因进行调控,个别miRNA还可以通过诱导靶基因DNA甲基化来调控靶基因的表达[3]。在这3种机制中,miRNA介导的靶基因被剪切和翻译被抑制方面的研究较为深入,因此,可以根据miRNA对靶基因的作用方式对靶基因进行试验验证。简要介绍了植物miRNA产生过程的最新研究进展以及靶基因的预测方法,重点综述了植物miRNA靶基因的验证方法,旨在更深入地研究植物miRNA介导的转录后调控机制,并为利用miRNA改良植物品种提供理论指导。
1 植物miRNA的产生
植物MIRNA(miRNA基因)首先在细胞核内由Ⅱ型RNA聚合酶(Pol Ⅱ)转录出能形成发夹结构的初级转录物pri-miRNA[4-5],再在以DCL1 (Dicer-like 1)、HYL1(Hyponastic leaves 1)和SE (Serrate)为核心组分的切割复合体的作用下经第一次剪切形成pre-miRNA,经第二次剪切形成miRNA∶miRNA*复合体[6-8]。该复合体在甲基转移酶HEN1 (Hua enhancer 1)作用下3′端被甲基化[9],然后在HASTY蛋白的作用下被释放到细胞质中[10-11]。复合体miRNA∶miRNA*转运到细胞质后,成熟的miRNA与AGO1 (Argonaute 1)蛋白结合形成RNA诱导沉默复合体(RNA-induced silencing complex,RISC),miRNA通过碱基互补配对方式引导RISC识别靶基因并对靶基因进行调控,而miRNA*则被降解[12-14](图1)。然而近年研究发现,有些miRNA*的丰度比miRNA还高,甚至可调控靶基因并导致靶基因被剪切,表明miRNA*不仅仅是miRNA的伴随链,可能还有其他生物学功能[15-19]。
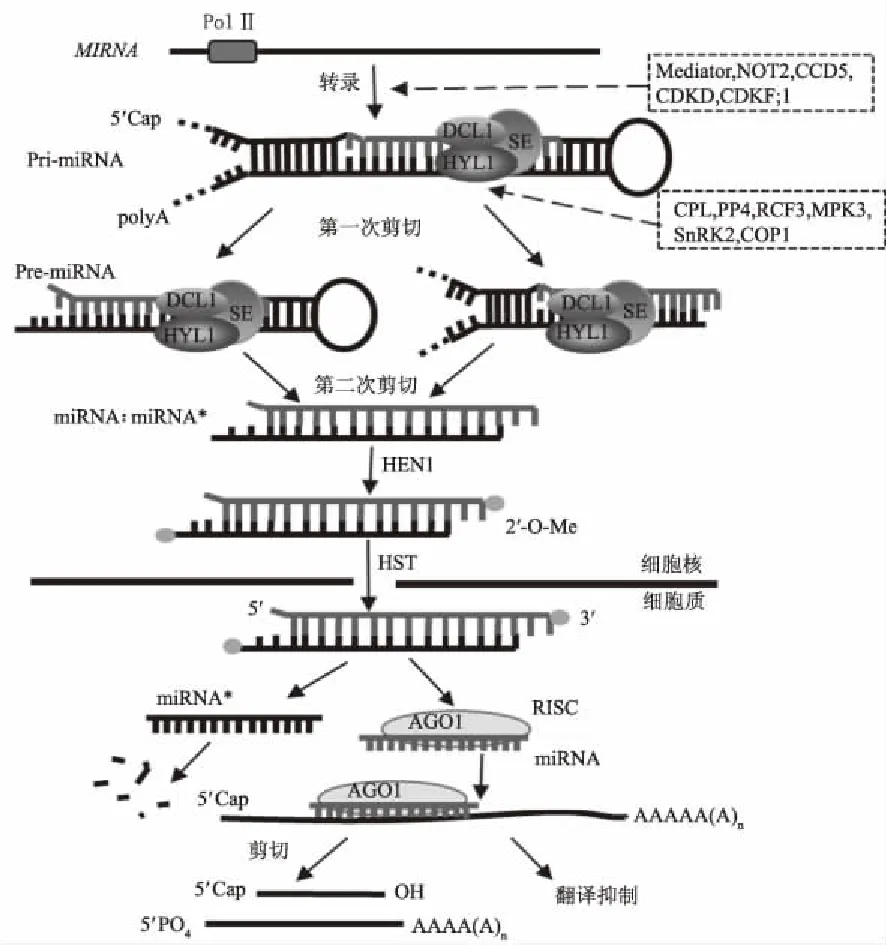
图1 植物miRNA的转录、成熟及对靶基因的调控作用
植物miRNA基因是一种非编码基因,近年来的研究发现,miRNA基因同编码基因一样也受到多种因子的调控。有些因子参与pri-miRNA的转录过程,如转录因子共激活因子Mediator 有助于Pol Ⅱ结合到MIRNA的启动子区域[20],转录因子NOT2(Negative on TATA less 2)、CCD5(Cell division cycle 5)及延伸复合体(Elongation complex)与Pol Ⅱ相互作用,可促进MIRNA的转录[21-23],此外,激酶CDKF;1(Cyclin-dependent kinase F;1)和CDKD (Cyclin-dependent kinase D)可通过磷酸化影响Pol Ⅱ的活性[24](图1)。另外一些因子参与miRNA的成熟过程,HYL1 有助于DCL1的精准剪切,最近研究表明,其活性和稳定性受磷酸化影响,CPL(C-terminal domain phosphatase-like)和PP4 (Protein phosphatase 4)通过对HYL1脱磷酸化稳定HYL1,从而促进miRNA的产生[25-26],RCF3 (Regulator of CBF gene expression 3) 可促进CPL对HYL1的脱磷酸化作用[27]。而激酶MPK3(Mitogen-activated protein kinase 3)和SnRK2 (SNF1-related protein kinase subfamily 2) 对HYL1的作用与CPL和PP4正好相反[28-29]。COP1 (Constitutive photomorphogenic 1) 受光信号影响在细胞质和细胞核中的移动也会影响HYL1的稳定性和生物活性[30](图1)。
此外,还有多种影响mRNA转录、拼接和成熟的蛋白质对miRNA的产生也有调控作用。Yu 等[31]根据作用特点将这些蛋白质分为2类:一类参与从pri-miRNA到miRNA的加工过程,如帽子结合蛋白CBP80、CBP20[22],STA1 (Stabilized 1)[32],SICKLE[33],RNA结合蛋白TOUGH和AtGRP7 (Glycine-rich RNA-binding protein 7)[34-35],PINP1 (PSR1-interacting protein 1)[36],MOS2 (Modifier of SNC1,2)[37],这些蛋白质基因突变导致体内miRNA丰度比野生型低,但miRNA前体累积;而另一类蛋白质基因的突变不仅导致miRNA的量减少,而且pri-miRNA的丰度也降低,PRL1(Protein pleiotropic regulatory locus 1) 和DDL (DAWDLE)[21-23]则属于这一类,这些基因不仅影响MIRNA的转录,也可能参与pri-miRNA的后续加工。
2 植物miRNA靶基因的预测
通常情况下植物miRNA与靶位点序列有着完美或近乎完美的互补配对,因此可以采用生物信息学方法预测靶基因[38]。预测的标准是根据两者的互补程度,通过罚分机制进行预测。在miRNA-靶位点之间1个错配碱基对罚分为1,G∶U摇摆配对罚分为0.5。在miRNA的2~12 nt的核心区域不允许超过1个错配碱基对,在10位和11位不允许有错配或G∶U配对,当miRNA与靶位点的罚分≤4时,认为该mRNA是miRNA的潜在靶位点[39-40]。miRU是第1个预测植物miRNA靶基因的软件[41],该软件后来升级为psRNATarget[42]。多种软件如Psrobot[40]、Targetfinder[43]、Target-align[44]、 p-TAREF[39]等陆续被研发用于植物miRNA靶基因的预测。此外,植物miRNA靶基因数据库PMTED (Plant miRNA target expression database)不仅可用于检索和分析miRNA靶基因的表达情况,还可用于预测新miRNA的靶基因并了解靶基因的表达信息[45]。这些软件为系统研究miRNA分子的功能提供了极大的便利,但是生物信息学预测的靶基因中存在大量的假阳性[46]。而有些真正的靶基因错配罚分高达5.5,甚至有些在miRNA的10位和11位之间存在错配或G∶U配对[47],因此预测植物miRNA靶基因的标准直接影响到预测结果的可信度。若采用罚分≤4,将丢失一些真正的靶基因,但若采用罚分<6,预测的靶基因中相当一部分并不是真正的靶基因,所以理论上预测的靶基因尚有待于进一步的试验验证。
3 植物miRNA靶基因的验证
植物miRNA靶基因的验证可以从miRNA对靶基因的作用方式入手。由miRNA介导的靶基因被剪切,可以用RLM-5′RACE(RNA ligase-mediated 5′rapid amplification of cDNA ends)[44]和降解组测序(Degradome sequencing)进行验证[48-50]。而miRNA介导的翻译被抑制则主要采用报告基因系统进行验证,根据验证靶基因的试验体系,又可分为瞬时共表达[47-51]和转基因系统[52]。
3.1 利用RLM-5′RACE 验证miRNA靶基因
miRNA介导的靶基因mRNA被降解时会产生2个片段(5′剪切片段和3′剪切片段),其中3′剪切片段比较稳定,有一个暴露的5′单磷酸基团和polyA尾巴。RLM-5′RACE和降解组测序均利用这2个特点对靶基因进行验证,并可对靶基因的剪切位点进行精准定位。
RLM-5′RACE的原理是用RNA连接酶在3′剪切片段的5′磷酸基团处连接一个RNA接头,然后用 Oligo(dT) 或基因特异性引物(GSP)进行反转录,再用与RNA接头对应的上游引物和基因特异性下游引物扩增mRNA断口附近的核苷酸序列,进一步通过克隆和测序分析来判断断口是否是miRNA的剪切位点,从而确定是否是miRNA的靶基因[48,53]。该方法具有相对简单、方便快捷等优点,已被广泛应用于植物miRNA靶基因的验证。但由于其验证结果单一,每个试验只能验证1个靶基因,如果要同时对多个或大量靶基因进行分析验证时,则存在工作量大、费时费力的缺点,随后发展起来的降解组文库测序分析则克服了这一弊端。
3.2 通过降解组测序验证miRNA靶基因
降解组测序技术结合了RLM-5′RACE、高通量测序和生物信息学分析的优势,可以对细胞或组织中miRNA介导的mRNA 的3′剪切片段进行深度分析[49-50]。Zhai等[54]对该技术进一步改进,将index序列引入不同文库中,因此可将多个降解组文库混合后测序。降解组测序技术包括文库构建、高通量测序和生物信息学分析。与RLM-5′RACE一样,首先连接一个RNA接头,该RNA接头的特点是3′端有IIS型限制性内切酶MmeⅠ的识别位点,经反转录得到cDNA和PCR简单扩增,然后用MmeⅠ对PCR产物进行酶切,再将此片段与3′双链DNA 接头连接并进行 PCR扩增,经纯化即得到降解组文库(图2)。再经高通量测序即可得到整个转录组水平miRNA剪切位点下游20~21个核苷酸序列[49-50]。
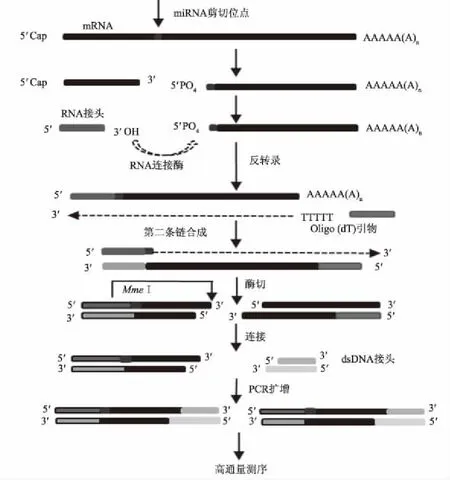
图2 降解组文库的构建
高通量测序产生的海量降解组序列需要结合生物信息学分析才能确定miRNA的靶标mRNA。目前常用的分析降解组文库的生物信息学软件有CleaveLand和SeqTar (SEQuencing-based sRNA TARget prediction),两者均是将降解组文库序列与待测物种的基因组序列或mRNA 数据库比对,舍弃不能匹配的序列,保留匹配的序列。其目的是将降解组序列匹配到待测物种的mRNA上,从而得到被剪切的mRNA的完整序列,由剪切位点向mRNA的5′端延伸即可得到完整的sRNA候选互补序列,再将此序列与测序物种的miRNA数据库比对,根据miRNA与靶位点的互补程度和第10位附近剪切信号强度确定潜在的miRNA靶基因[55-56]。CleaveLand严格限制了miRNA与靶位点间的碱基错配数目,不允许miRNA在10~11位与其靶基因存在错配或者G∶U摇摆配对;而SeqTar放宽了miRNA与靶位点互补配对出现错配的限定条件,允许miRNA的第10~11位存在错配或G∶U摇摆配对,并允许 miRNA有效剪切位点可对应于miRNA第9、10位及11位[56],这意味着CleaveLand会忽略一些真正的靶基因,而SeqTar通过放宽的标准可找到比CleaveLand更多的miRNA靶基因。
降解组测序是近年来研究植物miRNA靶基因的主要技术,具有高通量、高效率、低成本的特点,利用该技术已成功分析验证了拟南芥[49]、大豆[50]、水稻[47]、玉米[57]等20多种植物miRNA的靶基因[58-60]。
3.3 利用瞬时共表达系统检验miRNA靶基因
瞬时表达是一种高效的活体验证体系。应用瞬时共表达(Transient co-expression)验证miRNA与目的基因相互关系的原理是miRNA对靶基因的负调控作用,如果目的基因是miRNA的靶基因,则其表达蛋白质的量将受到过量表达的miRNA的限制,反之则不受影响。试验过程包括将miRNA前体和目的基因分别插入超表达载体,然后转化到农杆菌中,用注射或浸染方法使农杆菌感染烟草叶片下表皮细胞,之后即可检测外源基因的表达或蛋白质水平[47,51,56]。浸染时不仅有miRNA前体和目的基因共同浸染,同时有miRNA前体、目的基因和空载体单独浸染作为对照,可用Northern blot或RT-qPCR来检测目的基因mRNA表达水平的变化[47,56]。植物miRNA主要介导靶基因被降解,但是也存在翻译被抑制的现象,有些miRNA对翻译的抑制强度远远高于miRNA介导的靶基因降解[11,61],对于此类miRNA的靶基因可应用Western blot 技术进行验证,然而由于特异性抗体的限制,miRNA介导的靶基因翻译抑制主要是通过报告基因系统来验证。
双荧光素酶(Dual-luciferase)报告基因早已被广泛用于验证动物miRNA对靶基因的翻译抑制[62]。报告基因载体中含有2种能同时表达的荧光素酶基因,包括萤火虫萤光素酶基因(Fireflyluciferase,F-luc)和海肾萤光素酶基因(Renillaluciferase,R-luc),F-luc为主要报告基因,R-luc为内参报告基因。若目的基因片段被过量表达的miRNA抑制,则F-luc的翻译受阻,而R-luc的翻译不受影响,此时F-luc/R-luc活性比值下降,从而可以确定miRNA对目的基因具有直接调控作用。Liu等[51,57]根据植物miRNA与靶基因相互作用的特点构建了2个双荧光素酶表达载体系统,一个是将miRNA 靶位点序列插入F-luc的编码区,另一个是将miRNA 靶位点序列插入F-luc的3′UTR区(www.addgene.org,载体编号为55206、55207)。试验方法与前述的瞬时共表达一样,在烟草叶片细胞中进行,但是在构建表达载体时仅仅是将靶位点序列而非靶基因的序列克隆到表达载体中[51,57]。该技术的优势在于miRNA与靶基因的相互作用不仅可以在蛋白质水平(F-luc/R-luc活性)检测,而且还可以在mRNA水平(RT-qPCR)进行定量。此外,该方法是通过检测2种荧光素酶报告基因的蛋白质或mRNA水平来评判,因此该检测方法对所有的miRNA与靶位点都适用。
由于瞬时共表达不需要将外源基因整合到宿主染色体上,所以不受基因的位置效应和基因沉默的影响,因此具有操作简单、周期短、见效快和表达效率高的优势,且瞬时共表达不会产生可遗传的子代,生物安全性高,所以在分子生物学研究中被广泛应用。但这种方法与RLM 5′ RACE一样每次只能验证miRNA的单一靶基因,不适用于大量靶基因的验证,此外该技术根据miRNA与靶基因的负相关性判断目的基因是否是miRNA的靶基因,无法对miRNA的靶位点进行准确定位。
3.4 利用转基因植物验证miRNA的功能
Sunkar等[52]确认了miR398的靶基因Cu/Zn超氧化物歧化酶基因CSD1和CSD2,首次揭示了miRNA与胁迫的关系。通过对拟南芥转基因过量表达突变的miR398或对miR398具有抗性的CSD2,验证了miR398对CSD的转录后调控。贾小云等[63]以转基因番茄和野生番茄为材料,揭示了miR828与其靶基因SlMyb7-like的相关性。Chuck等[64]在柳枝稷中过量表达玉米miR156基因,结果发现,柳枝稷营养生长阶段延长且不开花,验证了其靶基因SPL调控生长发育进程。然而由于转基因需要周期长,该技术较少用于验证miRNA靶基因,其主要用途是靶基因的功能验证。
4 问题及展望
迄今为止,有关植物miRNA分析研究的报道很多,针对miRNA靶基因的预测及分析工具也不断涌现,这在一定程度上可以帮助人们挖掘miRNA的靶基因,但是如何提高预测靶基因的准确性是各种预测工具面临的共同问题。由于靶基因的试验验证技术要求高、费时费力、花费大,通过试验验证的miRNA靶基因大多属于保守型miRNA的靶基因,而验证的非保守型miRNA靶基因的数目非常有限,靶基因的验证已成为研究和应用miRNA的瓶颈。双荧光素酶报告基因系统能有效检测miRNA是否在翻译水平对靶基因有抑制作用,但是目前尚缺乏有效的从整体水平研究植物miRNA介导的翻译抑制的技术。降解组测序能有效地分析整个转录组水平miRNA介导的靶基因降解,但由于降解组测序只能得到20~21个有效碱基序列,有些片段能同时定位到多个位点,这些无法准确定位的潜在靶基因以及降解组分析预测的新靶基因还有待于采用RLM 5′RACE或瞬时共表达技术进一步验证,因此多种手段的综合利用是验证植物miRNA靶基因的趋势。此外,已验证的靶基因多是大量表达的保守型miRNA的靶基因,如何发现并验证低丰度miRNA的靶基因则是今后努力的方向。
