茶条槭SDH基因克隆及生物信息学分析
2017-12-19崔丽婷李文彦詹亚光齐凤慧
崔丽婷,李文彦,包 横,詹亚光,齐凤慧
(东北林业大学 生命科学学院,黑龙江 哈尔滨 150040)
茶条槭SDH基因克隆及生物信息学分析
崔丽婷,李文彦,包 横,詹亚光,齐凤慧
(东北林业大学 生命科学学院,黑龙江 哈尔滨 150040)
结合茶条槭转录组数据库,利用RT-PCR的方法克隆得到茶条槭SDH基因,命名为AgSDH,并对该基因及其蛋白进行了生物信息学分析。结果显示,AgSDH基因的ORF全长为1 563 bp,共编码520个氨基酸,推测蛋白相对分子量为57.219 kDa,理论等电点为5.61,无信号肽,预测亚细胞定位于细胞质中。生物信息学分析表明AgSDH蛋白为亲水性蛋白,没有跨膜结构。蛋白质二级结构α- 螺旋占35.38%,延伸链占18.65%,无规则卷曲占45.96%;三级结构预测表明AgSDH蛋白以单体形式存在。与目前已知的多种植物的SDH蛋白具有相似的结构功能域。系统进化分析表明,AgSDH蛋白与脐橙SDH蛋白亲缘关系最为接近。该研究结果为深入了解该基因的功能和茶条槭次生代谢产物合成的调控机理提供了基础数据。
茶条槭;SDH;生物信息学分析;表达载体构建
没食子酸(Gallic acid, GA),即3,4,5-三羟基苯甲酸,又称倍酸、五倍子酸,是在植物中发现的游离或酯化形式的主要内源性酚酸之一,是一种强效抗氧化剂,具有抗癌活性,对胃和肝具有保护作用[1]。
如果能通过基因工程手段提高植物中游离没食子酸的含量,则可大大提升这些植物的品质。而要做到这一点,也必须首先了解植物中GA的合成途径。生物中GA的合成途径有3个。早在1964年,就有研究表明途径C中的3,4,5-trihydroxycinn amic acid β氧化在植物中不存在[2]。利用14C标记的跟踪实验表明,在Acer buergerianum和Rhus succeedanea中GA的合成主要有2个途径,其一是通过对phenylpropanoid的 β氧化;其二是通过莽草酸(shikimic acid,SA)的脱氢反应[3]。
通常认为莽草酸途径发生于高等植物的质体(plastids)中,其第3、4步由一个双功能的酶,DHQ-SDH(dehydroquinate dehydratase-shikimate dehydro genase)来催化[4]。DHQ-SDH含有两个完全不同的功能域,DHQ 域和SDH 域。这两个域分别与细菌中单功能的 DHQ 和 SDH 相似[5]。有研究表明,SDH 不但能以SA为底物合成GA,也能以3-DHS为底物合成GA,表达核桃 SDH 的转基因烟草中GA提高了500%[6]。
茶条槭Acer ginnala Maxim.因含较高的没食子酸而被人们所关注,是生产天然没食子酸的重要替代原料。前期研究表明,外施信号分子可使茶条槭细胞没食子酸含量增加[7-8],同时合成途径的关键酶基因表达量有所增加。然而,茶条槭遗传信息相当薄弱,基础数据和基因组背景研究几乎为空白。因此,本研究根据转录组信息设计特异性引物,对关键酶基因SDH基因进行克隆,获得基因编码区全长,并对序列进行分析,构建植物表达载体,为今后利用遗传转化法提高没食子酸含量奠定基础。
1 材料与方法
1.1 材料与试剂
本实验所用材料取自东北林业大学森林生物工程实验室培养的茶条槭愈伤组织。总RNA用宝生物工程(大连)有限公司的 Plant RNA Extraction Kit试剂盒提取。反转录试剂Reverse Transcriptase M-MLV(RNase H-) 、PCR相关试剂、DNA marker、pMD18-T Simple Vector 载体、限制性内切酶和T4 DNA ligase均购自TAKARA-宝生物工程(大连)有限公司。DNA 琼脂糖凝胶回收试剂盒与质粒提取试剂盒均购自北京索莱宝科技有限公司。大肠杆菌DH5α感受态和pCAMBIA1301为实验室保存菌液,其他实验试剂为国产分析纯。引物合成和 DNA 测序由上海生物工程股份有限公司完成。
1.2 实验方法
1.2.1 茶条槭总RNA的提取与cDNA的合成
参照宝生物工程(大连)有限公司的 Plant RNA Extraction Kit试剂盒说明,提取茶条槭的愈伤组织细胞总RNA,检测其完整性及含量[9]。以0.5 μg总RNA为起始材料,参照Reverse Transcriptase M-MLV(RNase H-)说明合成反应所需要的cDNA第1链。
1.2.2 茶条槭SDH基因克隆及pMD18-T 载体的构建
以合成的单链 cDNA 为模板,结合茶条槭转录组和本地Blast比对结果,利用 Primer 5.0 设计 特 异 性 引 物 FW:SDH-F:5’-CGGGGTACC ATGGGTACTGTTGAAAT-3’,SDH-R:5’-CGGGGTACCTCAGAACTTGGCTAAAA-3’(下划线部为kpn I位点)进行 PCR 扩增。PCR扩增的反应体系为 20 μL,包含 cDNA 模板 2 μL,10×Buffer 2 μL,2.5 mmol/L dNTPs 2 μL,正反引物各1 μL,5 U/μL 高保真 Pfu 0.5 μL,ddH2O 11.5 μL。反应程序如下:94 ℃预变性 3 min,94 ℃变性40 s,50 ℃退火40 s,72 ℃延伸100 s,共进行30个循环,72 ℃再延伸10 min。反应终止后PCR产物经1.0% 琼脂糖凝胶电泳检测,回收目的片段,并与pMD18–T Simple Vector连接[10],连接体系为 10 μL,包含回收产物 3 μL,pMD18-T Simple Vector 1 μL,Solution I 5 μL,ddH2O 1 μL,16 ℃连接 1 h。采用热激法将连接产物转化至大肠杆菌,筛选出阳性克隆,送至上海生物工程股份有限公司测序。
1.2.3 茶条槭SDH基因序列及蛋白的生物信息学分析
将测序结果与NCBI数据库中的同源序列及实验室现有的茶条槭转录组中获得的SDH序列进行比对,并对茶条槭SDH蛋白结构域进行预测。同时,利用 protparam[11]在线软件计算氨基酸理化性质,各项指标包括:氨基酸数目、分子量、理论等电点、分子式和不稳度指数;通过在线 proscale[12]分析并预测蛋白亲/疏水性;通过SignalP[13]软件进行信号肽预测;WoLF PSORT[14]进行亚细胞定位分析;利用TMHMM Server.2.0软件对SDH蛋白进行了跨膜区分析[15];利用GOR4[16]分析二级结构;通过 Swiss-Model[17]进行三级结构分析,建立蛋白质空间结构模型;利用NetPhos2.0对其潜在的修饰位点进行预测;在NCBI数据库中查找不同物种中SDH基因的同源序列,利用MEGA 5.0 软件邻接算法,自检举1 000次,构建系统进化树[18]。
1.2.4 茶条槭SDH基因植物表达载体的构建
以提取的pMD18-T质粒为模版,扩增目的基因,PCR程序同上,并通过DNA纯化回收试剂盒进行纯化回收。利用限制性内切酶将纯化后的目的片段与pCAMBIA1301分别进行酶切,酶切产物纯化回收后用T4连接酶16 ℃过夜连接,连接产物转化至DH5α感受态细胞,在含有卡那抗生素的LB培养基上37 ℃过夜培养[19]。随机筛选白色菌斑进行PCR验证,验证成功的菌液送往上海生工进行测序,将测序结果与SDH基因序列及pCAMBIA1301载体序列进行比对,选取正向插入片段载体作为阳性克隆。
2 结果与分析
2.1 茶条槭SDH基因克隆
以茶条槭愈伤组织为材料提取总RNA的琼脂糖凝胶电泳(如图1),图1中18、28 s条带均清晰无弥散;经分光光度计检测A260/A280平均值为1.97,RNA纯度较高。以总RNA反转录得到的cDNA第1链为模板进行PCR扩增。扩增产物经琼脂糖凝胶电泳检测发现在2 000 bp左右处有1条特异性带(如图2),与推测的目的片段大小一致,将PCR产物链接到T载体中送去上海生工测序,测序结果表明载体构建成功,插入的片段编码区为1 820 bp。

图1 茶条槭总RNA凝胶电泳图谱Fig.1 Ginnala total RNA gel electrophoresis

图2 SDH基因的PCR扩增Fig.2 PCR ampli fi cation of SDH
ORFfi nder推导基因序列含1条长1 563 bp的开放阅读框,共编码520个氨基酸(如图3),编码的氨基酸序列与脐橙Citrus clementina(XP_006433146)、葡萄Vitis vinifera(XP_002270055)、胡杨Populus euphratica(XP_011048876)、桉树Eucalyptus grandis(XP_010041998)SDH蛋白的相似性分别为78%,74%,73%,71%,确定为茶条槭SDH基因编码区全长,命名为AgSDH。
2.2 AgSDH基因编码蛋白特性分析
2.2.1 蛋白质理化性质分析
利用分析软件Protparam分析显示AgSDH蛋白编码520个氨基酸,理论分子量为57.257 kDa,等电点(pI)为5.61,预测分子式为C2571H4061N685O753S20,以异亮氨酸(8.8%)、丙氨酸(8.7%)和缬氨酸(8.5%)含量为最高,脂溶指数为96.94,蛋白不稳定系数为37.54(不稳定系数小于40时,预测蛋白质稳定,反之则不稳定),属于稳定蛋白质。
2.2.2 亲水区域/疏水区域预测
采用在线分析软件ProtScale的Kyte & Doolittle标度计算,得到AgSDH的亲/疏水信号(如图4)。结果表明,AgSDH蛋白在第374位疏水性最强,最高值为2.156;在119处有最低值为-2.711,亲水性最强,共有37个疏水区和35个亲水区。蛋白峰值的分布在0.5以上的比在-0.5以下的要少,表明蛋白均为亲水蛋白。
2.2.3 信号肽、亚细胞定位及跨膜区预测分析
信号肽是 N 端的一段氨基酸序列,指导分泌性蛋白到内质网膜上合成,在蛋白质合成结束之前被切除,信号肽位于蛋白质的 N 端,一般由16~26 个氨基酸残基组成,其中包括疏水核心区、信号肽的 C 端和 N 端。利用在线分析工具SignalP的神经网络算法BpNOS 蛋白进行预测。结果表明,AgSDH 蛋白在20和21的位置可能存在信号肽,说明 AgSDH 可被分泌出来,作用到受体上,属于分泌蛋白(如图 5)。利用在线工具 WoLF PSORT对AgSDH 蛋白进行亚细胞定位分析,AgSDH 蛋白在细胞质中得分为6。
利用TMHMM Server.2.0软件对SDH蛋白进行了跨膜区分析,结果表明该蛋白不含有跨膜区,是非跨膜蛋白。根据NCBI CDS(conserved domainsearch)分析发现,AgSDH是双功能3-脱氢脱水/莽草酸脱氢酶多结构域蛋白,包括3-脱氢脱水酶或DHQase I型结构域、莽草酸脱氢酶底物结合域、NAD(P)莽草酸脱氢酶的结合结构域(如图6)。

图3 SDH基因序列及其编码的氨基酸序列Fig.3 Analysis of sequence of nucleotide and its deduced amino acid for SDH gene
2.2.4 AgSDH蛋白二级结构及三级结构分析
蛋白质分子的多肽链通常折叠和盘曲成比较稳定的空间结构,形成比较稳定的二级结构,进一步才能完成活性功能域构想的建设,以完成特定的生命活动。利用在线软件GOR4对SDH蛋白二级结构进行预测(如图7),结果显示SDH氨基酸组成为α-螺旋(Hh)为184个(35.38%),延伸链(Ee)为97个(18.65%),无规则卷曲(Cc)为239个(45.96%)。并且可以推测 α-螺旋最有可能的位置是21~37,39~44,55~61,84~97,99~118,142~152,154~157,166~176,222~231,313~ 321,343~353,380~390,401~412,420~422,450~461,470~479,486~493,508~515。总体上SDH蛋白为不规则结构。利用软件Swiss-Model,以拟南芥A.thalianaDHQSDH蛋白(2o7s.1)为模板对AgSDH蛋白进行三维结构同源建模,AgSDH蛋白三维结构以单体形式存在(如图8)。

图4 AgSDH 基因的亲水性/疏水性分析Fig.4 Analysis of hydrophilic/hydrophobic AgSDH gene in B.platyphylla

图5 AgSDH 基因蛋白信号肽的预测和分析Fig.5 Prediction and analysis of AgSDH gene protein signal peptide in B.Platyphylla

图6 SDH蛋白保守域分析Fig.6 Conserved domain in malus SDH protein

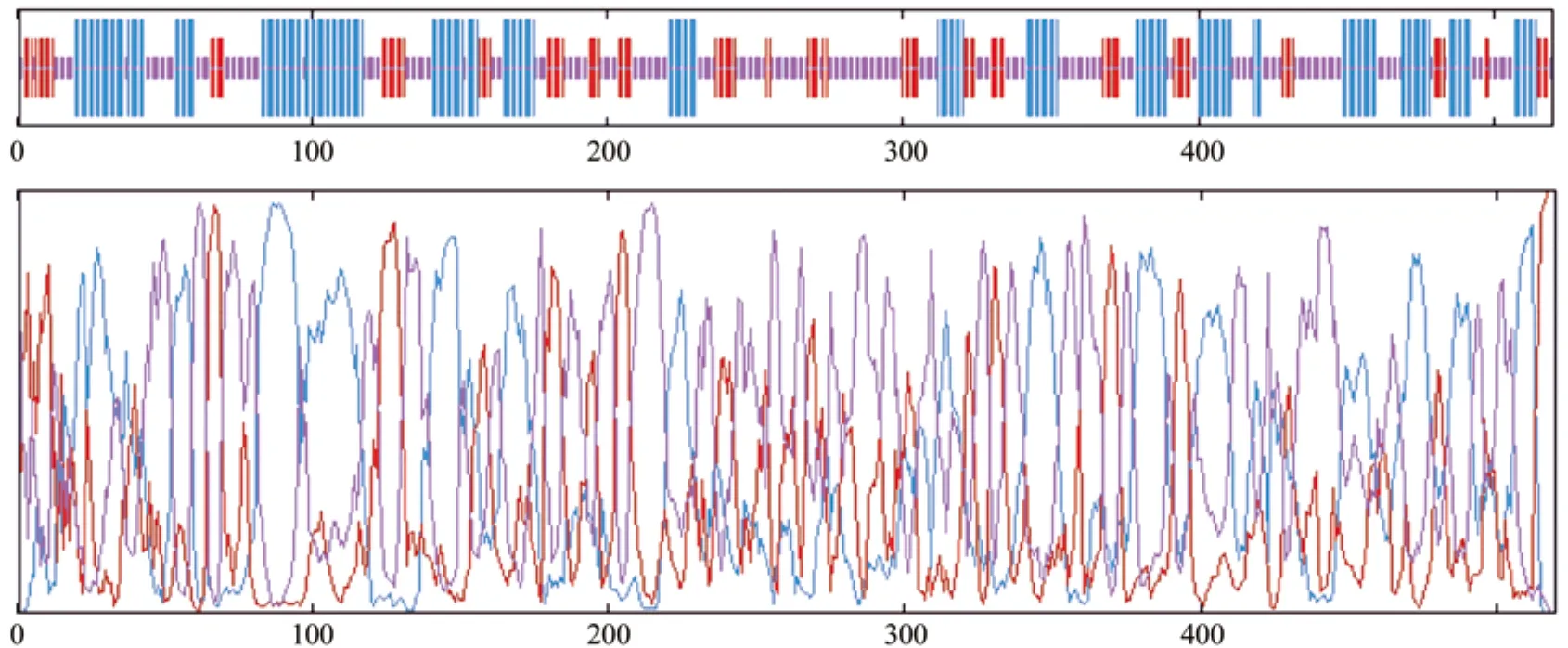
图7 SDH蛋白二级结构Fig.7 SDH protein secondary structure

图8 SDH蛋白三级结构Fig.8 SDH protein tertiary structure
2.2.5 AgSDH蛋白磷酸化修饰预测
利用NetPhos2.0对其潜在的修饰位点进行预测,发现SDH中分别含有18个(A21、A34、A138、A143、A187、A199、A210、A249、A251、A283、A286、A287、A328、A344、A364、A406、A409、A412)潜在的丝氨酸磷酸化位点,8 个(A124、A337、A367、A444、A453、A470、A471、A502)潜在的苏氨酸磷酸化位点和3个(A101、A270、A285)潜在的酪氨酸磷酸化位点(如图9)。
2.3 AgSDH同源蛋白聚类分析
氨基酸序列以及推测编码蛋白进行同源序列比对,经比对分析筛选出在其他植物中已经命名的SDH同源蛋白,这些蛋白主要存在于脐橙Citrus clementina、 葡 萄Vitis vinifera、 胡 杨Populus euphratica、桉树Eucalyptus grandis等中。这些蛋白同茶条槭SDH蛋白序列一致性达70% 以上;同时也选择了一些序列一致性在65% ~ 70%的蛋白序列,如大豆Glycine max、烟草Nicotiana tabacum等共11个同源序列,利用 MEGA5 软件的Neighbor- Joining算法进行系统发育树构建。结果(如图 10)可以看出不同来源的SDH蛋白属于不同的进化分支,裸子植物、单子叶植物及双子叶植物之间有较为清晰的界限。AgSDH蛋白与脐橙亲缘关系最为接近,进化距离为98,其次为葡萄。

图9 磷酸化位点预测Fig.9 Phosphorylation site prediction
2.4 茶条槭SDH基因植物表达载体的构建
利用限制性内切酶将纯化后的目的片段与pCAMBIA1301分别进行酶切,经1.0%琼脂糖凝胶电泳检测(如图11和图12),电泳结果表明已酶切完全。将转化有目的基因的菌液进行PCR验证(如图13),其中第二个点样孔点样品为质粒,后面六个点样孔缩点样品是三种不同引物的扩增结果,第一对和第三对引物扩增的基因片段在1 500 bp和2 000 bp之间有条带,说明菌体中转化有目的片段,验证成功的菌液送往上海生物工程股份有限公司进行测序,将测序结果分别与AgSDH基因编码区序列及载体两端序列进行比对,比对结果表明存在阳性克隆为正向插入片段。
3 讨 论
随着植物次生代谢物合成途径的研究进展,目前已在不同植物中分离出代谢途径相关的调控基因,如:樟叶越桔中的糖基转移酶VdUGT1基因[20],龙骨马尾杉中的调控萜类化合物的PcHDR1基因[21],马尾松的GGPPS基因[22],红豆杉中的bHLH基因[23]等。茶条槭中代谢途径SDH基因与其他代谢途径中的关键酶基因相比,序列数量较少,涉及到的蛋白活性、功能鉴定的研究工作还在起步阶段。
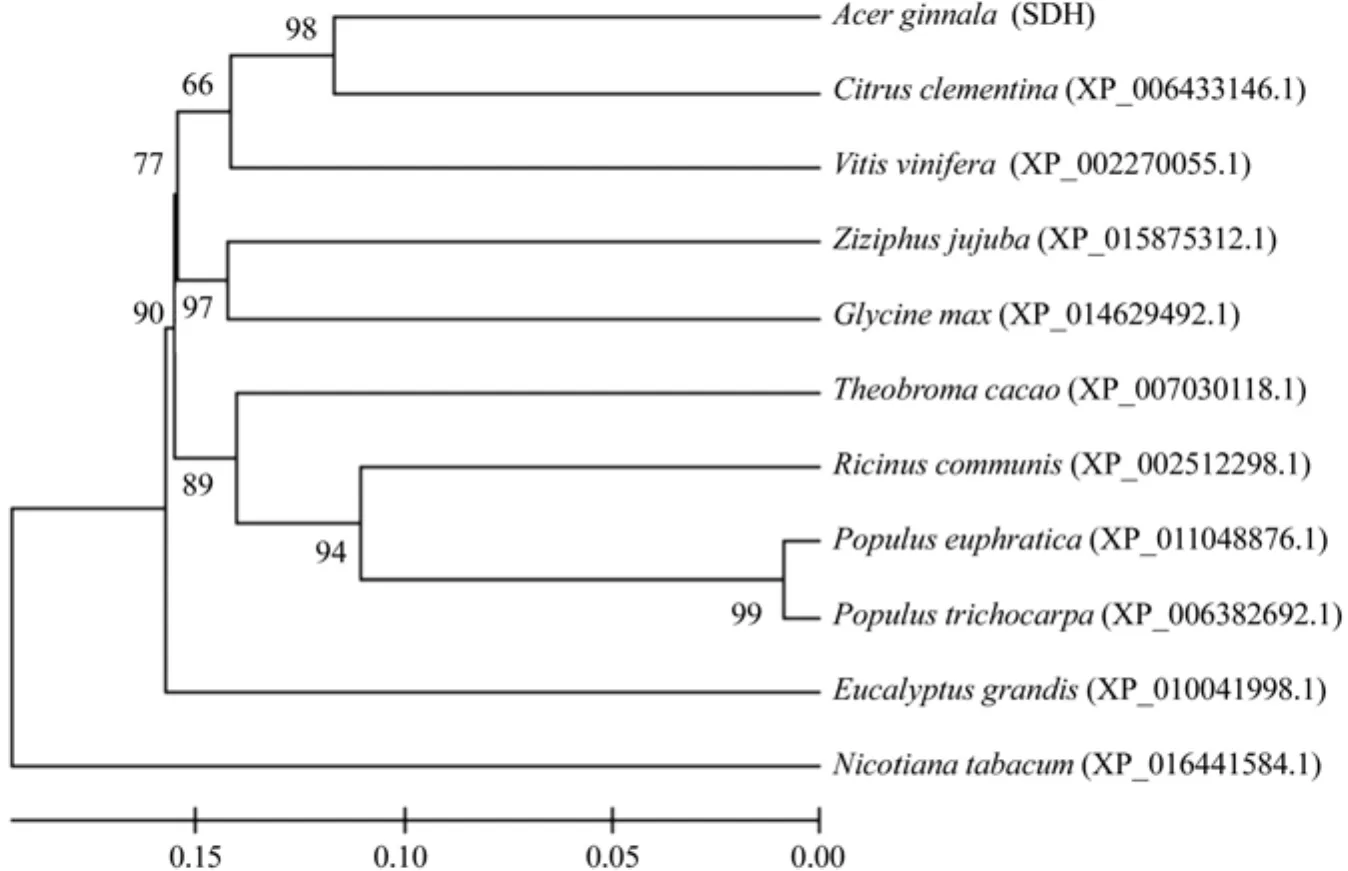
图10 AgSDH 基因系统发育进化树Fig.10 Phylogenetic tree of AgSDH gene in B.Platyphylla

图11 酶切pCAMBIA1301质粒电泳图Fig.11 PCAMBIA1301 digested plasmid electrophoresis
图12 酶切的基因片段电泳图Fig.12 The digested gene fragment electrophoresis pattern
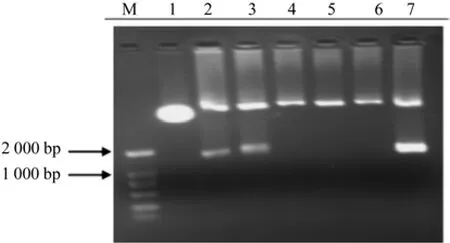
图13 菌液质粒PCRFig.13 Bacteria plasmid PCR
SDH 在代谢通路中,是催化莽草酸途径的一个关键酶。有研究发现用RNAi技术使烟草中DHQSDH(原文中称为NtDHD/SDH-1)(AAS90325)的活性低于对照的40%,莽草酸途径中下游的产物(如芳香氨基酸、木质素等)的产量减少,DHQ的积累增加,莽草酸的积累量也同时增加了。进一步的研究表明,在烟草中存在另一个可催化合成莽草酸的酶,DHQ-SDH-2(NtDHD/SDH-2)(AAS90324)。序列比对发现,二者的氨基酸序列相同性仅有48%。NtDHD/SDH-2仅存在于细胞液(cytosol)中,而NtDHD/SDH-1存在于质体中,因而二者的作用机制可能不同。此外,在土豆、杨树、大米等植物中也存在DHQ-SDH-2的相关序列[24]。在拟南芥中,研究人员通过气相悬滴法,第一次完成了DHQ-SDH酶和莽草酸的共结晶,之后将尼克酰胺腺嘌呤二核苷酸磷酸(NADP+)添加到晶体中形成三重复合物。脱氢莽草酸产物在DHQ位点产生,说明SDH-莽草酸-NADP(H)是一种利于莽草酸氧化的活性复合物,DHQ-SDH的凹型构造中存有活性位点。DHQ-SDH蛋白可以通过面对面定位,将莽草酸途径中的代谢物区分开,而且可增加代谢物从DHQ到SDH域的转移效率[25]。抗病毒药物奥司他韦(Oseltamivir) 的合成也受到莽草酸SDH的催化[26]。在微生物的生物合成中SDH也被广泛的应用[27-28]。
4 结 论
克隆得到的茶条槭SDH基因,ORF全长为1 563 bp,共编码520个氨基酸,推测其蛋白分子量为57.219 kDa,理论等电点为5.61,预测亚细胞定位于细胞质中,AgSDH蛋白为亲水性蛋白,没有信号肽和跨膜结构。该基因的获得,为深入研究其编码的酶学功能及活性提供了基础,在本研究的基础上,将SDH基因转化植物,研究其对酚类物质合成的影响,并分析该基因在植物代谢合成途径中的作用,具有重要的研究意义。
[1]冯立娟,尹燕雷,招雪晴,等.石榴没食子酸代谢与保健功能研究进展[J].果树学报, 2014, 31(4):710-716.
[2]M.H.Zenk, G.Müller.Über den Ein fl uss der Wund fl ächen auf die enzymatische Oxydation der Indol-3-essigsäure in vivo[J].Planta, 1964, 61(4): 346-351.
[3]Ishikura N, Hayashida S, Tazaki K.Biosynthesis of gallic and ellagic acids with 14 C-labeled compounds in Acer and Rhus leaves[J].Journal of Plant Research, 1984, 97(3): 355-367.
[4]Herrmann K M, Weaver L M.The Shikimate Pathway[J].Plant Biology, 1999, 50(50): 473-503.
[5]Singh, S., Kundu,S.S.,Negi, A S, et al.Cowpea (Vigna unguiculata) legume grains as protein source in the ration of growing sheep[J].Small Rumin.Res., 2006, 64(3): 247-254.
[6]Muir R M, Ibáñez A M, Uratsu S L, et al.Mechanism of gallic acid biosynthesis in bacteria (Escherichia coli) and walnut(Juglans regia)[J].Plant Molecular Biology, 2011, 75(6): 555-65.
[7]董 杰, 齐凤慧, 詹亚光.茶条槭悬浮培养体系的建立与没食子酸合成的优化条件[J].植物学通报, 2008, 25 (6):734-740.
[8]李海艳, 宋继园, 董 杰,等.茶条槭愈伤组织的再生体系建立及其没食子酸含量的测定[J].植物学通报, 2008, 25(2):212-219.
[9]庞新华, 张 宇, 黄国弟,等.菠萝叶片总RNA提取方法的比较[J].经济林研究, 2016, 34(1):153-157.
[10]杨 丹, 章怀云, 张党权,等.油桐LEC1基因的cDNA克隆与序列分析[J].经济林研究, 2016, 34(1):1-5.
[11]Burge C B, Karlin S.Finding the genes in genomic DNA[J].Current Opinion in Structural Biology, 1998, 8(3): 346-54.
[12]Gasteiger E, Hoogland C, Gattiker A, et al.The Proteomics Protocols Handbook[J].Humana Press, 2005, 71(6): 571- 607.
[13]Petersen T N, Brunak S, Von H G, et al.SIGNALP 4.0:discriminating signal peptides from transmembrane regions[J].Nature Methods, 2010, 8(10): 785-6.
[14]Horton P, Park K J, Obayashi T, et al.WoLF PSORT: protein localization predictor.[J].Nucleic Acids Research, 2007,35(2):585--587.
[15]宋 健, 熊 宏, 余进德,等.麻疯树油质蛋白JcOle14.3基因克隆及序列分析[J].中南林业科技大学学报,2016,36(6):15-22.
[16]Sen T Z, Jernigan R L, Garnier J, et al.GOR V server for protein secondary structure prediction[J].Bioinformatics, 2005, 21(11):2787-2788.
[17]付德山, 朱 珠, 崔丽婷,等.水曲柳 SOC1 基因表达载体的构建及生物信息学分析[J].西南林业大学学报,2015,35(6):6-13.
[18]Tamura K, Dudley J, Nei M, et al.MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0[J].Molecular Biology and Evolution, 2007, 24(8): 1596-1599.
[19]李蕾蕾, 孙丰坤, 李晓一,等.白桦BpGT14基因表达载体及RNA干扰载体的构建[J].中南林业科技大学学报, 2015,35(7):17-21.
[20]宋 健,熊 宏,朱东阳,等.樟叶越桔糖基转移酶VdUGT1基因克隆及序列分析[J].中南林业科技大学学报,2015, 35(6):80-86.
[21]张志利,海 舟,郭 溆,等.龙骨马尾杉PcHDR1基因克隆及序列分析[J].中国中药杂志,2016,41(22):4169-4174.
[22]陈博雯,覃子海,王鹏良,等.马尾松GGPPS基因克隆及序列分,西部林业科学[J].2016,45(2):1-6.
[23]周 华, 朱 祺, 杨艳芳,等.南方红豆杉bHLH基因克隆与序列分析[J].植物研究,2015, 35(1):52-59.
[24]Ding J, Kang G Z, Liu Y J, et al.Uniaxial time-dependent cyclic deformation of 6061-T6 aluminium alloy[J].Zhongguo Youse Jinshu Xuebao/chinese Journal of Nonferrous Metals, 2007,17(12): 1993-1998.
[25]Singh S A, Christendat D.The DHQ-dehydroshikimate-SDH-shikimate-NADP(H) Complex: Insights into Metabolite Transfer in the Shikimate Pathway[J].Crystal Growth & Design, 2007,7(11):2153-2160.
[26]Krämer M, Bongaerts J, Bovenberg R, et al.Metabolic engineering for microbial production of shikimic acid[J].Metabolic Engineering , 2003, 5(4): 277-283.
[27]Adachi O, Ano Y, Toyama H, et al.High shikimate production from quinate with two enzymatic systems of acetic acid bacteria.[J].Bioscience, Biotechnology, and Biochemistry, 2006,70(10):2579-2582.
[28]Liu X, Lin J, Hu H, et al.Metabolic engineering of Escherichia coli to enhance shikimic acid production from sorbitol.[J].World Journal of Microbiology and Biotechnology, 2014, 30(9): 2543-2550.
Cloning and Sequence Analysis of SDH Gene from Acer ginnala Maxim
CUI Liting, LI Wenyan, BAO Heng, ZHAN Yaguang, QI Fenghui
(Northeast Forestry University, Life Science College, Harbin 150040, Heilongjiang, China)
A Acer ginnala Maxim SDH gene was cloned by RT-PCR from the transcriptome database of Acer palmatum.The gene named AgSDH and the bioinformatics analysis of the gene and its protein was carried out.The results showed that the ORF of AgSDH gene was 1 563 bp, encoding 520 amino acids.The relative molecular mass of the AgSDH gene was 57.219 kDa, the theoretical isoelectric point was 5.61, and no signal peptide was detected.The subcellular localization was predicted in cytoplasm.Bioinformatic analysis showed that the AgSDH protein was a hydrophilic protein with no transmembrane structure.The secondary structure analysis showed that AgSDH comprised 35.38% of alpha-helix, 18.65 extended strains and 45.96% random coil.The tertiary structure prediction indicated that the AgSDH protein existed as monomer.And has similar structural domain to the SDH proteins of many known plant species.Phelogenetic analysis showed that the closest relative of AgSDH was that of Citrus clementina among known SDHs.The results provide the basic data for understanding the function of this gene and regulating mechanism of secondary metabolites synthesis.
Acer ginnala Maxim.; SDH; bioinformatics; expression vector construction
S718.46
A
1673-923X(2017)04-0044-08
10.14067/j.cnki.1673-923x.2017.04.008
2016-06-29
中央高校基本科研经费“茶条槭次生代谢合成途径调控基因表达谱分析” (DL13CA07);哈尔滨市青年科技创新项目“诱导子作用下茶条槭细胞次生代谢途径关键酶基因表达研究”(2013RFQXJ026)
崔丽婷,硕士研究生 通讯作者:齐凤慧,高级工程师,博士,硕士生导师; E-mail:qifenghui2001@126.com
崔丽婷,李文彦,包 横,等.茶条槭SDH基因克隆及生物信息学分析[J].中南林业科技大学学报,2017, 37(4): 44-51.
[本文编校:文凤鸣]
