鸡肉中假单胞菌的近红外光谱快速识别
2017-08-31陈全胜王名星郭志明赵杰文
陈全胜 王名星 郭志明 范 冲 孙 浩 赵杰文
(江苏大学食品与生物工程学院, 镇江 212013)
鸡肉中假单胞菌的近红外光谱快速识别
陈全胜 王名星 郭志明 范 冲 孙 浩 赵杰文
(江苏大学食品与生物工程学院, 镇江 212013)
假单胞菌是鸡肉腐败最主要的致腐菌,为了快速识别鸡肉中的假单胞菌,首先从腐败鸡肉中分离并筛选出致腐菌,进一步利用聚合酶链反应技术对目标菌株进行生物学鉴别(分别为盖氏假单胞菌、嗜冷假单胞菌、莓实假单胞菌和荧光假单胞菌);配置鉴定的4种假单胞菌和等体积混合的4种假单胞菌菌液,采集菌液近红外透射光谱信息;然后运用标准正态变量变换对光谱进行预处理,利用联合区间偏最小二乘法筛选出特征波段;最后有比较地运用K最近邻法、最小二乘支持向量机和反向传播人工神经网络建立5种假单胞菌菌液的近红外光谱分类识别模型。其中反向传播人工神经网络模型预测效果最佳,其训练集和预测集的识别率分别为99.17%和95.00%。研究结果表明,近红外光谱结合反向传播人工神经网络可以快速识别鸡肉中的假单胞菌。
鸡肉; 近红外光谱; 假单胞菌; 主成分分析; 人工神经网络
引言
鸡肉在屠宰、包装、运输过程中极易受微生物的污染,使鸡肉腐败变质,破坏鸡肉的食用安全以及营养价值[1]。假单胞菌是导致鸡肉腐败的最主要致腐菌,常用来作为肉制品有氧和真空包装产品的加工卫生、保存质量和货架期预测的指示菌种[2-3]。因此研究监控鸡肉中致腐假单胞菌的组成及其动态变化情况,对鸡肉食用安全的保障以及营养价值的发挥具有实际意义。
微生物鉴别方法主要有传统微生物培养法[4]、分子生物学技术鉴定法[5]和免疫学分析技术[6]等。传统微生物培养法和免疫学技术鉴别法前处理繁琐、鉴定结果准确率低,且鉴别的菌株种类有限;分子生物学技术鉴定法虽可准确鉴别相应菌株,但操作繁琐、试剂昂贵、测定过程耗时长,更难以实现菌种的快速鉴别及判断[7]。
近红外光谱分析技术是一种通过光谱信息反映物质内部成分的物理测试技术,具有分析成本低、操作简单、速度快、样本无需前处理、样本无损伤等特点,已被广泛用于食品[8]、农产品[9]的定量分析和定性分类。在微生物检测方面的应用也越来越广泛[10-14]。本文利用聚合酶链反应(Polymerase chain reaction,PCR)方法对鸡肉中分离出的致腐目标菌株进行分子生物学鉴别后,采集菌体扩大培养得到的单菌种菌悬液以及混合菌悬液的近红外透射光谱信息,并结合联合区间偏最小二乘法筛选出高相关性的特征波段,然后比较运用K最近邻法(K-nearest neighbor, KNN)、最小二乘支持向量机 (Support vector machine,SVM)和反向传播人工神经网络 (Back propagation-artificial neural network,BP-ANN) 建立5种假单胞菌的近红外光谱分类识别模型,以期得到一种可对鸡肉中致腐假单胞菌快速分类识别的方法。
1 材料与方法
1.1 鸡肉中假单胞菌分离筛选
1.1.1 鸡肉样品准备
实验所用的鸡肉均购于江苏省镇江市欧尚超市肉制品专柜,均为新鲜鸡胸脯肉。购买后立即封装于灭菌样品袋中,运回实验室后置于4℃冰箱保存。
1.1.2 鸡肉致腐菌筛选
首先用已杀菌的剪刀从腐败冷却鸡肉表面取样10 g,剪碎后放入装有玻璃珠和90 mL无菌生理盐水的锥形瓶中,封口后置于摇床120 r/min振摇30 min制成菌悬液。将菌悬液逐级进行10倍的稀释,选取3个合适稀释度(10-8~10-6)的菌液,各取100 μL涂布于CFC琼脂选择培养基[15]的平板上适温培养,每个稀释度做3个重复。然后挑取菌落典型特征不同的单菌落,进行平板划线分离2次,得到纯化的单菌落,转接于胰酪胨大豆琼脂培养基斜面保存待用。
1.1.3 鸡肉致腐菌生物学鉴定
采用PCR技术对筛选的菌株进行16S rRNA扩增测定。首先取培养12 h的单菌落于30 μL无菌双蒸水中制成菌悬液,置于PCR仪中100℃煮沸10 min破壁制得模板DNA。采用通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1541R(5′-AAGGAGGTGATCCAGCCGCA-3′)扩增菌株的16S rRNA基因;然后以模板DNA 2 μL、2×PCR mix 12.5 μL、上游引物27F(10.4 nmol/L)1 μL、下游引物1541R (9.9 nmol/L)1 μL和无菌双蒸水 8.5 μL作为25 μL的PCR反应体系;设置PCR参数为95℃预变性5 min,95℃变性1 min、55℃退火1 min、72℃延伸90 s(重复上述3个步骤35个循环),延伸72℃、10 min,最后10℃冷却5 min;再用1×TAE配制1%的琼脂糖凝胶,取5 μL PCR扩增产物与定量的上样缓冲液混合后进行琼脂糖凝胶电泳,电压100 V,电泳时间30 min;同时用凝胶成像系统记录电泳图谱;最后将PCR扩增产物送至北京诺赛基因有限公司进行测序,登录美国国立生物技术信息中心 (http:∥blast.ncbi.nlm.nih.gov/blast),所得序列与数据库已知序列进行比对,获得相似性99%以上的菌株序列,使用MEGA 5.0软件构建Neighbor-Joining系统发育树[16]。
1.2 假单胞菌菌液近红外光谱采集
1.2.1 菌液样品准备
将保藏备用的4种假单胞菌单菌落挑取出来,分别溶于胰酪胨大豆肉汤培养基(Trypticase soy broth, TSB) 中30℃培养12 h,分别取100 μL均匀菌液40管5 mL TSB培养基中30℃培养12 h(达到细菌生长的稳定期,菌体浓度为107CFU/mL)得到160个样本;考虑到在实际鸡肉样本致腐菌检测过程中,不仅存在单一的一种假单胞菌,而通常是多种类别假单胞菌同时存在,实验分别将4种细菌等体积混合的菌液100 μL加入到40管5 mL TSB培养基中相同条件培养作为第5种假单胞菌;由此共制得200个样本。
1.2.2 菌液光谱采集和仪器参数设置
采用Antaris II型傅里叶变换近红外光谱仪(美国Thermo Fisher公司)进行实验。光谱采集波数范围4 000~10 000 cm-1,波数间隔为3.856 cm-1,含有1 557个变量。扫描次数16次,分辨率8 cm-1。实验选用透射模式,以仪器内置背景为参比,采用仪器标配的光程为5 mm、容量约1 mL的标准管为样品池。在实验过程中,保持室温25℃、相对湿度60%。每个菌液样本分装于4个标准管,平行采集光谱,4个光谱数据的均值作为该样本对应的原始光谱数据。每种菌液配置40个样本,经傅里叶变换近红外测定后,总共得到200个样本的光谱数据。数据处理采用Matlab R2009b(美国Mathworks公司)软件完成。
1.3 光谱数据预处理
原始光谱数据存在干扰信息,如各种仪器噪声、样品背景以及杂散信号等,这些干扰信息对建立稳定可靠的模型会有很大的影响;标准正态变量变换(Standardized normal variable transformation, SNV)、多元散射校正、标准分数校正、一阶导数以及二阶导数等方法可对原始光谱进行预处理,期望减弱甚至消除这些干扰信息。原始光谱有1 557个变量,其中存在与待测样品无关的物质信息,比如菌液的大量水分子和大量有机成分的含氢基团(如O—H、C—H、N—H等)在近红外光谱区产生的各级倍频和合频的吸收;这些非相关物质信息的光谱区间导致建模过程繁琐、计算量大,也会导致模型的稳定性和鲁棒性不好。采用联合偏最小二乘法(Synergy interval PLS,SiPLS)筛选变量,SiPLS的基本原则是将整个光谱划分成若干较小的等距区间,然后将局部模型精度较高的2~4个子区间联合起来预测样本品质指标,利用交互验证均方根误差(Root mean square error of cross validation, RMSECV)来评价联合区间的预测能力,RMSECV最小时为最佳的区间组合,根据结果提取出区间的全部变量,完成光谱变量的筛选[17],剔除那些噪声较大和非相关的光谱区域,筛选出高度相关的特定光谱区间和具有代表的有效变量;期望模型计算得到简化,建立预测能力强、稳健性好的识别模型。
2 结果与分析
2.1 PCR鉴定结果
凝胶成像系统拍摄记录的电泳图谱如图1所示,可见4种筛选得到的致腐菌均得到了1.5 kb左右的条带,于是将PCR扩增产物进行进一步的测序;待测菌株序列与NCBI中GenBank数据库已知序列的比对,利用同源性在99%以上的菌株序列与目标菌株构建Neighbor-Joining系统发育树,如图2所示。由系统发育树可以看出:P9与盖氏假单胞菌(PseudomonasgessardiiIHBB 9179)亲缘关系最近,P8与嗜冷假单胞菌(Pseudomonaspsychrophilastrain Den-03) 亲缘关系最近,S2与莓实假单胞菌(Pseudomonasfragistrain PF04) 亲缘关系最近,T3与荧光假单胞菌(Pseudomonasfluorescensstrain PF01)亲缘关系最近。
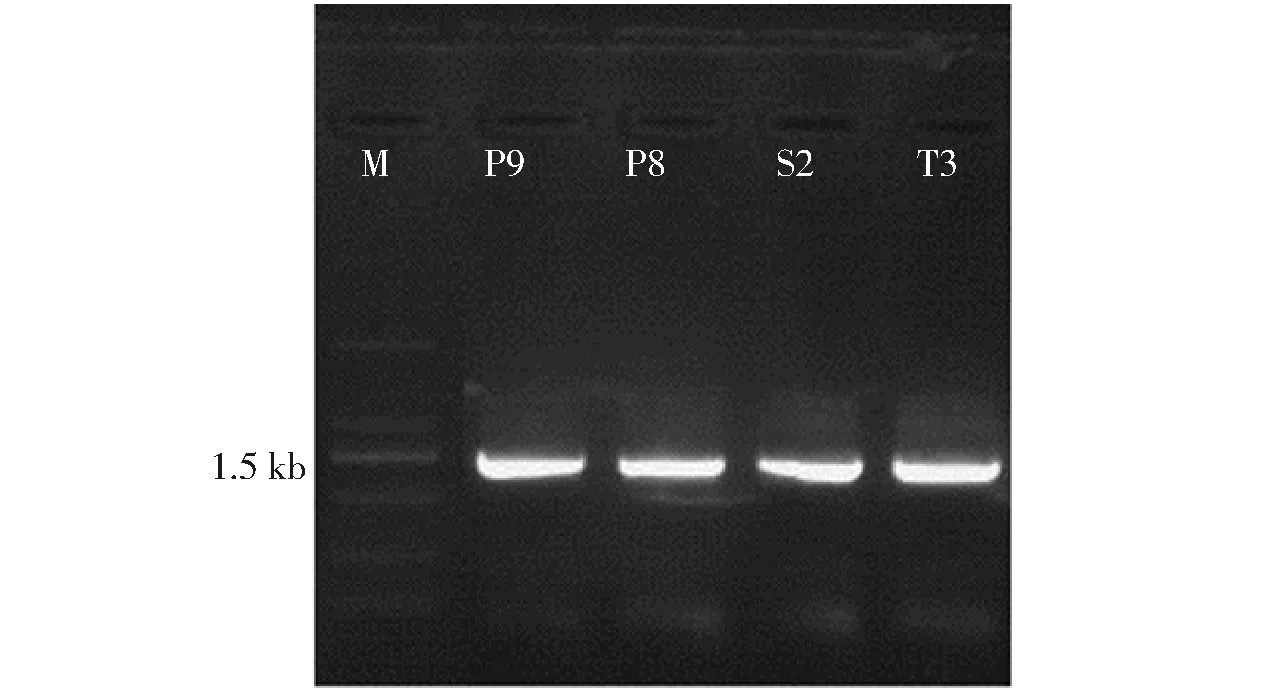
图1 4个菌株的16S rRNA PCR扩增产物电泳图谱Fig.1 Electropherogram of PCR amplification products of 16S rRNA

图2 基于16S rRNA序列同源性的4株致腐菌系统发育树Fig.2 Phylogenetic tree of four strains based on 16S rRNA gene sequences
2.2 特征波段筛选结果
2.2.1 光谱预处理
5种假单胞菌的原始光谱数据如图3a所示,可以看出假单胞菌光谱透射率具有相同的趋势,而且相同的波数下吸收度不同,表明通过近红外透射光谱信息能有效对菌株进行定性识别[18]。由于环境、仪器所带来的噪声以及菌液分子不断运动的影响,所采集的光谱数据掺杂了一些无关信息,若直接用原始数据建模会降低模型的预测效率,影响模型的准确性和稳定性。为了减少近红外光谱随机噪声的不良影响,使用多种预处理方法对原始光谱进行预处理。经反复比较,发现使用SNV的处理效果最佳。图3b为经过SNV预处理后的光谱,有效消除了光谱的背景和噪声干扰。

图3 5种假单胞菌的平均近红外光谱及SNV预处理后的近红外光谱Fig.3 Average original spectra and spectra processed by SNV of five Pseudomonas spp.
2.2.2 特征波段筛选
光谱数据变量筛选的结果如图4所示,表明将全光谱数据划分成16个子区间,主成分数为7时,联合[1 5 8 9]4个子区间,获得的模型最佳,RMSECV为0.19。[1 5 8 9]特征光谱区间所对应的波数区间为3 999.64~4 597.46 cm-1,6 406.37~7 004.19 cm-1,8 211.41~8 805.38 cm-1,8 809.24~9 403.20 cm-1,共622个变量。筛选的特征光谱区间中包含了假单胞菌细胞中核酸、蛋白质、脂肪和糖类等大分子物质含量、构型和构象等信息,可以很好地反映假单胞菌的特征性吸收,提高模型的稳定性和准确性。
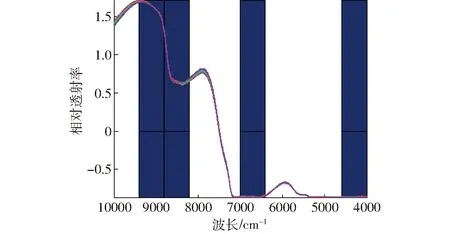
图4 联合区间偏最小二乘模型选择的最佳子区间[1 5 8 9]Fig.4 Optimal spectral regions [1 5 8 9] selected by SiPLS
2.3 识别模型
2.3.1 主成分聚类分析
主成分分析是在尽量不丢失原特征变量的信息前提下,通过正交变换将可能存在相关性的变量转换为一组线性不相关的变量(主成分),使主成分尽可能多地表征原变量的数据特征,实现数据降维的多元统计方法[19]。研究采用主成分分析对原始光谱数据中的1 557个变量进行数据降维压缩,去除光谱中的重叠信息以及与菌液内部化学成分不相关的信息。图5为5种菌液的三维主成分得分图。

图5 三维主成分得分图Fig.5 Three-dimensional projection of PCA results
图5表明,主成分分析聚类效果显著,盖氏假单胞菌、嗜冷假单胞菌、荧光假单胞菌都能很好地聚类分开;莓实假单胞菌分别与盖氏假单胞菌以及嗜冷假单胞菌有重叠,原因可能是这3种菌在液体生存条件下内部细胞核酸、蛋白质、脂肪较荧光假单胞菌更为类似;混合生长的假单胞菌菌液集合了所有的4种假单胞菌的化学物质和代谢成分,其中与盖氏假单胞菌的重叠最多,原因是盖氏假单胞菌的核酸、蛋白质、脂肪含量较其他更高;还有可能是前3个主成分不能完全代表原始光谱数据的信息,需要进一步利用模式识别算法对其进行分类。如图5所示,前3个主成分累计可信度已经达到99%,即前3个主成分基本能反映原始光谱数据的主要信息。
主成分因子数对模型的训练和预测都有一定的影响,研究依次选取前10个主成分对应的光谱数据为输入变量(几乎代表原始变量),建立分类识别模型。实验共采集200个样本的近红外光谱数据,采用随机分组的方式将全部数据分为120个训练集样本,80个预测集样本。
2.3.2 KNN识别模型
K最近邻法是一种典型的基于类比的算法,基本思想是给定一个样本,如果它与k个样本距离最近,则判别这个样本与k个样本为一类[20]。研究通过优化主成分数和k值,建立KNN识别模型,预测集识别率作为优化结果的评判标准。
如图6b所示,当主成分数为7,k为9时,KNN最佳预测集识别率为65.63%。当主成分数为8,参数k为7时,识别效果最优,训练集和预测集识别率分别为65.00%和63.75%。结果表明,KNN模型并不能很好地对5种假单胞菌进行分类识别。原因可能是5种假单胞菌的近红外光谱数据并不是线性关系,尝试用非线性的方法进行模型建立效果可能会更好。
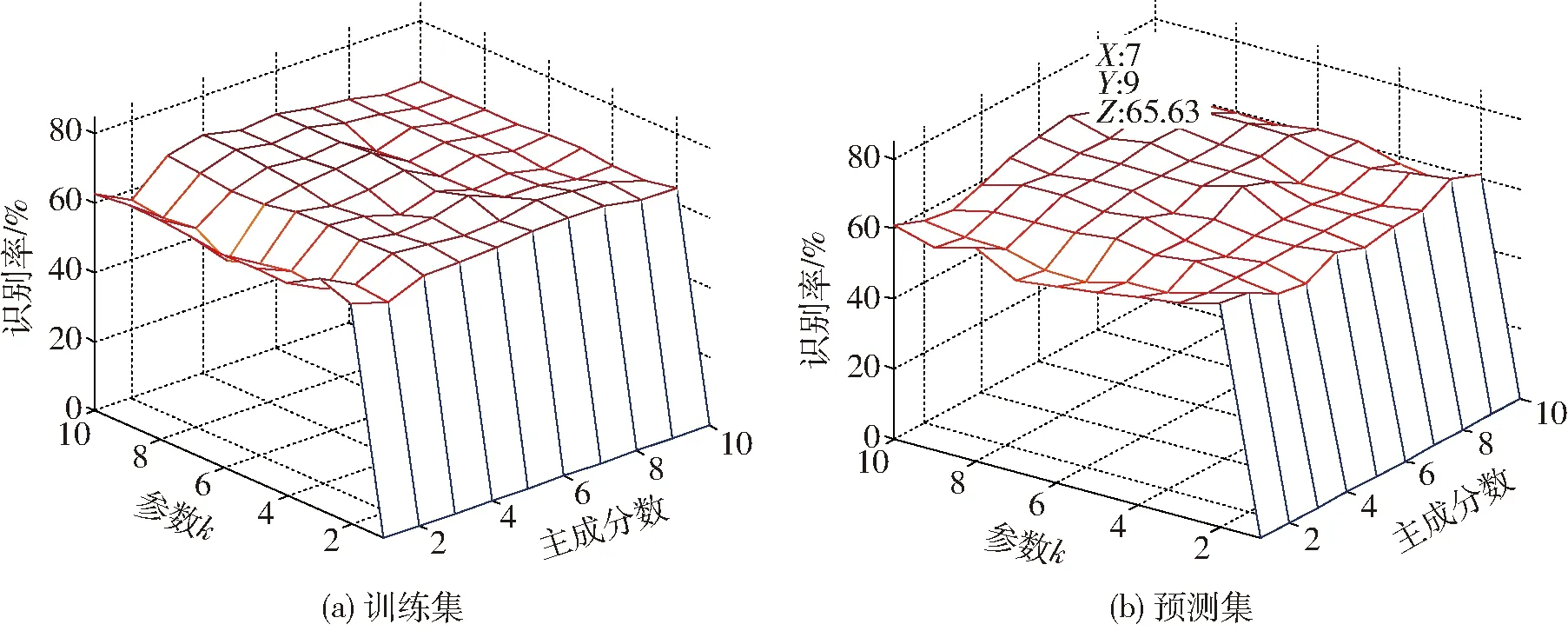
图6 KNN模式识别的训练集和预测集Fig.6 Calibration and prediction results for identification of five bacilli based on KNN
2.3.3 SVM识别模型
支持向量机是一个有监督功能的学习模型,基本原理是遵循结构风险最小化准则构造决策超平面,实现空间点的划分,从而进行分类识别,具有结构简单、适应性强、全局最优等特点,用于线性和非线性的多元建模[21]。SVM的分类精度主要取决于核函数与参数的选取,先确定惩罚系数C,然后采用网格搜索法对参数进行2次寻优计算,确定最优的参数(γ,σ2)组合(γ表示正则化参数,σ表示基于RBF核函数的参数)。图7为2次寻优计算过程,寻找的最终组合参数为(1.722 39,38.667 9)(图中以红色五角星标记),SVM训练集和预测集识别率为91.67%和86.25%。结果表明,基于SVM的5种假单胞菌近红外光谱分类识别模型效果显著。
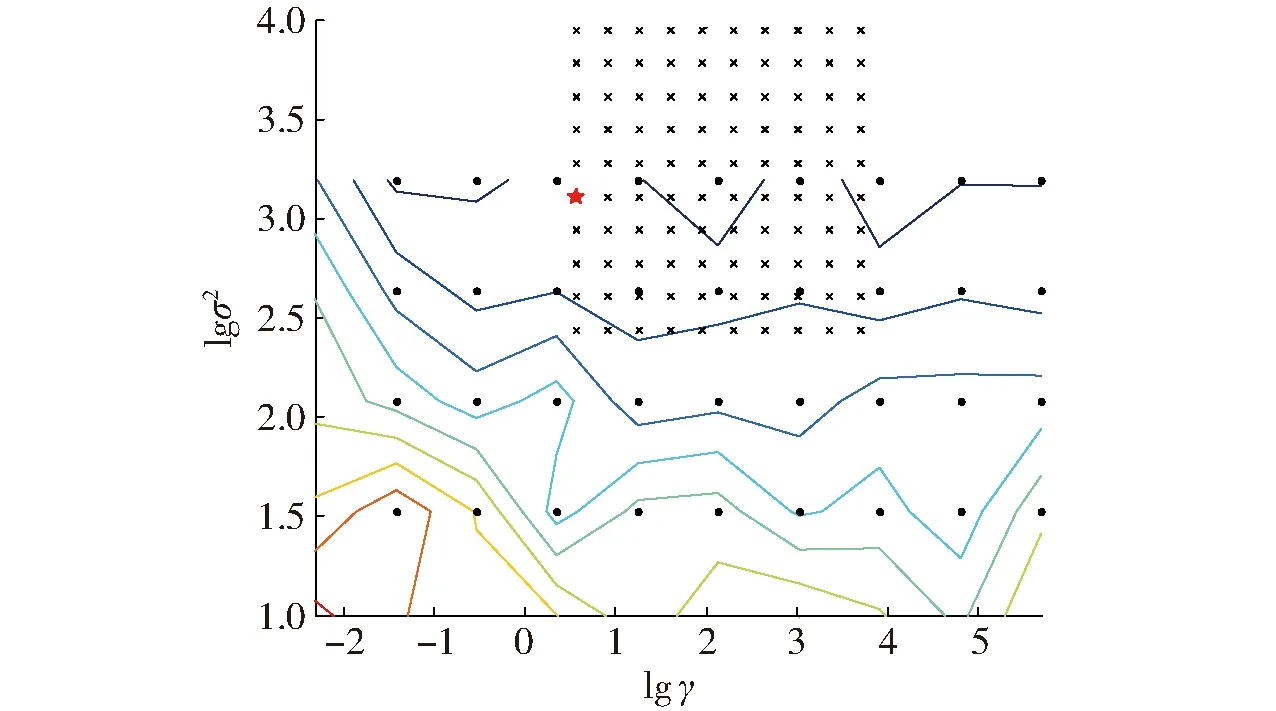
图7 网格搜索法寻找的最优参数组合(γ,σ2)Fig.7 Grid search method to find optimal parameters (γ,σ2)
2.3.4 BP-ANN识别模型
反向传播神经网络是一种多层前馈神经网络算法,基本原理是通过迭代优化网络的权值使得输入与输出之间的实际映射关系与所期望的映射关系一致,采用梯度下降算法调整优化各层权值,使目标函数最小化,具有很强的自学习性、自适应性、鲁棒性以及容错性等特点,是一种灵活的非线性建模算法[22]。依次选取前10个主成分对应的光谱数据为输入层,模型的输出层单元数设为1个(即菌株类别),传递函数为双曲正切函数(tanh),初始权重为0.95,训练过程中的学习因子和动量因子都为0.1,训练迭代 100次,结果如图8所示。当主成分为8时,BP-ANN的训练集和预测集的识别率最高,分别为99.17%和95.00%。

图8 基于BP-ANN的各主成分的识别率Fig.8 Identification rate of each principal component based on BP-ANN
2.3.5 3种模型比较分析
3种模式识别模型结果如表1所示,近红外光谱分析技术结合BP-ANN方法识别5种致腐假单胞菌的效果最好。KNN 算法是一种线性的惰性学习方法,该方法不事先建立分类模型,主要靠周围有限、邻近的样本,而不是靠判别类域的方法来确定所属类别,对于同种不同株的假单胞菌效果不明显;SVM和BP-ANN都是非线性监督机制的学习方法,能有效对同种不同株的假单胞菌进行识别,2种算法能够多维度地分析光谱信息特点,更适合对同种不同株的假单胞菌进行分类识别。因此,研究采用非线性的模式识别方法对5种假单胞菌进行分类识别,相较于SVM,BP-ANN利用误差反向传递迭代建模,能更有效根据光谱信息区分种内菌液的化学成分和代谢产物。使用BP-ANN算法对菌种识别效果最好,识别率可以达到95.00%。
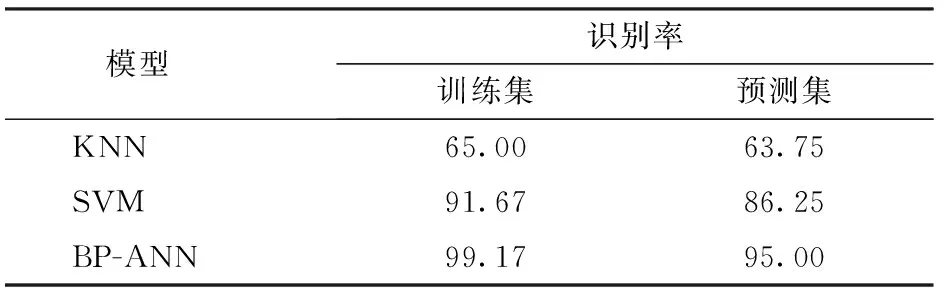
表1 基于KNN、SVM和BP-ANN模式的识别率Tab.1 Identification rates based on KNN,SVM and BP-ANN %
3 结论
利用近红外光谱分析技术结合模式识别方法,实现了鸡肉中5种致腐假单胞菌的快速识别:
(1)首先从腐败鸡肉中筛选分离出了相关致腐菌并利用PCR技术鉴定目标菌株分别为盖氏假单胞菌、嗜冷假单胞菌、莓实假单胞菌和荧光假单胞菌。
(2)运用SNV和SiPLS对光谱预处理和特征波段筛选,筛选的波段为3 999.64~4 597.46 cm-1,6 406.37~7 004.19 cm-1,8 211.41~8 805.38 cm-1和8 809.24~9 403.20 cm-1,这些波段与假单胞菌细胞中核酸、蛋白质、脂肪和糖类等大分子物质含量、构型和构象等信息极相关。
(3)有比较地运用KNN、SVM和BP-ANN建立了5种假单胞菌的分类识别模型。结果表明,主成分为8时的BP-ANN模型预测效果最佳,对应的训练集识别率为99.17%,预测集识别率为95.00%。
(4)研究表明近红外光谱分析技术可以快速、准确、无损地识别鸡肉中分离的4种致腐假单胞菌单菌种及其混合菌种菌液,将有效实现快速、准确地监控鸡肉中假单胞菌以及其他致腐微生物的群落组成及其动态变化,缩短检测时间,为鸡肉货架期的延长提供了有效的参考。
1 孙彦雨,周光宏,徐幸莲,等. 冰鲜鸡肉贮藏过程中微生物菌相变化分析[J]. 食品科学, 2011, 32(11): 146-151. SUN Y Y, ZHOU G H, XU X L, et al. Changes in microbial community composition of chilled chicken during storage[J]. Food Science, 2011, 32(11): 146-151. (in Chinese)
2 傅鹏,李平兰,周康,等. 冷却肉中假单胞菌温度预测模型的建立与验证[J]. 农业工程学报, 2008, 24(4): 229-234. FU P, LI P L, ZHOU K, et al. Development of models to predict the growth ofPseudomonasisolated from chilling pork[J]. Transactions of the CSAE, 2008, 24(4): 229-234. (in Chinese)
3 邱静,董庆利,程飞,等. 气调包装冷却猪肉中假单胞菌生长概率模型的构建[J]. 农业工程学报, 2012, 28(13): 257-262. QIU J, DONG Q L, CHENG F,et al. Establishment of growth probability model forPseudomonasspp. in chilled pork with modified atmosphere package[J]. Transactions of the CSAE, 2012, 28(13): 257-262. (in Chinese)
4 SHOBHARANI P, AGRAWAL R. Interception of quorum sensing signal molecule by furanone to enhance shelf life of fermented milk[J]. Food Control, 2010, 21(1): 61-69.
5 MARTY E, BUGHS J, EUGSTER-MEIER E,et al. Identification of staphylococci and dominant lactic acid bacteria in spontaneously fermented Swiss meat products using PCR-RFLP[J]. Food Microbiology, 2012, 29(2): 157-166.
6 WAGAR L E, CHAMPAGNE C P, BUCKLEY N D, et al. Immunomodulatory properties of fermented soy and dairy milks prepared with lactic acid bacteria[J]. Journal of Food Science, 2009, 74(8): 423-430.
7 CONG J, LIU X, LU H, et al. Analyses of the influencing factors of soil microbial functional gene diversity in tropical rainforest based on GeoChip 5.0[J]. Genomics Data, 2015, 5(1): 397-398.
8 GUO Z, HUANG W, PENG Y, et al. Color compensation and comparison of shortwave near infrared and long wave near infrared spectroscopy for determination of soluble solids content of ‘Fuji’ apple[J]. Postharvest Biology and Technology, 2016, 115: 81-90.
9 郝勇, 陈斌. 茶叶中低含量氨基酸近红外光谱定量分析模型研究[J/OL]. 农业机械学报, 2014, 45(6): 216-220. http:∥www.j-csam.org/jcsam/ch/reader/view_abstract.aspx?file_no=20140633&flag=1. DOI:10.6041/j.issn.1000-1298.2014.06.033. HAO Yong, CHEN Bin. Quantitative determination of low amino acid contents in tea by using near-infrared spectroscopy[J/OL]. Transactions of the Chinese Society for Agricultural Machinery, 2014, 45(6): 216-220. (in Chinese)
10 ZHANG Q, JIA F G, LIU C H, et al. Rapid detection of aflatoxin B1 in paddy rice as analytical quality assessment by near infrared spectroscopy[J]. International Journal of Agricultural & Biological Engineering, 2014, 7(4): 127-133.
11 石吉勇,胡雪桃,朱瑶迪,等. 基于近红外光谱技术的醋醅中常见杆菌快速识别[J/OL]. 农业机械学报, 2016, 47(2): 245-250. http:∥www.j-csam.org/jcsam/ch/reader/view_abstract.aspx?file_no=20160232&flag=1.DOI:10.6041/j.issn.1000-1298.2016.02.032. SHI J Y, HU X T, ZHU Y D, et al. Rapid identification of common bacilli in vinegar culture based on near-infrared spectroscopy [J/OL]. Transactions of the Chinese Society for Agricultural Machinery, 2016, 47(2): 245-250. (in Chinese)
12 刘建学, 张雅琪, 赵冰琳,等. 基于主成分分析的食源性致病菌的投影判别分析[J]. 中国食品学报, 2011, 11(8):159-162. LIU J X, ZHANG Y Q, ZHAO B L, et al. Projection discriminant analysis of food-borne pathogens based on principal component analysis[J]. Journal of Chinese Institute of Food Science and Technology, 2011, 11(8): 159-162. (in Chinese)
13 张雷蕾, 彭彦昆, 刘媛媛,等. 冷却肉微生物污染和肉色变化的Vis/NIR光谱无损检测[J/OL].农业机械学报, 2013, 44(增刊1):159-164. http:∥www.j-csam.org/jcsam/ch/reader/view_abstract.aspx?flag=1&file_no=2013s129&journal_id=jcsam.DOI:10.6041/j.issn.1000-1298.2013.S1.029. ZHANG L L, PENG Y K, LIU Y Y, et al. Non-invasive detection to TVC and color of chilled pork based on Vis/NIR spectroscopy[J/OL]. Transactions of the Chinese Society for Agricultural Machinery, 2013, 44(Supp.1): 159-164. (in Chinese)
14 SIRIPATRAWAN U, MAKINO Y, KAWAGOE Y, et al. Near infrared spectroscopy integrated with chemometrics for rapid detection of E. coli ATCC 25922 and E. coli K12[J]. Sensors and Actuators B: Chemical, 2010, 148(2): 366-370.
15 聂炎炎, 刘冬虹, 黄宇锋,等. 肉和肉制品中假单胞菌的检验和计数[J]. 现代食品科技, 2008, 24(2):184-187. NIE Y Y, LIU D H, HUANG Y F, et al. Examination and enumeration of pseudomonas in meat and meat products [J]. Modern Food Science and Technology, 2008, 24(2): 184-187. (in Chinese)
16 OLSSON C, AHRNE S, PETTERSSON B, et al. The bacterial flora of fresh and chill-stored pork: analysis by cloning and sequencing of 16S rRNA genes[J]. International Journal of Food Microbiology, 2003, 83(3): 245-252.
17 赵杰文,惠喆,黄林,等. 高光谱成像技术检测鸡肉中挥发性盐基氮含量[J]. 激光与光电子学进展, 2013, 50(7): 158-164. ZHAO J W, HUI Z, HUANG L, et al. Quantitative detection of TVB-N content in chicken meat with hyperspectral imaging technology[J]. Laser & Optoelectronics Progress, 2013, 50(7): 158-164. (in Chinese)
18 RODRIGUEZSAONA L E, KHAMBATY F M, FRY F S, et al. Rapid detection and identification of bacterial strains by Fourier transform near-infrared spectroscopy[J]. Journal of Agricultural & Food Chemistry, 2001, 49(2): 574-579.
19 王增丽,冯浩,余坤,等. 基于主成分分析的不同预处理麦秸改良土壤效果评价[J/OL]. 农业机械学报, 2016, 47(10): 150-155. http:∥www.j-csam.org/jcsam/ch/reader/view_abstract.aspx?file_no=20161020&flag=1. DOI:10.6041/j.issn.1000-1298.2016.10.020. WANG Zengli, FENG Hao, YU Kun, et al. Effect evaluation of different pretreated wheat straw utilizations on soil improvement based on principal component analysis[J/OL]. Transactions of the Chinese Society for Agricultural Machinery, 2016, 47(10): 150-155. (in Chinese)
20 DINIZ P H G D, BARBOSA M F, DE MELO M K D T, et al. Using UV-Vis spectroscopy for simultaneous geographical and varietal classification of tea infusions simulating a home-made tea cup[J]. Food Chemistry, 2016, 192: 374-379.
21 FENG Y Z, DOWNEY G, SUN D W,et al. Towards improvement in classification ofEscherichiacoli,Listeriainnocuaand their strains in isolated systems based on chemometric analysis of visible and near-infrared spectroscopic data[J]. Journal of Food Engineering, 2015, 149: 87-96.
22 BAO Y, REN J. Wetland landscape classification based on the bp neural network in daLinor lake area[J]. Procedia Environmental Sciences, 2011, 10(1): 2360-2366.
Rapid Identification ofPseudomonasspp. in Chicken by Near-infrared Spectroscopy
CHEN Quansheng WANG Mingxing GUO Zhiming FAN Chong SUN Hao ZHAO Jiewen
(SchoolofFoodandBiologicalEngineering,JiangsuUniversity,Zhenjiang212013,China)
Pseudomonasspp. is the main bacteria involved chicken degradation which ultimately affects the meat quality and the potential of posing health public health threats. The use of near infrared spectroscopy (NIRS) for rapid identification and monitoring of four strains ofPseudomonasspp. in degrading chicken was attempted. Initially, fourPseudomonasstrains namelyPseudomonasgessardii,Pseudomonaspsychrophila,PseudomonasfragiandPseudomonasfluorescenswere isolated from samples of degrading chicken and identified via polymerase chain reaction (PCR) technology. The different isolatedPseudomonasspp. were cultured in trypticase soy broth (TSB) and incubated at 30℃ for 12 h to growth. The four isolates ofPseudomonasspp. and their combined mixture in equal proportions were all prepared from the incubated inoculum by using 100 mL∶5 mL and each replicated 40 times. The preprocessed data outcomes of the 200 samples using standard normal variable transformation (SNV) exhibited superiority compared with other deployed data preprocessing algorithms such as multiplicative scatter correction (MSC), calibration standard score, first derivative (DB1) and second derivative (DB2). Synergy interval partial least squares (SiPLS) was employed to select relevant characteristics wavelengths such as 3 999.64~4 597.46 cm-1, 6 406.37~7 004.19 cm-1, 8 211.41~8 805.38 cm-1and 8 809.24~9 403.20 cm-1. Principal component analysis (PCA) was performed prior to the model development with loadings of 97.02% in PC1, 2.47% in PC2 and 0.27% in PC3 which indicated the possibility of developing models for the classification of the different samples ofPseudomonasspp. The recognition rates for KNN (65.00%, 63.75%), SVM (91.67%, 86.25%) and BP-ANN (99.17%, 95.00%) were obtained in the training and prediction sets. The model results obtained for SVM was sufficiently high and may be combined with NIRS system for the possiblePseudomonasspp. classification. However, the best result was obtained with BP-ANN built model. These high recognition rates implied near-infrared spectroscopy combined with BP-ANN can be deployed for the rapid detection of differentPseudomonasspp. strains in chicken for the purpose of safeguarding its deteriorating chicken quality.
chicken; near-infrared spectroscopy;Pseudomonasspp.; principal component analysis; artificial neural network
10.6041/j.issn.1000-1298.2017.08.039
2016-12-16
2017-01-09
国家自然科学基金面上项目(31371770)、江苏省高校自然科学研究面上项目(16KJB550002)和江苏大学高级人才基金项目(15JDG169)
陈全胜(1973—),男,教授,博士生导师,主要从事食品、农产品快速无损检测研究,E-mail: qschen@ujs.edu.cn
O657.33
A
1000-1298(2017)08-0328-07
