油用牡丹脂肪酸脱氢酶基因FAD3的克隆与表达分析
2017-06-15黄兴琳陆俊杏廖冰楠白辉扬管丽张涛
黄兴琳,陆俊杏,廖冰楠,白辉扬,管丽,张涛
(重庆师范大学生命科学学院,重庆 401331)
油用牡丹脂肪酸脱氢酶基因FAD3的克隆与表达分析
黄兴琳,陆俊杏,廖冰楠,白辉扬,管丽,张涛
(重庆师范大学生命科学学院,重庆 401331)
【目的】ω-3脂肪酸脱氢酶(ω-3 FAD)为植物脂肪酸生物合成途径中的关键酶,通过对油用牡丹‘凤丹’ω-3 FAD基因结构特征及其在不同组织中的表达模式进行分析,为研究FAD3在油用牡丹‘凤丹’脂肪酸形成过程中的调控提供理论基础。【方法】采用RACE和RT-PCR的方法克隆油用牡丹‘凤丹’ω-3 FAD基因,用Vector NTI Advance 11软件进行序列分析,BLAST进行同源性比较,并用MEGA7.0的邻接法(neighbor-joining,NJ)构建系统进化树,利用在线蛋白质分析工具ExPASy预测FAD3蛋白的基本参数和特性,利用Phyre2预测蛋白质二级和三级结构,并通过实时荧光定量PCR检测该基因在油用牡丹‘凤丹’不同发育期的种子以及不同组织中的特异性表达情况。【结果】获得油用牡丹‘凤丹’脂肪酸脱氢酶FAD3,命名为FAD3(GenBank登录号:KX906966)。序列分析表明该基因cDNA序列全长1 723 bp,其中,开放阅读框1 308 bp,编码435个氨基酸,3′端非编码区长287 bp,5′末端非编码区长99 bp,预测成熟蛋白分子量为49.9 kD,理论等电点(pI)为7.42,N端无信号肽,脂肪系数为83.08,不稳定指数35.67,总水平亲水性为-0.222。经FAD3蛋白的二级结构预测,FAD3以α-螺旋、随机卷曲为主,其次是延伸链、β-转角含量较少;多序列比对结果表明,油用牡丹FAD3氨基酸序列含有2个保守结构域,系统发育分析结果显示‘凤丹’与芍药处于同一分支,其亲缘关系最近。TMHMM和TargetP亚细胞定位分析得知,FAD3蛋白有3个跨膜区域,可能定位于内质网中发挥功能。组织特异性结果分析表明,FAD3在‘凤丹’的根、茎、叶、花瓣、雌蕊、雄蕊、种子中均有表达,其中,在叶中表达量最高,雌蕊次之,在雄蕊中表达量最低;不同时期种子中,10 d表达量最高,20 d次之,在60 d中表达量最低。【结论】成功从油用牡丹‘凤丹’中克隆获得FAD3全长cDNA序列,其在‘凤丹’不同组织中呈现出多种表达模式。
油用牡丹;脂肪酸脱氢酶;克隆;实时荧光定量PCR
0 引言
【研究意义】α-亚麻酸(ALA,C18:3)属ω-3系列不饱和脂肪酸,为人类必需脂肪酸,是DHA和EPA的合成前体,具有促进大脑发育、预防神经和心脑血管疾病、降低血脂和抑制衰老等重要作用,对人体正常发育和生长代谢起着至关重要的作用[1-3]。但人体由于缺乏ω-3脂肪酸脱氢酶,不能使亚油酸转化为ALA,所以必须依靠食物来获取[4-6]。目前,ALA的来源主要是深海鱼油,但中国深海鱼油资源较为贫乏,而日常食用菜籽油、豆油、葵花油等植物油虽然含有较为丰富的亚油酸,但ALA的含量却很低(5%左右)。在正常饮食情况下,人体往往不能足量摄入所需的ALA,致使中国人群膳食中普遍缺乏ALA,因此,挖掘和开发富含ALA的植物资源具有重要的应用价值[7-8]。牡丹(Paeonia suffruticosa Andr.)属芍药科(Paeoniaceae)芍药属(Paeonia)牡丹种(Sect.Moutan DC.),是一种闻名世界的多年生开花植物,在中国已有超过3 000年的栽培历史。油用牡丹是产籽量和出油率较高(≥22%)的牡丹统称,近年来,人们发现牡丹籽油不饱和脂肪酸含量达92%左右,特别是ALA含量高达40%以上,它除了具有独特的观赏和药用价值外,还是一种经济效益较高的新兴木本油料作物[9-11]。‘紫斑’(Paeonia rockii)和‘凤丹’(Paeonia ostii)牡丹是目前作为油料作物栽培的主要油用牡丹品种,它们不仅耐寒抗旱、适应力强,而且株型高大、结实率好,主要分布于安徽铜陵、山东菏泽、四川、甘肃、陕西秦岭、河南洛阳等地[12-14]。分离克隆控制ALA合成积累的关键酶基因,深入研究油用牡丹种子高效合成积累ALA的分子机理,可为油用牡丹种质资源的发掘和利用奠定基础。【前人研究进展】在植物脂肪酸合成代谢过程中,油脂合成关键酶基因的表达水平对合成脂肪酸的类型及数量起重要的作用。植物ALA由ω-3脂肪酸脱氢酶(FAD3、FAD7、FAD8)催化亚油酸而来,该酶是不饱和脂肪酸合成途径的一个限速酶,通过调节该酶的过量表达,可以使植物中ALA的含量增加。植物中一般存在 2种 ω-3 FAD,一种定位于内质网中(FAD3),另一种在质体中(FAD7、FAD8),植物种子中的ALA主要是通过ω-3 FAD3催化合成[15]。研究发现,在拟南芥种子中过表达ω-3 FAD3基因,可使ALA含量从19%增加到39.6%[16]。在大豆种子中的异位表达拟南芥ω-3 FAD3基因可使ALA含量获得提高[17-18]。将芝麻 FAD7质体定位信号序列替换为内质网定位信号,将其导入烟草后,转基因烟草种子中ALA含量达4.78%—6.77%[19]。将紫苏ω-3 FAD3基因导入酿酒酵母INVSc1中,发现工程菌可以将底物亚油酸转化为 ALA,其含量占总脂肪酸的 4%左右[20]。MURAKAMI等[21]使烟草中ω-3 FAD基因沉默,导致突变株的ALA含量比野生型植株明显减少,并能更好地适应高温环境。ZHOU等[22]发现ω-3 FAD基因的过量表达可显著提高白杨的抗冷性。以上研究结果表明,ω-3 FAD基因不但能够提高植物ALA的含量,而且还可改良植物对温度等逆境胁迫的适应性。近年来,人们陆续克隆了一些植物的微粒体或质体ω-3 FAD基因,并应用于基因工程研究,该方向已成为植物脂肪酸代谢积累调控和遗传改良研究的一个热点领域。【本研究切入点】油用牡丹作为目前发现的ALA含量最高的木本油料植物资源之一,是具有较好应用前景的油脂工程研究对象,但目前国内外有关油用牡丹的研究主要侧重于其栽培技术、成分分析、药理及相关产品开发等方面,而对油用牡丹的分子生物学研究相对较少,尤其是对ALA高效合成关键基因的生物学功能等决定其优异品质的重要环节缺乏相应的研究,目前国内外对于油用牡丹脂肪酸脱氢酶基因的分子作用机制等鲜有报道[10]。【拟解决的关键问题】本研究在对油用牡丹‘凤丹’FAD3基因进行克隆与生物信息学分析基础上,采用实时荧光定量PCR技术对不同组织中FAD3进行表达分析,为进一步开展FAD3在不饱和脂肪酸生物合成过程中的调控作用研究奠定基础。
1 材料与方法
1.1 材料
油用牡丹品种‘凤丹’(Paeonia ostii)种植于重庆师范大学牡丹试验基地。试验于2016年3月至2016 年6月在重庆师范大学油用牡丹种质资源创新与利用重点实验室完成。分别在开花时取材根、茎、叶、花瓣、雌蕊、雄蕊;授粉后以每10 d为间隔,分别取材10、20、40、60和80 d的牡丹种子,经液氮速冻后-80℃保存,备用。
工程菌Escherichia coli DH5α为细胞与遗传学实验室保存。Premix Taq酶、T4DNA连接酶、DL2000 Marker、T/A克隆载体pGEM-T、逆转录试剂盒购于TaKaRa公司。EASYspin植物RNA快速提取试剂盒(DNaseⅠ)购于北京博迈德公司。荧光定量试剂盒购于成都百乐公司。
1.2 引物设计和基因克隆
从NCBI核酸数据库中查询获得2条芍药EST序列信息,GenBank登录号分别为GBFN01008505.1和 KM575841.1。据此采用软件Primer Premier 5.0分别设计5′端和3′端RACE引物,上游引物PsFAD-GSP1.1:5′-GCTGCTGCCAATCCAAACACCACA-3′和PsFADGSP2.1:5′-CTCCATTAAAGCCACCACCGCCATC-3′;下游引物PsFAD-NGSP1.1:5′-CCTCACCAACCCCAT TAGTGGCATCT-3′和PsFAD-NGSP2.1:5′-GGGTTG GTGAGGAAGACGATTTTGAC-3′。按照试剂盒使用说明提取总RNA并反转录成cDNA,利用设计的引物,以上述cDNA为模板,进行RT-PCR扩增,将扩增产物纯化,回收,连接克隆载体pGEM-T,然后转化DH5α,进行抗性筛选,PCR鉴定后挑选单克隆送测序。引物及测序均由英潍捷基有限公司完成。
1.3 FAD3组织特异性表达分析
根据已克隆的油用牡丹‘凤丹’FAD3的cDNA序列分析结果,设计荧光定量 PCR引物,分别以根、茎、叶、花瓣、雌蕊、雄蕊、不同时期种子的cDNA为模板,以 PUF1639(Protein of unknown function 1639 gene)为内参基因[23],反应所用引物为PUF1639Forward:5′-AAACGAGTCGGTTGAAGA TGAG-3′,PUF1639Reverse:5′-TATGCGGTGGATTT CGGAG-3′。FAD3Forward:5′-TTGCTGAGATCCGAG CTGCCATTC-3′,FAD3Reverse:5′-GCAGCCCAATA GAATGGCCAAACAAC-3′。实时荧光定量PCR检测FAD3在不同器官中的表达水平。RT-PCR依照SYBR® Premix Ex TaqTM II(TaKaRa公司)使用说明在仪器CFX96 Real-Time PCR Detection System(Bio-Rad公司)上进行。每处理3个生物学重复,3个技术重复,结果用于平均数统计和方差分析。
1.4 FAD3生物信息学分析
用Vector NTI Advance 11软件进行序列比对、查找开放阅读框和翻译,ExPASy在线服务器分析FAD3蛋白的分子量、等电点pI、疏水性等。SignalP4.1 server分析信号肽,TMHMM Server v.2.0分析跨膜区等。利用 Phyre2在线分析蛋白质结构和结构域,并构建FAD3蛋白的三维结构。利用MEGA7.0软件采用邻接法对同源蛋白构建进化树。
2 结果
2.1 FAD3全长cDNA的克隆
分别通过RACE法,以油用牡丹叶片cDNA为模板进行PCR扩增、T/A克隆和测序分析,获得5′末端序列长度约700 bp(图1-A),获得3′末端序列长度约1 300 bp(图1-B)。经1%的琼脂糖凝胶电泳检测,结果与预测大小一致。测序获得两末端片段后通过NCBI上BLAST比对分析确定起始密码子AT的位置,即FAD3的5′端,3′端存在明显的poly(A)加尾现象。Vector NTI Advance 11软件分析后进行电子拼接,成功从油用牡丹中克隆到全长FAD3(GenBank登录号为 KX906966),发现该基因全长序列共1 723 bp,其中5′非翻译区99 bp,3′端非翻译区共287 bp,包含1 308 bp的完整开放阅读框。经BLASTn序列比对表明,克隆的FAD3与芍药(Paeonia lactiflora)FAD3 (KM575841.1)具有97%的相似性,远高于其他物种的基因。
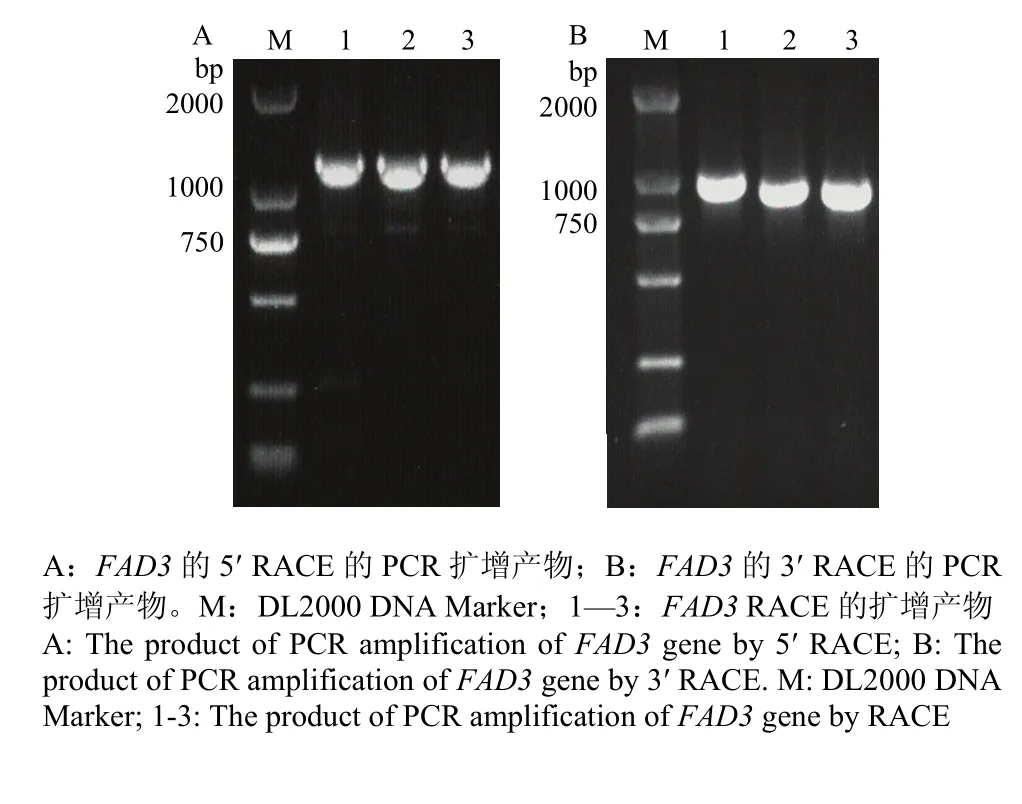
图1 FAD3的RACE PCR扩增产物Fig. 1 The product of PCR amplification of FAD3 gene by RACE
2.2 FAD3蛋白质的等电点、分子量及二级结构、三级结构的预测
FAD3编码435个氨基酸,预测分子量为49.9 kD,等电点为 7.42,带负电荷的酸性氨基酸(Asp+Glu)有 40个,带正电荷的碱性氨基酸(Arg+Lys)有 40个,该蛋白脂肪系数为83.08%,不稳定指数为35.67。分析FAD3蛋白的亲疏水性发现,总水平亲水性系数(GRAVY,grand average of hydropathicity)为-0.222,属亲水性蛋白。利用Phyre2软件对蛋白二级、三级结构预测结果表明,FAD3蛋白主要为α-螺旋(28.51%)、随机卷曲(37.93%)、延伸链(21.38%)和 β-转角(12.18%),推测该蛋白的结构功能域可能主要由α-螺旋构成(图2)。
2.3 FAD3蛋白质的跨膜域、信号肽、磷酸化位点、结构域预测
SignaIP server预测该蛋白无信号肽,TMHMM Server v.2.0分析具有3个跨膜域。亚细胞定位软件WoLF PSORT预测FAD3定位于内质网发挥功能。NetPhos2.0预测它具有18个潜在的磷酸化位点,主要包括10个丝氨酸(Serine)、4个苏氨酸(Threonine)和4个酪氨酸(Tyrosine)。由此推测FAD3蛋白活性可能与其磷酸化调控有一定联系。利用 NCBI在线分析工具CDD分析保守结构域(CD),结果表明,FAD3属于 Membrane-FADS-like蛋白超家族(图3)。
2.4 FAD3蛋白质与其他植物蛋白的进化分析
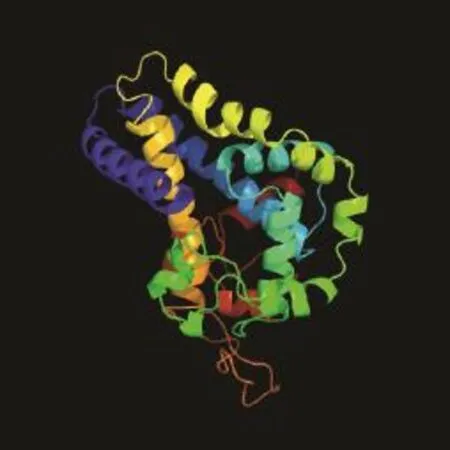
图2 Phyre2预测的FAD3三级结构Fig. 2 FAD3 tertiary structure predicted by Phyre2
从NCBI中查找到10种植物的FAD3蛋白序列,采用MEGA7.0邻接法与油用牡丹FAD3蛋白序列对比并建立无根系统进化树,结果表明,油用牡丹‘凤丹’与芍药(Paeonia lactiflora,AJA36814.1)、麻风树(Jatropha curcas,NP_001295746.1)、烟草(Nicotiana tabacum,NP_001313206.1)、雷蒙德氏棉(Gossypium raimondii,NP_001296282.1)、大豆(Glycine max,NP_001239777.1) 、 葡 萄 ( Vitis vinifera,XP_002277573.1)、拟南芥(Arabidopsis thaliana,AAL32546.1)、甘蓝(Brassica oleracea var. Oleracea,XP_013620518.1)、巨桉(Eucalyptus grandis,XP_010031203.1)、芝麻(Sesamum indicum,NP_001306619.1)等10个高等植物的FAD3蛋白序列比对显示,11个植物FAD3氨基酸序列被聚为两大类,‘凤丹’与芍药处于同一分支,其亲缘关系最近,其序列一致性为98.0%,其次与麻风树、烟草、雷蒙德氏棉蛋白亲缘关系较近,一致性分别为70.0%、73.0%和75.0%;与大豆、葡萄、拟南芥、甘蓝、巨桉、芝麻等物种的FAD3蛋白的亲缘关系较远(图4),推测‘凤丹’FAD3的功能可能与芍药属FAD3功能相似。
2.5 FAD3实时荧光定量分析
实时荧光定量分析表明(图 5),从其组织特异性表达来看,‘凤丹’FAD3在不同器官中均有表达,在叶中的表达量最高,与其他组织的差异达到极显著,是雌蕊中表达的3—4倍,且是根、茎的17—25倍,约是雄蕊的250倍。在不同发育时期的种子中,表达量随着种子成熟逐渐呈下调趋势,其中10 d的表达最高,约为60 d的25倍;20 d次之,40 d以后表达量明显降低(图6)。
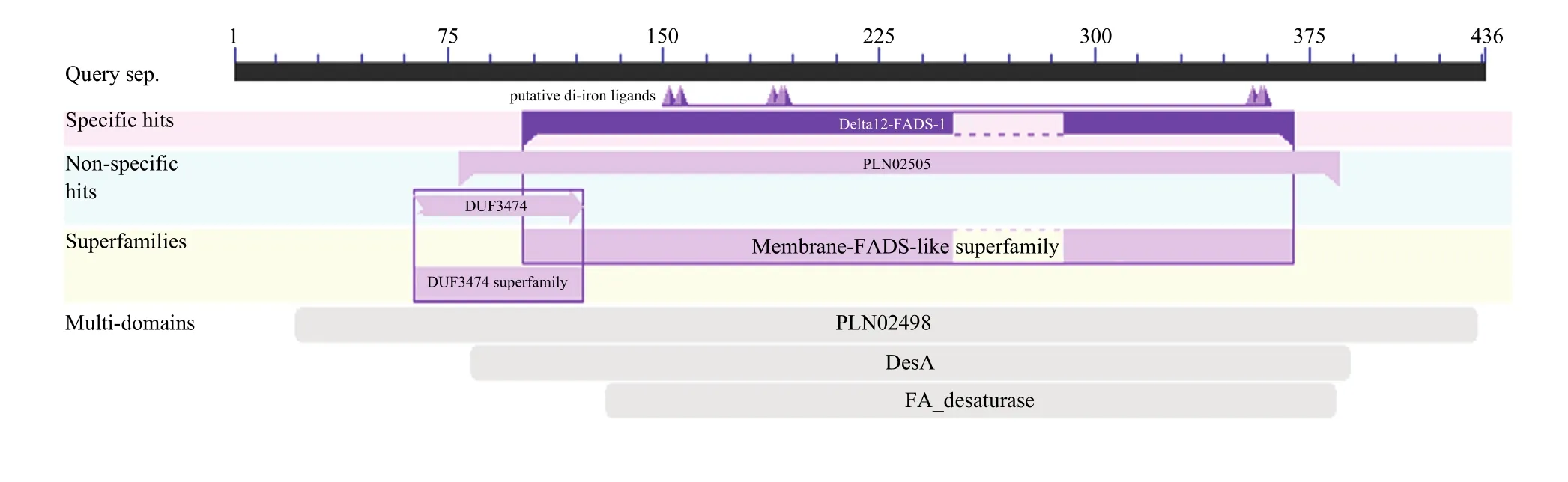
图3 FAD3保守结构域的预测结果Fig.3 Prediction of FAD3 conserved domain
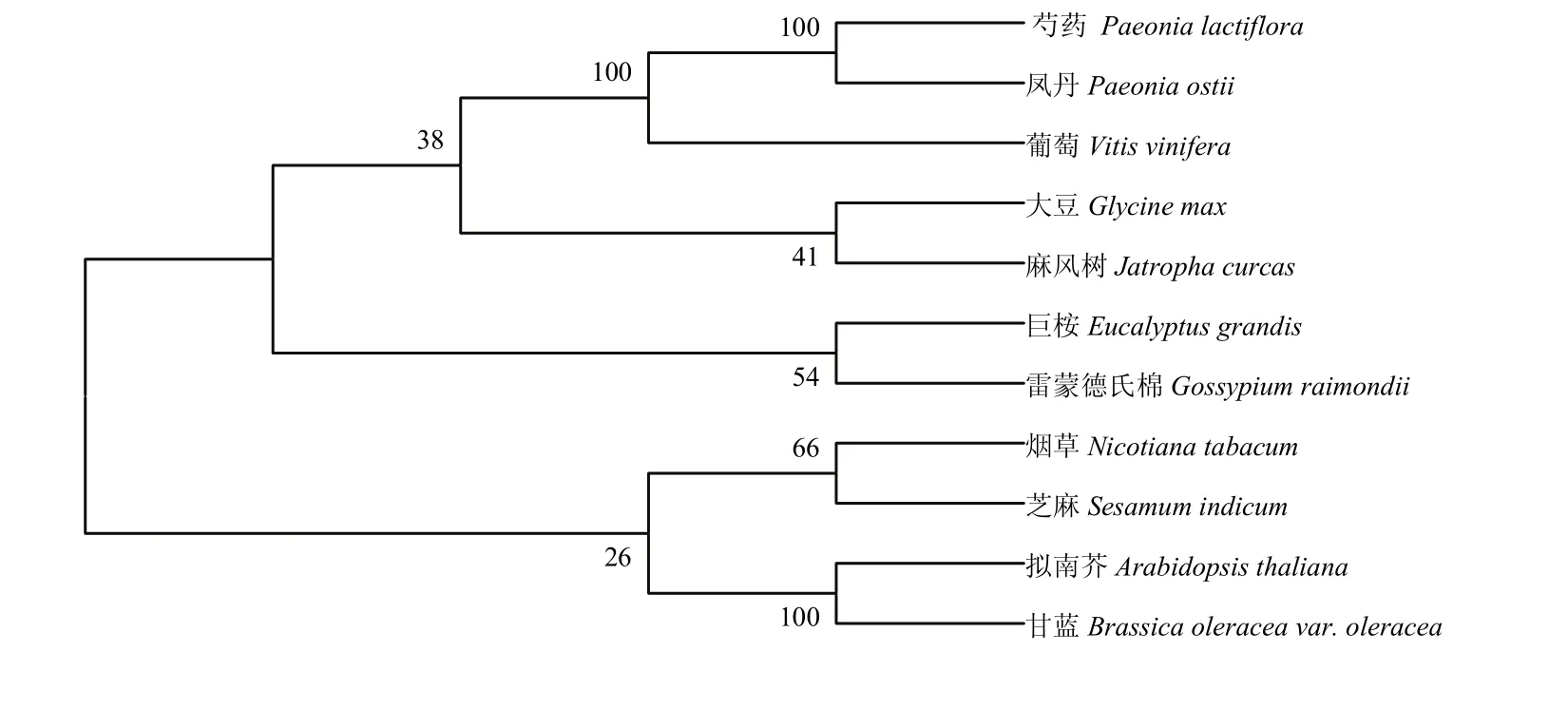
图4 ‘凤丹’FAD3与其他植物FAD3蛋白的系统进化树Fig. 4 Phylogenetic tree analysis of FAD3 proteins from Paeonia ostii and other plants
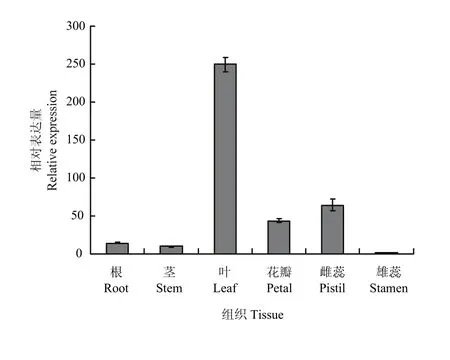
图5 FAD3在不同组织中的相对表达量Fig. 5 The relative expression of FAD3 gene in different tissues
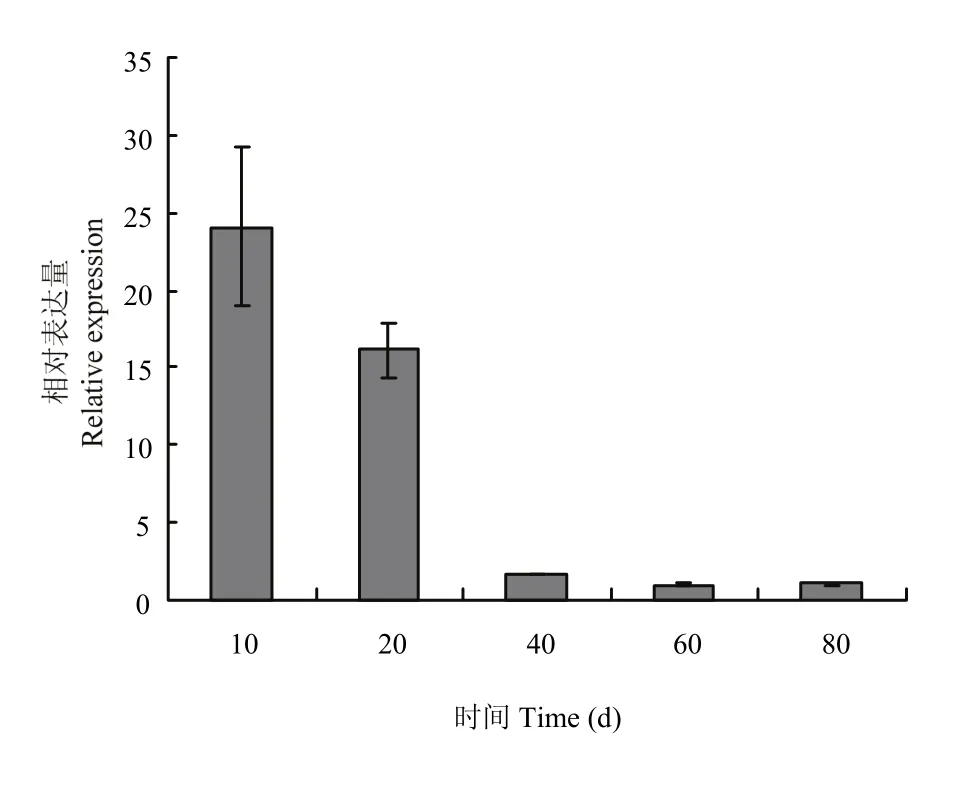
图6 FAD3在不同时期种子中的相对表达量Fig. 6 The relative expression of FAD3 gene in seeds of different growth stages
3 讨论
本研究以油用牡丹‘凤丹’为研究对象,在已知芍药FAD3部分片段的基础上进行RACE扩增,克隆获得‘凤丹’FAD3的全长cDNA序列,通过同源性比较发现,所得目的片段与同属植物芍药相似性极高。‘凤丹’FAD3编码的蛋白质具有植物ω-3脂肪酸脱氢酶的一般特征:在蛋白的二级结构中,主要以 α-螺旋和随机卷曲为主,推测其三维空间结构可能由 α-螺旋形成中心轴盘绕成螺旋状的结构,这表明跨膜蛋白FAD3的α-螺旋能够与生物膜表面发生相互作用,这种互作可能在蛋白跨膜过程中发挥重要作用[24-26]。序列比较分析表明,牡丹‘凤丹’FAD3同芍药、麻风树、烟草、雷蒙德氏棉等10种高等植物的FAD3氨基酸序列同源性高达70%以上,11种植物都具有 FAD3的 1个保守结构域,同属Membrane-FADS-like蛋白超家族,表明该结构域在进化过程中的稳定性较高,这可能与其在生长代谢中所具有的功能相关[27]。油用牡丹‘凤丹’FAD3氨基酸序列进化分析表明,‘凤丹’FAD3与芍药聚为一类,这说明同科同属相似性高的基因可能在脂肪酸合成中发挥相似的功能[28]。通过 TMHMM Server v.2.0跨膜结构预测和WoLF PSORT亚细胞定位分析得知,牡丹‘凤丹’FAD3蛋白具有3个跨膜结构域,可能定位于内质网中发挥功能,SignaIP server预测该蛋白无信号肽。
研究发现,红花ω-3脂肪酸脱氢酶基因CtFAD3在根、茎、叶、花、叶柄及种子中均能表达,且在花中的表达量最高,其次是叶片[29]。紫苏 FAD3在紫苏根、茎、叶、花、种子中均有表达,但表达量存在明显差异,其中在种子中的表达量最高,远高于其他组织器官,分别是花、叶、茎和根中表达量的8倍、9倍、43倍和58倍[30]。而本研究发现,FAD3在‘凤丹’的7个组织器官中均能检测到表达(图5和图6),但其特异性差异较显著,其中FAD3在牡丹的叶片中表达量最高,雄蕊中表达最低,前者约为雄蕊的250倍;叶片的相对表达约为10 d幼种的14倍;在不同发育时期的种子中都有表达,结实期10 d幼种的表达积累达到峰值,是成熟期种子(60和80 d)的25倍左右;20 d种子的表达是成熟期种子(60和80 d)的15倍左右。该结果与紫苏中有所不同,可能在牡丹种子不饱和脂肪酸形成过程中还存在其他基因的共同作用。结合LI等[10]研究来看,表明授粉后种子中不饱和脂肪酸脱氢酶迅速积累,此时正为亚油酸脱氢形成亚麻酸做准备;随着种子逐渐成熟,FAD3表达量呈下调趋势,而亚麻酸含量随之增加,在此过程中该基因的去饱和作用在峰值后慢慢变小,可能FAD3的活性与底物亚油酸含量的变化相关,说明本研究克隆得到的FAD3可能在牡丹不饱和脂肪酸合成途径中发挥重要作用[31]。
通过对FAD3在油用牡丹‘凤丹’中的组织特异性表达分析,表明FAD3在油用牡丹‘凤丹’不饱和脂肪酸的积累过程中发挥重要作用,为研究脂肪酸生物合成的分子机理及利用基因工程手段来调控α-亚麻酸含量提供了理论依据。
4 结论
成功从油用牡丹‘凤丹’中克隆获得FAD3全长cDNA序列,其在不同组织中呈现出多种表达模式。
[1] RUIZ-LOPEZ N, HASLAM R P, USHER S, NAPIER J A, SAYANOVA1 O. An alternative pathway for the effective production of the omega-3 long-chain polyunsaturates EPA and ETA in transgenic oil seeds. Plant Biotechnology Journal, 2015, 13(9): 1264-1275.
[2] KÖNIGS A, KILIAAN A J. Critical appraisal of omega-3 fatty acids in attention-deficit/hyperactivity disorder treatment. Neuropsychiatric Disease & Treatment, 2016, 12(1): 1869-1882.
[3] MAZZA M, POMPONI M, JANIRI L, BRIA P, MAZZA S. Omega-3 fatty acids and antioxidants in neurological and psychiatric diseases: An overview. Progress in Neuro-Psychopharmacology and Biological Psychiatry, 2007, 31(1): 12-26.
[4] SIMOPOULOS A P. n-3 fatty acids and human health: Defining strategies for public policy. Lipids, 2001, 36(1): S83-S89.
[5] HU F B, STAMPFER M J, MANSON J E, RIMM E B, WOLK A, COLDITZ G A, HENNEKENS C H, WILLETT W C. Dietary intake of alpha-linolenic acid and risk of fatal ischemic heart disease among women. American Journal of Clinical Nutrition, 1999, 69(5): 890-897.
[6] MCLENNAN P L, ABEYWARDENA M Y, CHARNOCK J S. Dietary fish oil prevents ventricular fibrillation following coronary artery occlusion and reperfusion. American Heart Journal, 1988, 116(3): 709-717.
[7] WHELAN J, BROUGHTON K S, KINSELLA J E. The comparative effects of dietary alpha-linolenic acid and fish oil on 4-and 5-series leukotriene formation in vivo. Lipids, 1991, 26(2): 119-126.
[8] PITTAWAY J K, CHUANG L T, AHUJA K D K, BECKETT J M,GLEW R H, BALL M J. Omega-3 dietary fatty acid status of healthy older adults in Tasmania, Australia: An observational study. Journal of Nutrition Health & Aging, 2015, 19(5): 505-510.
[9] BERUTO M, CURIR P. In vitro culture of tree peony through axillary budding. Protocols for Micropropagation of Woody Trees and Fruits, 2007: 477-497.
[10] LI S S, WANG L S, SHU Q Y, WU J, CHEN L G, SHAO S, YIN D D. Fatty acid composition of developing tree peony (Paeonia section Moutan DC.) seeds and transcriptome analysis during seed development. BMC Genomics, 2015, 16(1): 1-14.
[11] 陈景震, 李培旺, 张良波, 肖志红, 吴苏喜, 吴红, 赵志伟. 湖南油用牡丹籽油的理化性质及脂肪酸组分分析. 经济林研究, 2015, 4(33): 119-122. CHEN J Z, LI P W, ZHANG L B, XIAO Z H, WU S X, WU H, ZHAO Z W. Analysis of physicochemical properties and fatty acid composition of peony seed oil in Hunan. Nonwood Forest Research, 2015, 4(33): 119-122. (in Chinese)
[12] 张涛, 高天姝, 白瑞英, 黄兴琳, 郭楠, 陆俊杏, 罗安才. 油用牡丹利用与研究进展. 重庆师范大学学报(自然科学版), 2015, 2(32): 143-149. ZHANG T, GAO T S, BAI R Y, HUANG X L, GUO N, LU J X, LUO A C. Utilization and research progress of oil tree peony. Journal of Chongqing Normal University (Natural Science), 2015, 2(32): 143-149. (in Chinese)
[13] 林萍, 姚小华, 曹永庆, 薛亮, 王开良. 油用牡丹‘凤丹’果实性状及其脂肪酸组分的变异分析. 经济林研究, 2015, 1(33): 67-72. LIN P, YAO X H, CAO Y Q, XUE L, WANG K L. Variation analysis of fruit characteristics and fatty acid components in Paeonia ostii ‘Phoenix White’. Nonwood Forest Research, 2015, 1(33): 67-72. (in Chinese)
[14] 周志钦, 潘开玉, 洪德元. 牡丹组野生种间亲缘关系和栽培牡丹起源研究进展. 园艺学报, 2003, 30(6): 751-757. ZHOU Z Q, PAN K Y, HONG D Y. Advance in studies on relationships among wild tree peony species and the origin of cultivated tree peonies, Acta Horticulturae Sinica, 2003, 30(6): 751-757. (in Chinese)
[15] VRINTEN P, HU Z, MUNCHINSKY M A, ROWLAND G, QIU X. Two FAD3 desaturase genes control the level of linolenic acid in flax seed. Plant Physiology, 2005, 139(1): 79-87.
[16] PUTTICK D, DAUK M, LOZINSKY S, SMITH M A. Overexpression of a FAD3 desaturase increases synthesis of a polymethyleneinterrupted dienoic fatty acid in seeds of Arabidopsis thaliana L.. Lipids, 2009, 44(8): 753-757.
[17] DAMUDE H G, ZHANG H, FARRALL L, RIPP K G, TOMB J F, HOLLERBACH D, YADAV N S. Identification of bifunctional Δ12/ω-3 fatty acid desaturases for improving the ratio of ω-3 to ω-6 fatty acid in microbes and plants. Proceedings of the National Academy of Sciences of the USA, 2006, 103(25): 9446-9451.
[18] ECKERT H, LAVALLEE B, SCHWEIGER B J, KINNEY A J, CAHOON E B, CLEMENTE T. Co-expression of the borage Δ6 desaturase and the Arabidopsis Δ15 desaturase results in high accumulation of stearidonic acid in the seeds of transgenic soybean. Planta, 2006, 224(5): 1050-1057.
[19] BHUNIA R K, ANIRBAN C, RANJEET K, MRINAL K M, SOUMITRA K S. Enhancement of a-linolenic acid content in transgenic tobacco seeds by targeting a plastidial x-3 fatty acid desaturase (fad7) gene of Sesamum indicum to ER. Plant Cell Report, 2016, 35(1): 213-226.
[20] LEE K R, LEE Y, KIM E H, LEE S B, ROh K H, KIM J B, KANG H C, KIM H U. Functional identification of oleate 12-desaturase and ω-3 fatty acid desaturase genes from Perilla frutescens var. frutescens. Plant Cell Report, 2016, 12(35): 2523-2537.
[21] MURAKAMI Y, TSUYANA M, KOBAYASHI Y. Trienoic fatty acids and plant tolerance of high temperature. Science, 2000, 287: 476-479. [22] ZHOU Z, WANG M J, HU J J, LU M Z, WANG J H. Improve freezing tolerance in transgenic poplar by overexpressing a ω-3 fatty acid desaturase gene. Molecular Breeding, 2010, 25(4): 571-579.
[23] 刘洪峰, 高乐旋, 胡永红. 牡丹不同发育阶段种子和花瓣组织实时荧光定量 PCR中内参基因的挖掘与筛选. 农业生物技术学报, 2015, 23(12): 1639-1648. LIU H F, GAO L X, HU Y H. Reference genes discovery and selection for quantitative real-time PCR in tree peony seed and petal tissue of different development stages. Journal of Agricultural Biotechnology, 2015, 23(12): 1639-1648. (in Chinese)
[24] 宋淑香, 郭先锋, 马燕, 李俊杰, 韩璐璐. 凤丹(Paeonia ostii)脂肪酸去饱和酶基因 PoFAD2的克隆及表达分析. 园艺学报, 2016, 43(2): 347-355. SONG S X, GUO X F, MA Y, LI J J, HAN L L. Isolation and expression analysis of PoFAD2 gene in Paeonia ostii. Acta Horticulturae Sinica, 2016, 43(2): 347-355. (in Chinese)
[25] 王俊娟, 穆敏, 王帅, 陆许可, 陈修贵, 王德龙, 樊伟丽, 阴祖军,郭丽雪, 叶武威, 喻树迅. 棉花脱水素 GhDHN1的克隆及其表达.中国农业科学, 2016, 49(15): 2867-2878. WANG J J, MU M, WANG S, LU X K, CHEN X G, WANG D L, FAN W L, YIN Z J, GUO L X, YE W W, YU S X. Molecular clone and expression of GhDHN1 gene in cotton (Gossypium hirsutumL.). Scientia Agricultura Sinica, 2016, 49(15): 2867-2878. (in Chinese)
[26] 曹新文, 王秀珍, 李永梅, 赵李婧, 马海霞, 闫洁. 橡胶草法尼基焦磷酸合酶基因的克隆与功能分析. 中国农业科学, 2016, 49(6): 1034-1046. CAO X W, WANG X Z, LI Y M, ZHAO L J, MA H X, YAN J. Molecular cloning and functional analysis of farnesyl pyrophosphate synthase genes from Taraxacum kok-saghyz Rodin. Scientia Agricultura Sinica, 2016, 49(6): 1034-1046. (in Chinese)
[27] 王慧飞, 孙艳香, 冯雪, 张一名, 杨江涛, 马梅芳, 陈光. 棉花精氨琥珀酸合成酶基因 GhASS1的克隆及表达分析. 中国农业科学, 2016, 49(7): 24-35. WANG H F, SUN Y X, FENG X, ZHANG Y M, YANG J T, MA M F, CHEN G. Cloning and expression analysis of argininosuccinate synthetase gene GhASS1 from Gossypium hirsutum. Scientia Agricultura Sinica, 2016, 49(7): 24-35. (in Chinese)
[28] 梁文裕, 杨佳, 吴诗杰, 焦广飞, 王淑萍, 徐婷婷. 发菜耐旱相关蛋白NXL-01的基因克隆与表达分析. 西北植物学报, 2015, 35(1): 44-49. LIANG W Y, YANG J, WU S J, JIAO G F, WANG S P, XU T T. Clone and expression of HypOthetical protein NXL-01 related to droughttolerant in Nostoc flagelliforme. Acta Botanica Boreali- Occidentalia Sinica, 2015, 35(1): 44-49. (in Chinese)
[29] 官玲亮. 红花(Carthamus tinctor ius L.)不同组织多不饱和脂肪酸积累模式及调控机制[D]. 成都: 四川农业大学, 2011. GUAN L L. Accumulation pattern and regulatory mechanisms of fatty acid in different safflower (Carthamus tinctorius L.) tissues[D]. Chengdu: Sichuan Agricultural University. 2011. (in Chinese)
[30] 白瑞英, 黄兴琳, 陆俊杏, 袁中厚, 高天姝, 郭楠, 张涛. 紫苏 ω-3脂肪酸脱氢酶基因FAD3的克隆与表达分析. 分子植物育种, 2015, 13(12): 2728-2735. BAI R Y, HUANG X L, LU J X, YUAN Z H, GAO T S, GUO N, ZHANG T. Cloning and expression analysis of ω-3 fatty acid desaturase gene FAD3 from Perilla frutescens. Molecular Plant Breeding, 2015, 13(12): 2728-2735. (in Chinese)
[31] MARTÍNEZ-RIVAS J M, SPERLING P, LÜHS W, SPERLING P, LÜHS W, HEINZ E. Spatial and temporal regulation of three different microsomal oleate desaturase genes (FAD2) from normal-type and high-oleic varieties of sunflower (Helianthus annuus L.). Molecular Breeding, 2001, 8(2): 159-168.
(责任编辑 李莉)
Cloning and Expression Analysis of Fatty Acid Desaturase Gene FAD3 from Oil Peony
HUANG XingLin, LU JunXing, LIAO BingNan, BAI HuiYang, GUAN Li, ZHANG Tao
(College of Life Sciences, Chongqing Normal University, Chongqing 401331)
【Objective】 The Omega -3 fatty acid desaturase (ω-3 FAD) is a key enzyme in the fatty acid biosynthesis pathway of plant. Through analysis of omega-3 fatty acid desaturase gene structure and its expression in different tissues from Paeonia ostii, this study will lay a foundation for the research of regulating effects of FAD3 gene on fatty acid biosynthesis and provide a theoretical basis for the formation in the process of regulation. 【Method】 The FAD gene from Paeonia ostii (FAD3) was cloned using RT-PCR and RACE-PCR strategies. Nucleotide sequence was analyzed using Vector NTI Advance 11 software. Homology was analyzed using BLAST. A phylogenetic tree was constructed using neighbor-joining of MEGA 7.0. The secondary structure and three-dimensional model of FAD3 were predicted using ExPASy and Phyre2, respectively. Expression profiles of FAD3 at different developmental stages and in different tissues of the P. ostii were assayed using real-time quantitative PCR. 【Result】 The FAD genein P. ostii was cloned and named as FAD3 (GenBank accession number: KX906966). The full length of FAD3 cDNA is 1 723 bp, contains a 1 308 bp open reading frame (ORF) encoding a putative protein of 435 amino acids with a molecular mass of 49.9 kD and an isoelectric point (pI) of 7.42. The 3′-untranslated region of 287 bp and 5′-untranslated region of 99 bp were obtained from the FAD3 gene of P. ostii. N end without signal peptide, fat coefficient is 83.08, instability index 35.67, the grand average of hydrophobicity value of -0.222. By FAD3 protein secondary structure prediction, FAD3 mainly in the alpha helix and random coil, followed by less extended strand, beta turn content; multiple sequence alignment results showed that it contains two conserved domains of FAD3 gene. Phylogenetic tree analysis showed that the P. ostii has the closest evolutionary relationship with P. lactiflora. Subcellular localization analysis of TMHMM and Target P indicated that it might be targeted to the endoplasmic reticulum with three transmembrane regions. FAD3 gene was expressed in the root, stem, leaf, petal, pistil, stamen and seed of P. ostii by the tissue-specificity expression. The expressive content of FAD3 gene in different tissues of P. ostii was different: The highest was leaf, the next was pistil, and the lowest level was in stamen; In different periods of seeds, the highest expression was seeds of 10d, the next was 20d, in 60d expression was the lowest. 【Conclusion】 The full length cDNA sequence of FAD3 gene was successfully cloned from P. ostii, It shows a variety of expression patterns in different tissues, which laid a foundation for the further study on the function and expression regulation mechanism of FAD3 gene in the process of unsaturated fatty acid biosynthesis.
Paeonia suffruticosa Andr.; fatty acid desaturase; cloning; real-time PCR
2016-10-27;接受日期:2017-01-24
国家自然科学基金(31171588)、重庆市林业重点科技攻关项目(渝林科研2015-3)、重庆市社会民生科技创新专项(cstc2016shmszx80051)、重庆师范大学基金(14XLB015)
联系方式:黄兴琳,Tel:18716439877;E-mail:huangxlcf@163.com。通信作者张涛,Tel:15902359879;E-mail:zht2188@126.com
