42个花生品种的SSR标记指纹图谱构建
2017-06-05李双铃石延茂
尹 亮,李双铃,任 艳,石延茂,袁 美
(山东省花生研究所,山东 青岛 266100)
42个花生品种的SSR标记指纹图谱构建
尹 亮,李双铃,任 艳,石延茂,袁 美*
(山东省花生研究所,山东 青岛 266100)
以不同地区的42个花生品种为材料,利用23对SSR引物进行分析。23对SSR引物在42个花生品种中共检测到92个等位变异,平均每对引物4个等位基因,平均PIC值为0.49。进一步分析表明,8对引物就可以将42个花生品种完全区分开,8对引物共产生40个等位变异,利用这40个等位变异构建了42个花生品种的DNA指纹图谱。聚类分析结果显示,在相似系数0.74处,可将42个供试品种分为4个类群。本研究结果为花生品种保护和育种实践提供了理论依据。
花生;SSR;DNA指纹图谱;聚类分析
花生(ArachishypogaeaL.)是我国重要的油料作物和经济作物。近年来,随着大量花生新品种的推广与应用,以及在育种过程中高频率使用少数骨干亲本,造成花生品种遗传基础趋于狭窄,遗传多样性降低,使以形态性状鉴定为主的传统品种鉴定方法已经很难满足现在的生产需要。以分子标记为基础的DNA指纹图谱技术的出现为品种鉴定提供了新的快捷途径。目前,用于DNA指纹图谱构建的分子标记包括RFLP、RAPD、AFLP、SSR、ISSR和STS等[1]。与传统的形态学鉴定相比,DNA指纹图谱技术具有简单、快速、易于自动化等优点,已在水稻[2]、玉米[3]、大豆[4]、棉花[5]、高粱[6]等农作物品种鉴定及遗传分析中得到广泛使用。
在花生品种鉴定研究方面,已有多家单位构建了特定类型或部分区域的花生主栽品种的DNA指纹图谱。翁跃进等[7]利用AFLP技术对从国际半干旱所引进的9份花生抗旱品种绘制指纹图谱;李双铃等[8]利用MseI和EcoRI酶切和9对引物组合构建了10个山东花生主栽品种的AFLP指纹图谱。近年来,随着花生SSR标记引物的不断开发[9-13],其以数量丰富、共显性遗传、信息含量高、结果重现性好、检测手段简便易行等优点,现已成为构建花生品种DNA指纹图谱的主要分子标记。国内相关研究报道,已利用SSR分子标记分别构建了20个南方花生品种[14]、40个珍珠豆型花生品种[15]、山东审定的46个花生品种[16]以及河南审定的90个花生品种[17]的指纹图谱。此外,任小平等[18]利用SSR荧光引物构建了100份花生的指纹图谱。本研究利用SSR标记构建了不同地区的42个花生品种的DNA指纹图谱,并对SSR标记多态性进行分析,为品种保护和育种实践提供理论依据。
1 材料与方法
1.1 试验材料
采用来自山东、四川、河南、河北、广东、福建等省的42个品种为材料(表1)。所有花生材料均种植于山东省花生研究所莱西试验农场。
1.2 DNA提取
选取花生幼嫩健康叶片,使用天根生化科技(北京)有限公司的植物基因组DNA提取试剂盒提取花生叶片基因组DNA。利用1%琼脂糖凝胶电泳和紫外分光光度计检测DNA纯度及浓度,并将DNA稀释为30ng/μL。
1.3 SSR标记分析
选取经本研究组初筛具有多态性的23对SSR引物(表2)对42份花生品种进行检测。SSR引物序列均来源于Peanut Marker Database网站(http://marker.kazusa.or.jp/peanut/#),引物均由北京六合华大基因科技有限公司合成。PCR反应使用MDBIO(青岛)的Taq DNA聚合酶,反应体系参照说明书。PCR反应条件为:95℃下变性3min;95℃下30s,55℃下30s,72℃下30s,30个循环;72℃下延伸3min。PCR产物采用变性聚丙烯酰胺凝胶电泳检测,方法参考郑永胜等的方法[19]。
1.4 数据分析
分别以0和1记载各品种SSR等位变异,在相同迁移率位置上,有带记为1、无带记为0,位点数据缺失记为9,组成原始矩阵。利用SSR数据处理宏程序DataTrans 1.0对数据进行格式转换,并使用PowerMarker V3.25对SSR标记进行分析,计算出每个SSR标记的主基因频率、基因型数、等位基因数、基因多样性指数、杂合度以及PIC值。使用NTSYS-pc 2.10e软件的SAHN程序和UPGMA方法对供试材料进行聚类分析,生成聚类图并计算出各品种间的遗传相似系数。

表1 试验材料及来源

表2 引物及序列
2 结果与分析
2.1 SSR标记多态性分析
对23对SSR引物的电泳图谱进行分析(图1),在42个花生品种中共检测到92个等位变异,其中每个标记等位基因数3~8个,平均每个标记4个等位基因,其中AHGS1446和TC23H10分别检测到8个和7个等位变异;引物PIC值为0.27~0.76,平均0.49,PIC值超过0.5的引物有10对,其中AHGS1446、TC23H10、GNB827的PIC值超过0.7,GNB613和Seq11G07的PIC值最低,为0.27;引物基因多样性指数为0.32~0.79(表3);主基因频率为0.29~0.83;IPAHM455、PM183、Seq2G03、Seq3A08、Seq16F01、Seq18C05在1个品种中存在杂合位点,GNB317、GNB613、Seq19B12在2个品种中存在杂合位点。综合分析比较各参数,PIC值变化趋势与基因多样性指数变化趋势一致,PIC值越高则该引物对于不同品种的鉴别能力越强。
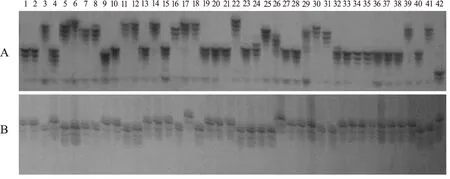
图1 引物AHGS1446和AHGS2535在42个花生品种中的扩增结果Fig.1 DNA fragments amplified by SSR primers AHGS1446 and AHGS2535 among 42 peanut varieties注:A:AHGS1446;B:AHGS2535;1~42:对应供试材料编号,编号与表1相同。Note: A: AHGS1446; B: AHGS2535; 1~42: The code of varieties corresponding with those in Table 1.

表3 SSR引物扩增等位基因数及多态性信息
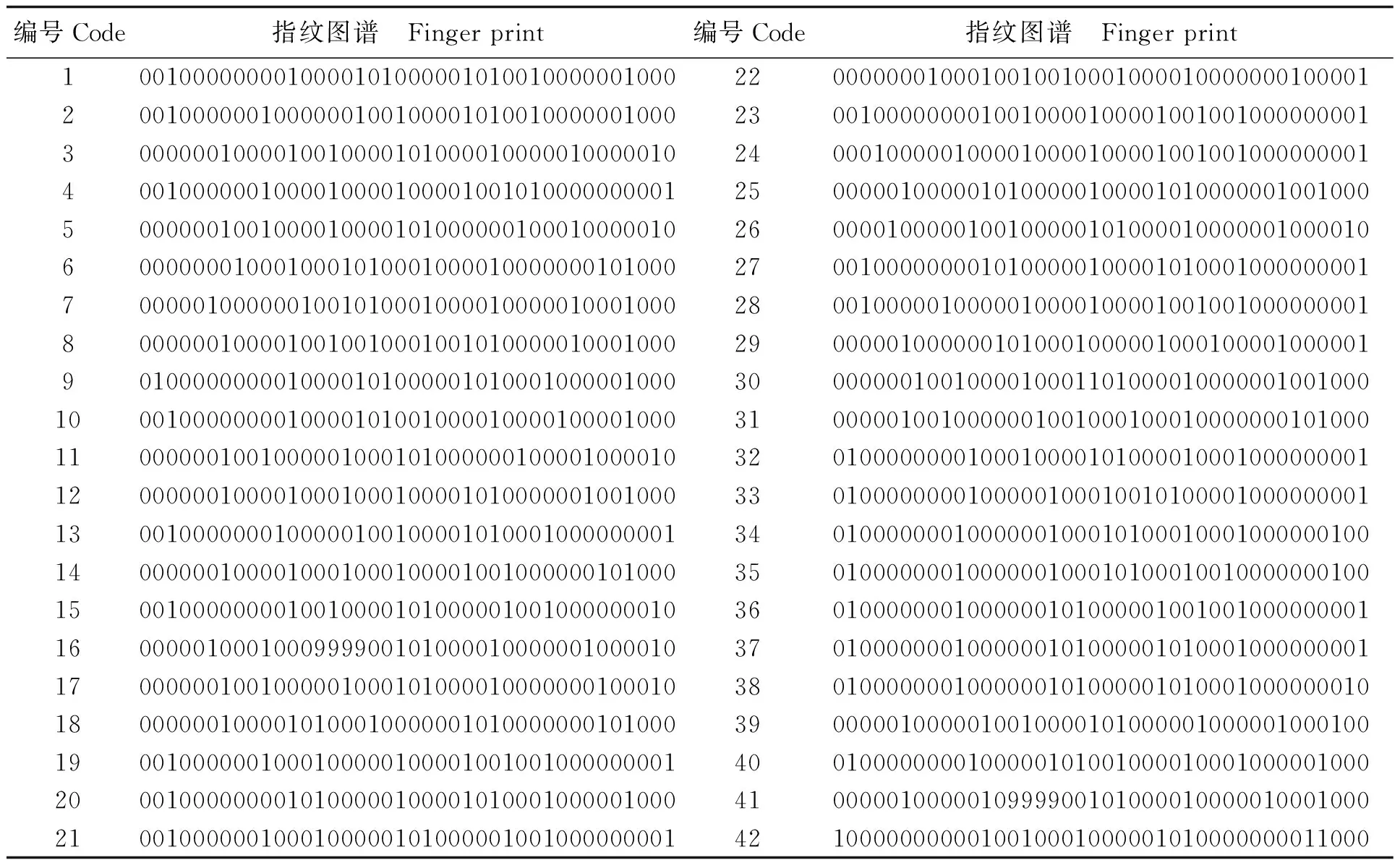
表4 8个SSR标记构建的42个花生品种的指纹图谱
2.2 42个花生品种DNA指纹图谱构建
采用23对SSR引物对42个花生品种进行指纹分析,远杂9102、郑农花12、冀花2号在引物AHGS1446检测图谱中有特征谱带,中花9号、中花15在引物IPAHM352检测图谱中有特征谱带,仲恺花1号在引物PM183和Seq3A01检测图谱中均有特征谱带,山花10号、远杂9102在引物TC23H10检测图谱中有特征谱带。除上述少数品种外,其他品种都不具有特征谱带,因此需要利用引物组合进行鉴定。通过分析,仅需要8对引物AHGS1446、AHGS1661、GNB827、IPAHM455、PM183、Seq3A01、TC23H10、TC25G11进行组合,即可将42个花生品种完全区分开。利用上述8对引物扩增结果构建42个花生品种的DNA指纹图谱(表4)。

图2 42个花生品种的UPGMA聚类分析 Fig.2 UPGMA clustering of 42 peanut varieties
2.3 42个花生品种特异性分析
利用23对引物扩增得到的92条多态性条带进行聚类分析,得到供试材料的遗传关系树状图(图2)。结果表明,所有供试品种间差异位点数均≥2,其中山花8号与山花10号、豫花15与冀0212-4、郑农花12与郑农花13相似系数较高,仅有2对引物可以将其分开。在相似系数0.74处,可以将42个花生品种分为4个类群。第I类群包含17个花生品种,分别为花育56、花育45、山花8号、山花10号、花育33、豫花15、冀0212-4、豫花9620、郑农花13、郑农花12、潍花9号、冀0607-17、豫花9306、天府23、豫花9925、中花8号和徐花9号。第II类群包含17个花生品种,分别为花育31、山花12、铁花15、山花15、海花3号、濮花30、驻29150、远杂9102、天府20、山花9号、中花9号、濮花22、冀花2号、濮花28、中花15、山花7号和丰花1号。第III类群包含2个花生品种,分别是中花12和中花5号。第IV类群包含6个花生品种,分别是仲恺花1号、泉花865、闽花6号、泉花327、仲恺花10号和仲恺花12。
3 讨论与结论
SSR(Simple Sequence Repeat),简单序列重复,又称为微卫星DNA(microsatellite DNA)或短串联重复(STR),是一种真核生物基因组中普遍存在的重复序列。Zhao等[20]研究表明,在9274对已开发的SSR分子标记中,1323对具有多态性,多态性比率为14.5%。SSR标记因其在基因组分布广泛、多态性高以及共显性遗传等优点,是构建DNA指纹图谱的理想技术。本研究首先从实验室已筛选出的200余对具有多态性的SSR引物中选出23对扩增稳定、带型简单清晰的SSR引物对42个不同地区花生品种进行遗传分析。结果表明所有品种均有2个或2个以上差异位点,其中远杂9102、郑农花12、冀花2号、中花9号、中花15、仲恺花1号、山花10号7个品种可以用单一引物进行区分,其他品种用8对引物进行组合就可以全部区分开,并构建了42个花生品种DNA指纹图谱。从聚类分析结果来看,同一地区及同一育种单位培育的花生品种相似系数较高,这与各地区、各育种单位在育种过程中高频使用少数骨干亲本有关,导致花生品种遗传基础趋于狭窄[21-22]。本研究结果为品种保护和育种实践提供了理论依据。
[1] 佘花娣,陈景堂,黄亚群,等. 利用DNA指纹图谱进行农作物品种鉴定的研究进展[J]. 河北农业大学学报, 2003, 26: 28-30, 33.
[2] 唐浩,余汉勇,张新明,等. 水稻新品种测试的标准品种DNA指纹图谱多样性分析[J]. 植物遗传资源学报, 2015, 16(1): 100-106.
[3] 王伟,杨文鹏,戴保威,等. DNA指纹图谱技术及其在玉米遗传育种上的应用[J]. 种子,2008, 27(1): 46-50.
[4] 陈亮,郑宇宏,范旭红,等. 大豆SSR指纹图谱身份证的研究进展与展望[J]. 大豆科技, 2015(2): 38-43.
[5] 匡猛,杨伟华,许红霞,等. 中国棉花主栽品种DNA指纹图谱构建及SSR标记遗传多样性分析[J]. 中国农业科学, 2011, 44(1): 20-27.
[6] 王瑞,张福耀,程庆军,等. 利用SSR荧光标记构建20个高粱品种指纹图谱[J]. 作物学报, 2015, 41(4): 658-665.
[7] 翁跃进, Gurtu S, Nigam S N. 花生AFLP指纹图谱[J]. 中国油料作物学报, 1999, 21(1): 10-12.
[8] 李双铃,任艳,陶海腾,等. 山东花生主栽品种AFLP指纹图谱的构建[J]. 花生学报,2006, 35(1): 18-21.
[9] He G H, Meng R H, Newman M, et al. Microsatellites as DNA markers in cultivated peanut (ArachishypogaeaL.) [J]. BMC Plant Biology, 2003,3: 3.
[10] Cuc L M, Mace E S, Crouch J H, et al. Isolation and characterization of novel microsatellite markers and their application for diversity assessment in cultivated groundnut (Arachishypogaea) [J]. BMC Plant Biology, 2008, 8: 55.
[11] Macedo S E, Moretzsohn M C, Leal-Bertioli S C M, et al. Development and characterization of highly polymorphic long TC repeat microsatellite markers for genetic analysis of peanut [J]. BMC Research Notes, 2012, 5: 86.
[12] Wang H, Penmetsa R V, Yuan M, et al. Development and characterization of BAC-end sequence derived SSRs, and their incorporation into a new higher density genetic map for cultivated peanut (ArachishypogaeaL.) [J]. BMC Plant Biology, 2012, 12: 10.
[13] Shirasawa K, Koilkonda P, Aoki K, et al.Insilicopolymorphism analysis for the development of simple sequence repeat and transposon markers and construction of linkage map in cultivated peanut [J]. BMC Plant Biology, 2012, 12: 80.
[14] 刘冠明,郑奕雄,黎国良. 20个花生品种的SSR标记指纹图谱构建[J]. 中国农学通报, 2006, 22(6): 49-51.
[15] 詹世雄,刘冠明,郑奕雄,等. 40个珍珠豆型花生SSR指纹图谱的构建[J]. 种子,2012,31(6): 23-27.
[16] 胡晓辉,毛瑞喜,苗华荣,等. 山东省46个花生品种SSR指纹图谱构建与遗传多样性分析[J]. 核农学报, 2016, 30(10): 1952-1933.
[17] 孙子淇,张新友,徐静,等. 河南省审定花生品种的指纹图谱构建[J]. 作物学报,2016,42(10): 1448-1461.
[18] 任小平,郑艳丽,黄莉,等. 花生SSR核心引物筛选及育成品种DNA指纹图谱构建[J]. 中国油料作物学报, 2016, 38(5): 563-571.
[19] 郑永胜,张晗,王东建,等. 基于荧光检测技术的小麦品种SSR鉴定体系的建立[J]. 中国农业科学, 2014, 47(19): 3725-3735.
[20] Zhao Y, Prakash C S, He G. Characterization and compilation of polymorphic simple sequence repeat (SSR) markers of peanut from public database [J]. BMC Res Notes, 2012, 5: 362.
[21] 陈静, 胡晓辉, 苗华荣, 等. SSR标记分析国家北方花生区试品种的遗传多样性[J]. 植物资源遗传学报, 2009, 10(3): 360-366.
[22] 殷冬梅, 王允, 尚明照, 等. 花生优异种质的分子标记与遗传多样性分析[J]. 中国农业科学, 2010, 43(11): 2220-2228.
Construction of Molecular Fingerprint for 42 Peanut Varieties Using SSR Markers
YIN Liang, LI Shuang-ling, REN Yan, SHI Yan-mao, YUAN Mei*
(Shandong Peanut Research Institute, Qingdao 266100, China)
42 peanut varieties from different area of China were used as materials. The varieties were analyzed by using 23 pairs of polymorphic SSR primers. A total 92 alleles were amplified from 23 SSR loci. The average of alleles is 4, and the average of polymorphic information content (PIC) is 0.49. Further analysis showed that, only 8 pairs of primer can fully distinguish 42 peanut varieties. A total 40 alleles were amplified from these 8 pairs of primers. The fingerprint clustering analysis of 42 peanut varieties was constructed by 40 alleles. Cluster analysis revealed that 42 peanut varieties could be classified into four groups at a 0.74 genetic similarity coefficient.This result provide theoretical basis for peanut varieties protection and breeding improvement.
peanut; SSR; DNA fingerprinting; clustering analysis
10.14001/j.issn.1002-4093.2017.01.002
2017-02-16
国家自然科学基金(31471533);“十二五”农村领域国家科技计划(2013AA102602-7);山东省农业科学院农业科技创新工程(CXGC2016B02)
尹亮(1983-),男,山东胶南人,山东省花生研究所助理研究员,硕士,从事花生遗传育种研究。
*通讯作者:袁美(1972-),女,研究员,博士,主要从事花生生物技术研究。E-mail: yuanbeauty@163.com
S565.2024; Q755
A
