柔嫩艾美耳球虫江苏株SAG10基因的克隆及分析
2017-05-02仇保丰景瑾董蓉莲宋鸿雁刘春朱顺
仇保丰 景瑾 董蓉莲 宋鸿雁 刘春 朱顺星 邵义祥 刘文斌 李建


摘要:根据GenBank中已发表的柔嫩艾美耳球虫(Eimeria tenella) SAG10基因序列,利用计算机软件设计1对引物,从E. tenella江苏株的第2代裂殖子中成功克隆出SAG10基因。将该基因与GenBank中国内外不同E. tenella分离株的SAG10基因进行比对,发现E. tenella江苏株与北京株的SAG10基因同源性最高,高达99%,其后依次为杨凌株、豪顿株。4种不同分离株SAG10基因的ORF之间有11个碱基不同,其中3个为无义突变,8个为有义突变。分析4种分离株SAG10基因推导蛋白质的亲水性、抗原指数、表面可及性发现,国内外不同E. tenella分离株SAG10蛋白的抗原性总体比较接近且很强,但国内不同分离株之间的抗原性更加接近,并优于国外的豪顿株,这可能是由于氨基酸突变引起蛋白质的空间构象和亲水性等发生变化而引起。将上述SAG10基因序列与复顶门原虫的其他相关SAGs基因序列进行遗传进化分析,发现Eimeria属球虫SAGs基因之间相对比较保守,且与犬新孢子虫(Neospora caninum)、N. hughesi、刚地弓形虫(Toxoplasma gondii)的SAG基因之间同源性较低,与已有观点一致;但神经肉孢子虫(Sarcocystis neurona)的2条SAG1基因序列却与E. tenella的SAG1基因序列表现出更高的同源性,这与其他研究结论不符。
关键词:柔嫩艾美耳球虫;SAG10基因;克隆;分析
中图分类号: Q785文献标志码: A
文章编号:1002-1302(2017)03-0024-04
鸡球虫基因克隆始于1987年,迄今已有240多个基因被成功克隆,鸡球虫的重组疫苗也得到了广泛研究[1]。有分析认为,参与艾美耳球虫宿主细胞入侵过程的相关分子是最具应用潜力的保护性抗原,如子孢子和裂殖子表面抗原(surface antigens,簡称SAGs)以及微线、棒状体、折光体等分泌型亚细胞器的分泌蛋白[1-2]。柔嫩艾美耳球虫(Eimeria tenella)表面抗原SAG10(surface antigen 10)是一种C端具有疏水性的糖基化磷脂酰肌醇(glycosylphosphatidylinositol,简称GPI)锚定结构域的表面蛋白[3]。由于SAG10基因在E. tenella子孢子和第2代裂殖子阶段均有表达,且SAGs被认为在启动顶复门原虫识别、黏附和侵袭宿主细胞、免疫调节、免疫逃避、宿主特异性等方面均发挥着重要作用,因此国外很多学者正加大对SAG10等表面抗原的研究力度[3-6]。韩德强等[7]、麦博等[2]分别对E. tenella北京株、杨凌株的SAG10基因进行了研究,但目前尚无对E. tenella江苏株SAG10基因进行研究的报道。麦博等研究发现,SAG10在不同E. tenella株间存在抗原性差异,这与认为E. tenella不存在明显株间抗原性差异的传统观点相对立[2]。本研究通过对E. tenella江苏(Jiangsu)株的SAG10基因进行克隆,并将其与国内外学者在GenBank中公布的E. tenella豪顿(Houghton)株、北京(Beijing)株、杨凌(Yangling)株的SAG10基因进行比对和分析,为研究 E. tenella SAG10的生物学功能、筛选E. tenella保护性抗原等工作提供依据。
1材料与方法
1.1试验材料
1.1.1球虫卵囊
E. tenella分离自江苏省某鸡场,由南通市出入境检验检疫局有害生物检疫实验室鉴定并保存。
1.1.2试验鸡
1日龄AA肉鸡,购自江苏省南通市某商品鸡孵化场,饲养于严格消毒且无球虫的笼舍中,饲料中不含抗球虫药。
1.1.3试剂
焦碳酸二乙酯(DEPC),购自Amresco公司;莫洛尼氏鼠白血病病毒(M-MLV)、Oligo(dT)15 Primer,购自Promega公司;pMD18-T克隆载体、Taq DNA聚合酶、dNTP、DL 2 000 Marker、限制性内切酶BamHⅠ、EcoRⅠ、DNA胶回收试剂盒,均购自宝生物工程(大连)有限公司。
1.2试验方法
1.2.1引物设计与合成
根据Tabares等在GenBank中发表的E. tenella SAG10基因序列[3](登录号:AJ586552),利用Primer Premier 5.0软件设计1对特异性扩增引物,上游引物为5′-CA[ZZ(Z]GGATCC[ZZ)]ATGCTACAGCGGAAGCTAC-3′,下游引物为5′-TG[ZZ(Z]GAATTC[ZZ)]TAAAGTCATAATGCCGAAC-3′,扩增的目的片段长度为799 bp。为便于后续操作,分别在上、下游引物中设计了酶切位点BamHⅠ、EcoRⅠ(如下划线部分所示)。引物由宝生物工程(大连)有限公司合成。
1.2.2cDNA模板的制备
将30羽1日龄AA肉鸡饲养至14日龄,经口接种新鲜孢子化卵囊1×105个/羽,接种后 120 h 杀鸡取盲肠。参考文献[8]的方法分离和纯化 E. tenella 第2代裂殖子,再采用一步法[9]提取总RNA并进行反转录,以反转录cDNA作为PCR模板。
1.2.3SAG10基因的克隆及鉴定
采用特异性引物进行SAG10基因的PCR扩增。PCR反应体系:10×Taq buffer 2.5 μL、MgCl2(25 mmol/L)1.0 μL、dNTP Mix(10 mmol/L each)0.5 μL、上游及下游引物(50 μmol/L)各0.5 μL、Taq DNA聚合酶(5 U/μL)0.5 μL、灭菌双蒸水14.5 μL、cDNA模板5 μL。PCR反应条件:94 ℃ 5 min;94 ℃ 55 s,58 ℃ 55 s,72 ℃ 55 s,35个循环;72 ℃延伸15 min。循环完毕后,向扩增产物中加入5 μL上样缓冲液,在1%琼脂糖凝胶上电泳,并在紫外灯下观察结果。回收SAG10基因目的条带,连接pMD18-T载体并转化DH5α,用BamHⅠ+EcoRⅠ双酶切及PCR鉴定出阳性克隆,挑取3个克隆送至宝生物工程(大连)有限公司测序,将测序结果输入“http://www.ncbi.nlm.nih.gov/BLAST/”进行网上比对。
1.2.4SAG10蛋白的特性分析
采用DNAStar 4.0软件分别编辑GenBank中报道的E. tenella豪顿株、北京株、杨凌株、本研究克隆的江苏(Jiangsu)株的SAG10基因序列,分析开放阅读框(ORF)的碱基序列,并推导出编码蛋白的氨基酸序列。采用MegAlign Clustal V软件对SAG10基因ORF的碱基序列和推导氨基酸序列进行比对,分析E. tenella不同分离株之间SAG10基因碱基和推导氨基酸的突变情况,进一步采用DNAStar Protean软件预测这些突变引起SAG10蛋白亲水性结构(Hydrophilicity Plot)、抗原性指数(Antigenicindex)、表面可及性(Surface Probability Plot)的变化情况。
1.2.5SAG10基因遗传进化分析
分别收集若干条GenBank中公布的E. tenella和毒害艾美耳球虫、新孢子虫、肉孢子虫、弓形虫等其他顶复门原虫SAGs基因家族(SAGs gene family)序列,结合GenBank中已公布及本研究克隆的E. tenella SAG10基因序列,共同采用MEGA 6.06软件构建遗传进化发生树,分析本研究克隆SAG10基因和其他SAG基因之间的遗传进化关系。
2结果与分析
2.1SAG10基因的克隆
从E. tenella第2代裂殖子中提取总RNA并进行逆转录PCR(RT-PCR)扩增,产物经1%琼脂糖凝胶电泳检测,发现2个泳道中均有1条约799 bp的条带(图1),与预期大小相吻合。切胶并回收目的条带后,连接pMD18-T载体,转化大肠杆菌DH5α。
2.2SAG10基因的鉴定
提取pMD18-T-SAG10基因重组质粒,经双酶切鉴定,可获得与预期大小一致的目的條带,进一步采用PCR方法进行鉴定,也能得到约799 bp的条带(图2),与理论值相吻合。从双酶切和PCR鉴定的阳性克隆中挑取3个菌落进行DNA测序,然后将测序结果输入GenBank进行Blast比对, 发现本研究克隆的E. tenella江苏株SAG10基因与GenBank中公布的E. tenella北京株SAG10基因同源性最高,高达99%,其后依次为杨凌株、豪顿株,表明SAG10基因克隆成功。
2.3SAG10的蛋白特性分析
目前,GenBank中公布的E. tenella SAG10基因序列共有4条,其中同源性为100%的2条豪顿株SAG10基因序列由国外不同研究小组发表,北京株、杨凌株SAG10基因序列均由国内学者发表(表1)。这4条SAG10基因均只有完整的ORF部分,无两端序列,全长786 bp,编码261个氨基酸,蛋白分子量约为27.9 ku。本研究克隆的E. tenella江苏(Jiangsu)株SAG10基因全长799 bp,其中不仅包含E. tenella SAG10基因完整ORF,同时还包含引物设计过程中人为添加的保护碱基和酶切位点。采用DNAStar 4.0软件分析本研究克隆的SAG10基因的ORF,并推导出氨基酸序列,与GenBank中发表的4条序列共同采用MegAlign Clustal V软件比对ORF内碱基序列和推导氨基酸序列。结果发现,4种不同E. tenella分离株SAG10基因之间共有11个位点存在差异(本研究中引物设计时人为引入的突变未计算在内,下同),除去第426、435、552位3个位点的碱基突变为无义突变外,其余8个碱基突变均为有义突变,引起了8个氨基酸突变(表1)。进一步采用DNAStar Protean软件中的Kety-Doolittle、Jameson-Wolf、Emini算法,依次分析SAG10蛋白的亲水性结构、抗原指数、表面可及性的变化情况。结果显示,不同E. tenella分离株SAG10蛋白的N端和C端疏水性均较强,而中间段则有很多亲水性区域。国内外不同E. tenella分离株之间,SAG10蛋白抗原性总体比较接近,且抗原性很强,但国内不同分离株之间的抗原性更加接近,它们与国外豪顿株的氨基酸序列在140~160位差异均较大,且国内分离株的抗原性优于国外豪顿株(图3)。这可能是由于E. tenella国内分离株和国外豪顿株的SAG10氨基酸序列在140~160位变化频率较高(表1),引起蛋白质的空间构象和亲水性发生一定变化而导致。
160位氨基酸序列位于SAG10蛋白表面的可能性较大,一定程度上可解释为何该区域氨基酸突变频率较高会对国内外分离株之间抗原性差异产生影响。
2.4SAG10基因遗传进化分析
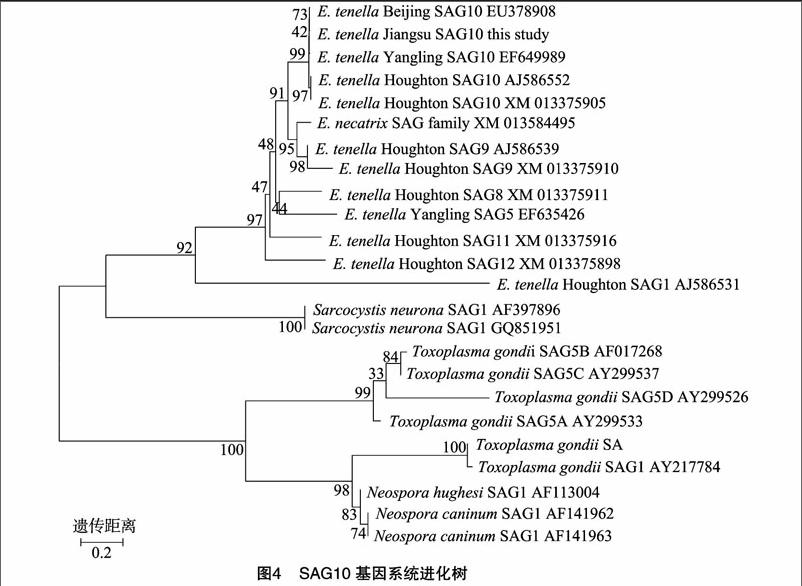
本研究收集了GenBank中已发表的E. tenella 4条SAG10基因和1~2条不等的SAG1、SAG5、SAG8、SAG9、SAG11、SAG12基因序列。同时收集了毒害艾美耳球虫(Eimeria necatrix)SAG基因家族序列和犬新孢子虫(Neospora caninum)、洪氏新孢子虫(Neospora hughesi)、神经肉孢子虫(Sarcocystis neurona)的SAG1基因序列,以及刚地弓形虫(Toxoplasma gondii)SAG1(别称P30)和SAG5(可分为A、B、C、D共4种多肽)的基因序列,结合本研究克隆的E. tenella SAG10基因序列,共同采用MEGA 6.06软件构建遗传进化发生树(图4)。结果表明,E. tenella和E. necatrix的SAGs基因家族序列均分布在进化树的同一较大分支上,表明艾美耳属(Eimeria)球虫的SAGs基因之间可能相对比较保守。N. caninum、N. hughesi、T. gondii的SAG基因则分布在进化树的另一较大分支上,其中同为新孢子虫属(Neospora)的N. caninum和N. hughesi SAG1基因序列也分布在同一亲缘关系较近的分支上,这与Eimeria属球虫SAGs基因的特性相似。另外,GenBank中已发表的4条E. tenella SAG10基因与本研究克隆SAG10基因的亲缘关系较近,分布在同一较小分支上,且E. tenella江苏株和北京株的SAG10基因分布最近,与“2.2”节中江苏株SAG10基因的BLAST结果一致。
已有研究认为,顶复门原虫的SAGs编码基因在组织寄生型原虫(包囊形成型原虫,如T. gondii、S. neurona、N. caninum)间是高度同源的,但Eimeria与隐孢子虫、疟原虫及这三者与弓形虫间无任何同源性[10-11]。本研究发现,T. gondii、N. caninum、N. hughesi、Eimeria球虫SAGs基因之间确实表现出了上述特点,但例外的是,S. neurona的2条SAG1基因却与T. gondii、N. caninum、N. hughesi的SAGs基因序列均表现出了较低的同源性,反而与Eimeria球虫SAGs基因分布在进化树的同一较大分支上,且与E. tenella的SAG1基因间表现出较高的同源性,这与上述已有观点明显不符。
3结论与讨论
GPI锚定蛋白是一类通过其羧基末端的糖基化磷脂酰肌醇结构锚定于真核细胞膜表面的蛋白,与传统的跨膜型表面蛋白不同,它们不跨越细胞膜脂质双层,只通过其羧基末端的GPI锚定于细胞上,同时这种GPI锚在磷脂酶C的处理下又可从细胞膜上脱落[12]。已发现的GPI锚定蛋白种类很多,包括水解酶、细胞黏附分子、表面抗原、受体、朊病毒等[13]。GPI锚定蛋白功能广泛,涉及细胞识别、信号转导、生长发育、分化[CM(25]、程序性死亡等重要生命过程,与血栓形成、白血病等很多[CM)][FL)]
[FK(W22][TPCBF4.tif][FK)]
疾病有着一定联系[14]。SAGs是一类GPI锚定蛋白,大量分布于弓形虫、疟原虫、隐孢子虫、新孢子虫等侵袭性阶段虫体细胞表面,是虫体与宿主细胞黏附的主要分子[10]。目前,弓形虫的SAGs已被深入研究,但针对E. tenella SAGs的研究仍有待进一步加强[3,10]。
麦博等对E. tenella杨凌株SAG10基因及其氨基酸序列进行了研究,发现SAG10在不同E. tenella株间存在抗原性差异[2]。简永利等对E. tenella杨凌株SAG2基因进行研究后也发现了类似现象[10]。但这些发现与认为E. tenella不存在明显株间抗原性差异的传统观点相对立。韩德强等对E. tenella北京株SAG10基因进行了研究,未报道是否发现这一现象[7]。为澄清这一问题,同时为研究E. tenella SAG10的生物学功能以及为筛选理想的E. tenella保护性抗原等提供信息,本研究对E. tenella江苏株的SAG10基因进行了克隆,并将它与国内外在GenBank中发表的E. tenella豪顿株、北京株、杨凌株的SAG10基因进行了系统比对和分析。结果表明,E. tenella江苏株SAG10基因与GenBank中收录的E. tenella北京株SAG10基因同源性最高,高達99%,其后依次为杨凌株、豪顿株SAG10基因。对4种不同E. tenella分离株SAG10基因和推导氨基酸序列进行研究发现,SAG10基因之间共有11个位点存在差异,其中8个碱基突变为有义突变,引起了8个氨基酸突变。分析SAG10蛋白的亲水性、抗原性、表面可能性等特性发现,国内外不同E. tenella分离株之间的SAG10蛋白抗原性总体比较接近,且抗原性很强,但国内不同分离株之间的抗原性更加接近,它们均与国外豪顿株的氨基酸序列在140~160位差异较大,且国内分离株的抗原性优于国外豪顿株。这可能是由于E. tenella国内分离株和国外豪顿株的SAG10氨基酸序列在140~160位变化频率较高,引起蛋白的空间构象、亲水性等发生变化而导致。
目前对于E. tenella SAGs的研究深度远远落后于弓形虫等其他复顶门原虫SAGs,因此在开展E. tenella SAGs的蛋白结构、生物学功能等研究过程中,常须要从弓形虫等其他复顶门原虫SAGs的研究中获得启发,并进行对比和推测。简永利等研究发现,E. tenella杨凌株与豪顿株的SAG2基因序列高度同源,但与T.gondii、Neospora caninum、Sarcosystis neurona SAG2无任何同源性[10]。更有观点认为,顶复门原虫的SAGs编码基因在组织寄生型原虫(包囊形成型原虫,如T. gondii、S. neurona、N. caninum)间是高度同源的,但Eimeria与隐孢子虫、疟原虫及这三者与弓形虫间无任何同源性[10-11]。为证实这一观点,本研究收集了GenBank中已发表的E. tenella 4条SAG10基因和1~2条不等的SAG1、SAG5、SAG8、SAG9、SAG11、SAG12的基因序列,同时收集了E. necatrix、N. caninum、N. hughesi、S. neurona、T. gondii的相关SAG基因序列,结合本研究克隆的E. tenella SAG10基因序列,共同采用MEGA 6.06软件构建遗传进化发生树。结果发现,E. tenella和E. necatrix的SAGs基因家族的序列均分布在进化树的同一较大分支上,表明同为Eimeria属的球虫SAGs之间可能相对比较保守。N. caninum、N. hughesi、T. gondii的SAG基因则分布在进化树的另一较大分支上,其中,同为Neospora属的N. caninum和N. hughesi SAG1基因序列也分布在同一亲缘关系较近的分支上,这与Eimeria属球虫SAGs基因的特性相似,也与上述其他学者的观点一致。但例外的是,S. neurona的2条SAG1基因却均与T. gondii、N. caninum、N. hughesi的SAG1基因序列表现出了较低的同源性,反而与Eimeria球虫SAGs基因分布在进化树的同一较大分支上,且与E. tenella的SAG1基因间表现出较高的同源性,这与已有研究结果[10-11]明显不符。导致这一异常现象的原因有待进一步研究,同时须分析比对更多的序列。
参考文献:
[1]曹利利,姚新华,侯洪烈,等. 柔嫩艾美耳球虫长春株SAG2基因的克隆与序列分析[J]. 动物医学进展,2013,34(12):115-120.
[2]麦博,覃宗华,于三科,等. 柔嫩艾美球虫杨凌株表面抗原SAG10基因的克隆与原核表达[J]. 中国兽医科学,2007,37(9):751-755.
[3]Tabarés E,Ferguson D,Clark J,et al. Eimeria tenella sporozoites and merozoites differentially express glycosylphosphatidylinositol-anchored variant surface proteins[J]. Molecular&Biochemical Parasitology,2004,135(1):123-132.
[4]Ngo H M,Hoppe H,Joiner K. Differential sorting and post-secretory targeting of proteins in parasitic invasion[J]. Trends in Cell Biology,2000,10(2):67-72.
[5]Frasch A C. Functional diversity in the trans-sialidase and mucin families in Trypanosoma cruzi[J]. Parasitology Today,2000,16(7):282-286.
[6]Lekutis C,Ferguson D,Grigg M,et al. Surface antigens of Toxoplasma gondii:variations on a theme[J]. International Journal for Parasitology,2001,31(12):1285-1292.
[7]韓德强,丁宏标,乔宇,等. 鸡柔嫩艾美耳球虫表面抗原基因sag10在毕赤酵母中的表达及鉴定[J]. 寄生虫与医学昆虫学报,2006,13(4):199-203.[HJ1.5mm]
[8]蒋建林,蒋金书. 柔嫩艾美耳球虫各阶段虫体纯化方法的改进[J]. 中国农业大学学报,1996,1(5):99-102.
[9]萨姆布鲁克 J,拉塞尔 D W. 分子克隆实验指南[M]. 3版.北京:科学出版社,2002:1256-1259.
[10]简永利,蔡建平,于三科,等. 柔嫩艾美耳球虫表面抗原SAG2基因的克隆与表达[J]. 畜牧与兽医,2006,38(6):10-13.
[11]Jung C,Lee C Y,Grigg M E. The SRS superfamily of Toxoplasma surface proteins[J]. International Journal for Parasitology,2004,34(3):285-296.
[12]熊茂林,宋畅,罗荣城,等. 人糖基磷脂酰肌醇(GPI)-B7-1真核表达载体的构建及表达[J]. 中国病理生理杂志,2005,21(1):154-158.
[13]苏建亚,沈晋良. 棉铃虫幼虫中肠非糖基磷酯酰肌醇锚着氨肽酶N基因的克隆和测序[J]. 昆虫学报,2005,48(3):444-449.
[14]张云华,黄瑾. GPI锚定蛋白的合成及功能[J]. 农垦医学,2012,34(1):59-62.
