金黄色葡萄球菌肠毒素及移动基因元件研究进展
2016-11-11唐俊妮
王 琼,唐俊妮*
(西南民族大学生命科学与技术学院,四川 成都 61004 1)
金黄色葡萄球菌肠毒素及移动基因元件研究进展
王 琼,唐俊妮*
(西南民族大学生命科学与技术学院,四川 成都 61004 1)
金黄色葡萄球菌肠毒素是一种热源性的超抗原。食用被肠毒素污染的食物能够引起食物中毒,导致恶心、呕吐、腹痛有时伴随腹泻。本文综述了肠毒素的分类命名、理化性质、以及编码不同肠毒素的基因在移动基因元件中的存在情况。这些移动基因元件在金黄色葡萄球菌毒力传播和进化过程中起着至关重要的作用,了解它们对于金黄色葡萄球菌流行病学溯源以及理解毒力机制具有一定的指导意义。
金黄色葡萄球菌;葡萄球菌肠毒素;理化 性质;移动基因元件
金黄色葡萄球菌(Staphylococcus aureus)是一种常见的条件致病菌,是引起医院和社区获得性感染的主要病原菌,也是导致肠毒素型食物中毒的主要病原菌。一些产毒素的金黄色葡萄球菌在生长的对数期或是从指数期到稳定期的转化过程中能够合成葡萄球菌肠毒素(staphylococcal enterotoxins,SEs)[1]。这类毒素蛋白对热、低pH值、蛋白酶均具有一定的抵抗力。当摄取了受金黄色葡萄球菌污染的食物后,其产生的肠毒素仍可以在消化道内保持一定的活性[2],从而导致食物中毒。特别是在食品加工过程中由操作人员直接接触或是呼吸道分泌物与食品表面间接接触,都可使金黄色葡萄球菌转移到食物当中。食用在加工过程中或后续常温贮藏时被金黄色葡萄球菌污染的食品,其产生的肠毒素是引起食物中毒的主要原因。引起葡萄球菌食物中毒频率较高的食物通常有:肉及肉制品,家禽及蛋制品,奶及奶制品,沙拉,焙烤 制品,尤其是奶油填充的甜点、蛋糕以及三明治的馅料等[3-4]。另外,金黄色葡萄球菌能够在较低的水分活度(aw<0.90[5])中生长,故腌制品如火腿中也经常能够检测出[6]。金黄色葡萄球菌肠毒素引起的食物中毒发生比较迅速(2~8 h),其症状包括恶心、剧烈呕吐、有时会伴随腹泻[7]。婴儿、老人和操劳过度的人属于易感人群[8]。因此,预防由金黄色葡萄球菌引起的食物中毒具有重要的意义。
本文综述了金黄色葡萄球菌肠毒素的分类、理化性质、以及金黄色葡萄球菌的移动基因元件(mobilegenetic elements,MGEs),并重点对能够编码金黄色葡萄球菌肠毒素的质粒、噬菌体、葡萄球菌毒力岛(staphylococcal pathogenicity islands,SaPIs)以及葡萄球菌盒式 染色体等移动基因元件进行了介绍。这些移动基因元件在金黄色葡萄球菌毒力传播和进化中起着非常重要的作用,了解它 们对于金黄色葡萄球菌流行病学溯源以及理解金黄色葡萄球菌引起食物中毒的毒力机制具有一定的理论与实践指导意义。
1 金黄色葡萄球菌肠毒素
1.1 分类与命名
金黄色葡萄球菌可以产生多种类型葡萄球菌肠毒素(SEs),并根据发现时间先后顺序按照英文字母进行区分,通常把这些肠毒素分为传统肠毒素(SEA~SEE)和新型肠毒素(SEG~SElX)。另一些学者根据这些肠毒素是否具有呕吐活性对金黄色葡萄球菌肠毒素做了新的分类:其中SEA~SEE、SEG、SEH、SEI、SER、SES和SET这11 种肠毒素已被证 明具有催吐活性,称为肠毒素SEs;而那些在灵长目动物实验中没有表现出催吐活性(如SElL和SElQ),或是还没有被证实是否具有催吐活性的肠毒素,如SElJ、SElK、SElM、SElN、SElO、SElP、SElU、SElU2、SElV、SElX称为金黄色葡萄球菌类肠毒素蛋白(staphylococcal enterotoxin-like proteins,SEls)[9]。值得一提的是,葡萄球菌中毒休克性毒素(toxic shock syndrome toxin-1,TSST-1)起初是被命名为SEF,后来改为TSST-1,该毒素无呕吐活性[10]。葡萄球菌肠毒素食物中毒症状通常是由传统和新型肠毒素共同作用引起。2013年,Omoe等[11]对7 种新型的肠毒素SElK、SElL、SElM、SElN、SElO、SElP和SElQ进行了灵长目动物毒素喂养实验,先用传统肠毒素SEA和SEB进行了实验动物(猕猴)毒素易感性实验,表明该组猕猴对不同浓度的SEA和SE B均表现出一定程度的呕吐反应。然后再对该组猕猴按每千克体质量100 μg的剂量喂养新型毒素蛋白,实时监控记录喂养5 h内猕猴的呕吐次数、开始呕吐时间以及行为变化等,实验结果显示所有受试猕猴对7 种新型肠毒素均能产生不同程度的呕吐反应,只是受影响的猕猴数目明显少于传统的SEA和S EB影响的猕猴数目。7 种新型肠毒素在1~4.5 h内产生了呕吐 现象,呕吐次数在2~36 次不等,由此建议国际命名委员会(葡萄球菌超抗原)(International Nomenclature Committee for Staphylococcal Superantigens,INCSS)将这些类肠毒素蛋白SEls重命名为肠毒素SEs[12]。
1.2 肠毒素结构与性质
所有的金黄色葡萄球菌肠毒素蛋白均含有二硫键和单一多肽链,不含有碳水化合物、脂肪和核酸,是一类基本结构相似 的胞外蛋白[13]。组成这些胞外蛋白的氨基酸数目各不相同,约在220~240 个之间,平均分子质量约为25 kD,序列之间存在较显著的差异性,但折叠组装后的三维结构相对保守,一般是由多个β-折叠和少数α-螺旋结构构成的两个不均等区域,这两个区域被一条凹槽分开,整体呈现椭圆体结构,较大的区域包含氨基末端和羧基末端[14]。肠毒素的本质是超抗原(super antigen,SAg),可以结合于抗原呈递细胞(antigen presenting cell,APC)表面的主要组织相容性复合体Ⅱ分子(major histocompatibility complexⅡ, MHCⅡ)上的α-螺旋区凹槽或T细胞受体(T cell receptor,TCR)可变的Vβ区域,刺激T细胞的增殖并释放细胞因子[15],产生强烈的细胞毒性作用,导致严重的炎症反应。但是不同肠毒素与MHCⅡ以及TCR相互作用时的位点不一样[16]。一些研究还发现肠毒素的超抗原特性对肿瘤细胞也具有一定的杀伤作用[17]。肠毒素蛋白具有很强的耐热耐酸性能,被肠毒素污染的食物如果加工不彻底,毒素仍可保持活性。进入胃肠道后,胃肠道的蛋白酶包括胃蛋白酶、胰蛋白酶、木瓜蛋白酶以及凝乳酶等不易使其失活。因此,食用被金黄色葡萄球菌污染的食物后,其产生的肠毒素在体内存留的时间较菌体本身更长,从而更容易造成呕吐和胃肠道疾病[2]。
2 编码金黄色葡萄球菌肠毒素的移动基因元件
金黄色葡萄球菌存在多种不同类型的MGEs,如质粒、转座子(transposons,Tn)、插入序列(insertion sequences,IS)、噬菌体、SaPIs以及葡萄球菌盒式染色体(staphylococcal cassette chromosome mec,SCCmec)等。它们在金黄色葡萄球菌适应环境以及细菌物种间遗传信息传递过程中起着中心作用[18]。移动基因元件对于金黄色葡萄球菌毒力的传播和进化具有重要作用。其中噬菌体、质粒、葡萄球菌毒力岛等移动基因元件能够编码 金黄色葡萄球菌肠毒素[19]。肠毒素基因在这些元件中的转移能够导致新的毒素基因产生,而菌株间基因的水平转移,又可导致不同菌株基因组间的差异以及高致病性菌株的产生。
2.1 质粒
质粒具有水平基因转移能力,使其成为金黄色葡萄球菌抗性基因和毒力基因传递的有效载体。目前,已经证实有2 种质粒可以携带金黄色葡萄球菌肠毒素基因。第一个被证实含有肠毒素基因的质粒是pIB485,它含有编码肠毒素SED和SElJ的基因。随后的研究表明selj基因可能出现在所有能够编码SED肠毒素的质粒上[20]。Ono等[21]研究发现2 种新型的超抗原肠毒素SES和SET,均存在于质粒pF5中。该质粒在1997年从日本福冈发生的食物中毒有关的金黄色葡萄球菌Fukuoka 5中分离得到。SES、SER和 SET在麝香鼩鼱以及灵长目动物喂养实验中,经验证均能表现出呕吐反应。目前,国内对肠毒素基因位点也有一些研究,如吴可可等[22]对15 株多重耐药金黄色葡萄球菌质粒消除前后进行肠毒素测定的比较,发现肠毒素sea、sec基因位于染色体上,sed基因位于质粒上,而seb基因可以位于染色体或质粒上。Omoe等[23]发现肠毒素SER也可由2 类质粒编码:一类可能与众所周知编码SED和SEJ肠毒素的质粒pIB485有关,称 为pIB485-like质粒;另外一类质粒是从Fukuoka 5、Fukuoka 6和Fukuoka 7菌株中纯化出来的3 种质粒,分别命名 为pF5、pF6和pF7。通过把这3 种质粒和pIB485-like质粒利用ser探针通过Southern 杂交分析,发现ser基因也存在于pF5、pF6和pF73种质粒中。目前已经发现的可由质粒编码的肠毒素基因可归纳为sed、selj、ser、ses及set,详见表1。
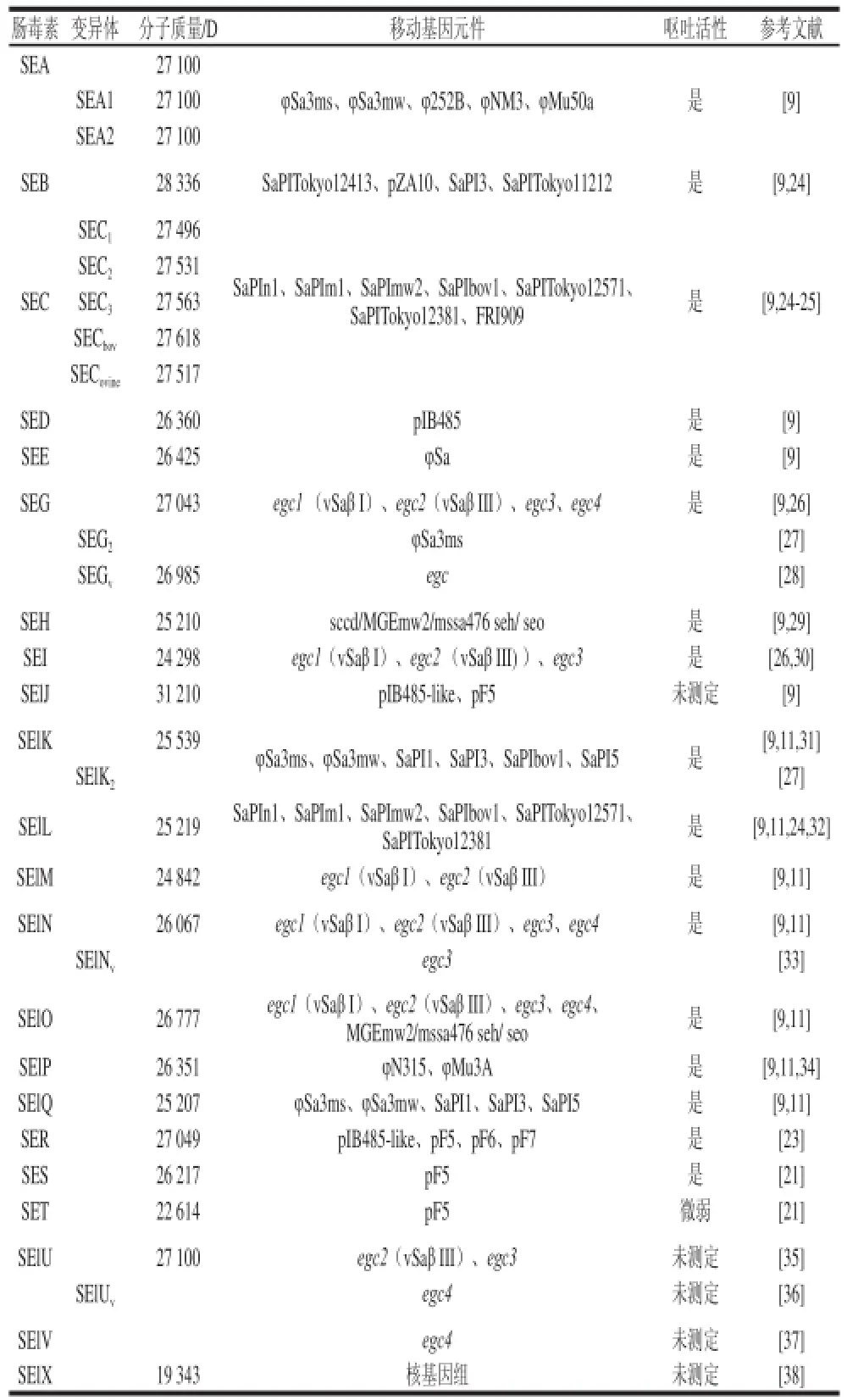
表1 肠毒素以及类肠毒素蛋白基因的位点及性质Table1 General properties of SEs and SEls and genomic location of the encoding genes
2.2 噬菌体
噬菌体也是水平基因转移的主要载体,可为金黄色葡萄球菌提供种类广泛的遗传变异潜力。它能在感染过程中增加金黄色葡萄球菌基因组的可塑性,提高病原体适应各种宿主环境的能力[39]。大多数已报道的携带肠毒素基因(sea、selp、selk、selq)的金黄色葡萄球菌噬菌体均属于长尾噬菌体科[9]。其中,编码SEA肠毒素的噬菌体有φSa3ms、φSa3mw、φ252B、φNM3、φMu50A;编码SElP肠毒素的噬菌体有φN315、φMu3A;编码SElK和SElQ肠毒素的噬菌体有φSa3ms、φSa3mw[39];肠毒素基因seg2和selk2存在于噬菌体φSa3ms的两端[27]。由噬菌体编码的肠毒素基因详见表1,可归纳为sea、see、seg、selk、selp及selq。
2.3 葡萄球菌毒力岛
SaPIs通常携带超抗原基因和抗性基因,在金黄色葡萄球菌和其他革兰氏阳性细菌中广泛存在。其整体基因结构高度保守,与典型的温和噬菌体基因结构类似。每个SaPI占据一个特定的染色体位点,被一个或多个特定的金黄色葡萄球菌噬菌体诱导进行删除和复制。复制之后,SaPI的DNA片段可被辅助噬菌体包被,导致非常高的转换频率。大多数金黄色葡萄球菌含有一种以上的SaPI,SaPIs之间存在重组现象,如引起中毒休克综合征的临床菌株都包含2 种或2 种以上由多种基因联合在一起的SaPIs。目前,临床分离的产肠毒素金黄色葡萄球菌菌株的毒力岛有SaPI1、SaPI2、SaPI3、SaPI4、SaPI5、SaPIn1/m1、SaPI1mw2以及从牛乳房炎中分离得到的突变体SaPIbov1和SaPIbov2[40]。最近,Suzuki等[24]利用SaPI扫描方法又发现了4 种新的SaPIs,它们分别是SaPITokyo12413、SaPITokyo11212、SaPITokyo12571以及SaPITokyo12381。通过鉴定这4 种新型编码肠毒素的SaPIs,并检测它们的毒性以及复制能力,发现这4 种新型的SaPIs均能够使金黄色葡萄球菌产生足够的肠毒素引起食物中毒。其中SaPITokyo12413的复制能力受到限制,它缺乏其他SaPIs的转移能力。Alibayov等[41]对93 株金黄色葡萄球菌已知的8 种SaPI整合酶基因和13 种金黄色葡萄球菌肠毒素基因(sea、seb、sec、sed、see、seg、seh、sei、selj、sell、selk、selq、tst)的分布情况进行调查。利用多重聚合酶链式反应(polymerase chain reaction,PCR)技术与脉冲凝胶电泳方法分析了已知所有的SaPIs和这些肠毒素基因之间的关系,从而建立了一种快速检测金黄色葡萄球菌肠毒素基因型的方法,对于金黄色葡萄球菌的分型研究具有一定意义。金黄色葡萄球菌毒力岛中肠毒素基因的存在情况具体归纳见表2。
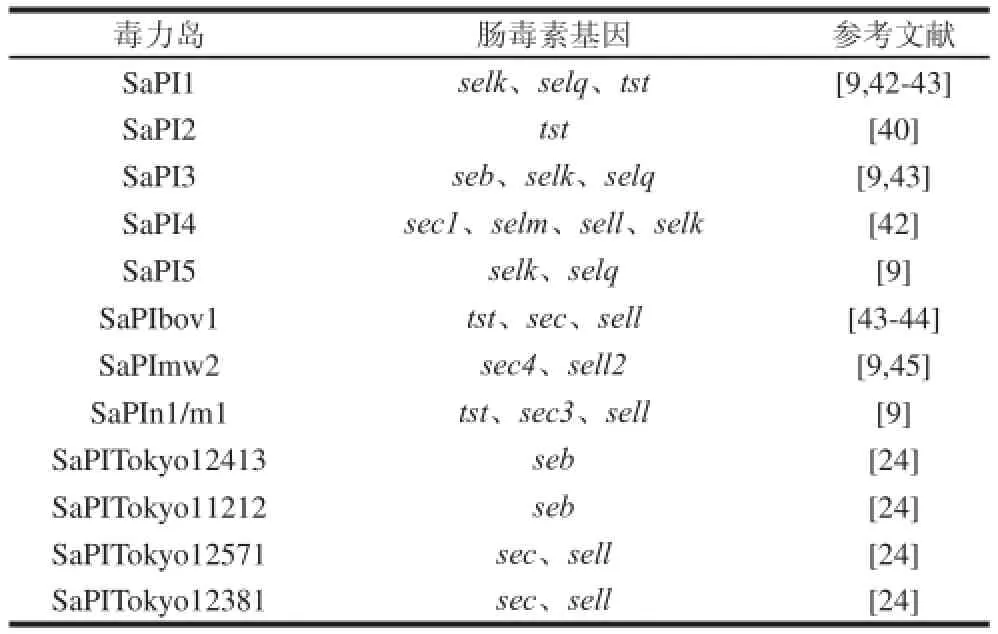
表2 肠毒素基因在毒力岛中的存在情况Table2 le 2 SEsSEs and and SElsSEls location on SaPIs
2.4 肠毒素基因簇(enterotoxin gene cluster,egc)
Jarraud等[26]在研究2 种新型肠毒素SEG和SEI时,经测序分析sei和seg基因间的DNA序列以及相邻区域的核酸序列发现了3 种与seg和sei相关的肠毒素类似的开放阅读框,分别命名为sell、selm、seln以及2 种假想基因φent1和φent2。随后,经实时荧光定量PCR(real-time quantitative polymerase chain reaction,RT-qPCR)分析,所有这些基因共同拥有同一个启动子,命名为肠毒素基因簇(enterotoxin gene cluster,egc)。随后Letertre等[35]发现在egc 2 种假想基因φent1和φent2序列之间插入15 个碱基可形成一种新的开放阅读框,编码肠毒素SEU,这一假定源于SEU蛋白与目前已知的肠毒素具有同源性但又不同于任何一种肠毒素。一些菌株的egc位点不完整,通常含有一个插入序列和转座子酶基因,这些情况也许代表egc位点在进化上存在中间阶段。Thomas等[36]研究发现一株具有非典型egc位点的金黄色葡萄球菌可产生2 种新型的肠毒素,命名为SElV和SElU2。其中SElV是由selm和sei重组得到,限制性删除φent1和φent2假基因之间的序列则形成SElU2。egc在临床分离的金黄色葡萄球菌中广泛分布。Rosec等[46]对从不同食品中采集的332 株金黄色葡萄球菌进行PCR检测,发现编码SEG、SEH、SEI、SElJ等新型肠毒素基因的菌株比编码传统型肠毒素的菌株数目高2 倍。Smyth等[47]针对从动物中分离的191 株金黄色葡萄球菌进行基因检测,发现egc编码的新型肠毒素基因seg、sei、sell、selm、seln和selo以及传统肠毒素基因sec在动物源的金黄色葡萄球菌中检出率较高。通过对所有已知肠毒素进行分类和进化分析,推断egc可能是肠毒素基因的温床,它存在4 种变异型,分别命名为:egc1、egc2、egc3和egc4。其中,egc1编码selo、selm、sei、φent1、φent2、seln、seg;egc2编码selo、selm、sei、selu、seln、seg;egc3编码selov、selmv、seiv、seluv、selnv、segv;以及egc4编码selo、sellv、selu2、seln、seg[48]。目前已验证egc编码的SEG、SEI、SElM、SElN、SElO蛋白均能够使灵长目动物产生呕吐反应[11,49],Chen等[50]也报道了由SEG和SEI引起的食物中毒事件,说明大部分由egc编码的肠毒素均可能引起食物中毒。
2.5 其他基因位点
前面对大部分肠毒素基因在移动基因元件上的编码情况进行了介绍,还有2 种肠毒素基因seh和selx并未出现在这些常见的移动基因元件中。肠毒素SEH的编码基因seh存在于耐甲氧西林菌株的盒式染色体下游的一个移动元件上,获取这一移动元件时发现这个已知的毒力基因能够稳定地在这些菌株的甲氧西林耐药基因染色体中整合[51]。Wilson等[37]发现了一种新型的肠毒素SElX,它是由核基因组(core genome)编码,具有超抗原生物活性。selx基因能够在体外表达,人、牛、绵羊等均能被携带selx基因的金黄色葡萄球菌感染。进化分析显示selx基因是通过点突变和至少17 个不同的等位基因重组形成的突变体。SElX对兔子具有致死性,是一种新型毒力因子。这种由特殊的核基因组编码的超抗原,为金黄色葡萄球菌进化提供了一种新的视野,也为金黄色葡萄球菌引起的感染提供新的分子基础。Remortel等[25]完整测序含有超抗原基因组的表皮葡萄球菌FRI909,发现它能够转录和翻译表达肠毒素SEC3和肠毒素类似毒素蛋白SElL。由此看来,葡萄球菌肠毒素不只属于金黄色葡萄球菌这个种所特有,这一现象可能与其编码肠毒素的移动元件的水平转移密切相关,未来通过对更多临床分离菌株全基因组进行测序,发现更多关于肠毒素基因的新信息。
3 移动基因元件与毒素基因表达
金黄色葡萄球菌基本代谢功能是由核基因组编码,而大多数毒力基因则需要其他移动基因元件编码,移动基因元件在细菌进化以及毒力传播过程中起着关键作用[52]。金黄色葡萄球菌的移动基因元件大约占整个基因组的15%[40]。当环境条件变化时,细菌体内的移动基因元件能够在辅助噬菌体协助下有效移动,进行高频率转换,使金黄色葡萄球菌的超抗原和毒素具有多样性[53],这有助于金葡对不同宿主以及不同环境条件的适应性,提高其作为一种病原菌的致病力。
最近,一些学者对毒素基因的表达进行研究,如Pocsfalvi等[54]研究发现在细菌对数生长后期和稳定期通过双向凝胶电泳检测不到egc启动子编码的蛋白。Derzelle等[1]也发现egc编码的肠毒素基因selo、selm、sei、seln、seg和selu在对数生长后期转录量相对于其他肠毒素要低。另外,研究发现seb基因主要存在于SaPI中,sed基因主要存在于质粒上,但二者表达上存在一致性。Tseng等[55-56]发现附属基因调节系统(accessory gene regulatory system,Agr)能够在生长的后期抑制毒素阻遏子(repressor of toxins,Rot)的活性,从而提高seb和sed基因的表达。同样,Kusch等[57]等研究发现egc启动子主要在细菌浓度较低时转录,seb基因的转录则主要出现于细菌浓度较高的情形,由噬菌体编码的sea、sek、selq、selp的转录则在生长过程中变化不大;此外,还发现σB(Sigma B)因子能够刺激seh、tst1和egc的转录。另外,毒素基因的表达与环境条件也紧密相连,如食物基质对毒素基因表达的影响等,未来还需要做进一步的研究。
4 结 语
金黄色葡萄球菌肠毒素能够引起食物中毒,对人类健康和食品安全造成极大威胁。由于金黄色葡萄球菌肠毒素种类繁多,同一菌株可同时携带多种肠毒素基因。金黄色葡萄球菌移 动基因元件能够编码肠毒素,在细菌进化以及毒力传播过程中起着关键作用,肠毒素基因在这些元件中的转移与重组导致新的肠毒素基因产生。通过研究金黄色葡萄球菌不同肠毒素基因型在不同移动基因元件中分布差异性,可对了解不同菌株之间毒力基因转移,金黄色葡萄球菌肠毒素引起食物中毒的监控、检测以及追踪溯源提供依据。
[1] DERZELLE S, DILASSER F, DUQUENNE M, et al. Differential temporal expression of the staphylococcal enterotoxins genes during cell growth[J]. Food Microbiology, 2009, 26(8): 896-904. DOI:10.1016/j.fm.2009.06.007.
[2] EVENSON M L, HINDS M W, BERNSTEIN R S, et al. Estimation of human dose of staphylococcal enterotoxin A from a large outbreak of staphylococcal food poisoning involving chocolate milk[J]. International Journal of Food Microbiology, 1988, 7(4): 311-316. DOI:10.1016/0168-1605(88)90057-8.
[3] TAMARAPU S, MCKILLI J L, DRAKE M. Development of a multiplex polymerase chain reaction assay for detection and differentiation of Staphylococcus aureus in dairy products[J]. Journal of Food Protection, 2001, 64(5): 664-668.
[4] WIENEKE A A, ROBERTS D, GILBERT R J. Staphylococcal food poisoning in the United Kingdom, 1969-90[J]. Epidemiology and Infection, 1993, 110(3): 519-531. DOI:10.1017/S0950268800050949.
[5] SCOTT W J. Water relations of Staphylococcus aureus at 30 ℃[J]. Australian Journal of Biological Sciences, 1953, 6(4): 549-564. DOI:10.1071/BI9530549.
[6] QI Y, MILLER K J. Effect of low water activity on staphylococcal enterotoxin A and B biosynthesis[J]. Journal of Food Protection, 2000, 63(4): 473-478.
[7] BALABA N, RASOOLY A. Staphylococcal enterotoxins[J]. International Journal of Food Microbiology, 2000, 61(1): 1-10.
[8] MURRAY R J. Recognition and management of Staphylococcus aureus toxin-mediated disease[J]. Internal Medicine Journal, 2005, 35(Suppl 2): 106-119. DOI:10.1111/j.1444-0903.2005.00984.x.
[9] ARGUDIN M A, MENDOZA M C,RODICIO M R. Food poisoning and Staphylococcus aureus enterotoxins[J]. Toxins, 2010, 2(7): 1751-1773. DOI:10.3390/toxins2071751.
[10] BERGDOLL M S, CRASS B A, REISER R F, et al. A new staphylococcal enterotoxin, enterotoxin F, associated with toxic-shocksyndrome Staphylococcus aureus isolates[J]. The Lancet, 1981, 1: 1017-1021. DOI:10.1016/S0140- 6736(81)92186-3.
[11] OMOE K, HU D L, ONO H K, et al. Emetic potentials of newly identified staphylococcal enterotoxin-like toxins[J]. Infection and Immunity, 2013, 81(1 0): 3627-3631. DOI:10.1128/IAI.00550-13.
[12] LIN A G, BOHACH G A, NAIR S P, et al. Standard nomenclature for the superantigens expressed by staphylococci[J]. Journal of Infectious Diseases, 2004, 189(12): 2334-2336. DOI:10.1086/420852.
[13] MARRACK P, KAPPLER J. The staphylococcal enterot oxins and their relatives[J]. Science, 1990, 248: 705-711. DOI:10.1126/ science.2185544.
[14] THOMAS D, CHOU S, DAUWALDER O, et al. Diversity in Staphylococcus aureus enterotoxins[J]. Chemical Immunology and Allergy, 2007, 93: 24-41. DOI:10.1159/000100856.
[15] FRIEDMA N M, RASOOLY R. Review of the inhibition of biological activities of food-related selected toxins by natural compounds[J]. Toxins, 2013, 5(4): 743-7 75. DOI:10.339 0/toxins5040743.
[16] MCCORMICK J K, YARWOOD J M, SCHLIEVERT P M. Toxic shock syndrome and bacterial superantigens: an update[J]. Annual Revie w of Microbiology, 20 01, 55(2): 77-104. DOI:10.1146/annurev. micro.55.1.77.
[17] BUZBY J C, ROBERTS T. Economic costs and trade impacts of microbial foodbor ne il lness[J]. World Healt h Statistics Quarterly, 1997, 50 (1/2): 57-66.
[18] MALACHOWA N, DELEO F R. Mobile genetic ele ments of Staphylococcus aureus[J]. Cellular and Molecular Life Sciences, 2010, 67(18): 3057-3071. DOI:10.1007/s00018-010-0389-4.
[19] BABA T, BAE T, SCHNEEWIND O, et al. Genome sequence o f Staphylococcus aureus strain newman and comparative analysis of staphylococcal genomes: polymorphism and evolution of two major pathogenicity islands[J]. Jour nal of Bacteriology, 2008, 190(1): 300-310. DOI:10.1128/JB.01000-07.
[20] ZHANG S, IANDOLO J J, STEWART G C. The enterotoxin D plasmid of Staphylococcus aureus encodes a second enterotoxin determinant (sej)[J]. FEMS Microbiology Letters, 1998, 16(2): 227-233. DOI:http://d x.doi. org/10.1111/j.1574-6968.1998.tb13278.x.
[21] ONO H K, OMOE K, IMANISHI K, et al. Identification and characterization of two novel staphylococcal enterot oxins, types S and T[J]. Infection and Im munity, 2008, 76(11): 4999-5005. DOI:10.1128/ IAI.00045-08.
[22] 吴可可, 吴跃进, 陈慧燕. 食源性金黄色葡萄球菌耐药基因和肠毒素基因定位[J]. 中国卫生检验杂志, 2013, 23(2): 498-499; 502.
[23] O MOE K, HU D L, TAKAHASHI-OM OE H, et al. Identification and characterization of a new staphylococcal enterotoxin-related putative toxin encoded by two kinds of plasmids[J]. Infection an d Immunity, 2003, 71(10): 6088-6094. DOI:10.1128/IAI.71.10.6088-6094.2003.
[24] SUZUKI Y, KUBOTA H, SATO’O Y, et al. Identific ation and characterization o f novel Staphylococcus aureus pathogenicity islands encoding staphylococcal enterotoxins originating from staphylococcal food poisoning isolates[J]. Journal of Appl ied Microbiology, 2015, 118(6): 1507-1520. DOI:1 0.1111/jam.12786.
[25] REMORTEL B, PARK J Y, HWANG S Y, et al. An enterotoxinbearing pathogenicity island in Staphylococcus epidermidis[J]. Journal of Bacteriology, 2011, 193(8): 1854-1862. DOI:10.1128/JB.00162-10.
[26] JARRAUD S, PEYRAT M A, LIM A, et al. egc, A highly prevalent operon of enterotoxin g ene, forms a putative nursery of superantigens in Staphylococcus aureus[J]. The Journal of Immunology, 2001, 166(1): 669-677. DOI:10.4049/ jimmunol.166.1.669.
[27] SUMBY P, WALD OR M K. Transcription of the toxin genes present within the staphylococcal phageφSa3ms is intimately linked with the phage’s life cycle[J]. Journal of Bacteriology, 2003, 185(23): 6841-6851. DOI:10.1128/JB.185.23.6841-6851.2003.
[28] ABE J, ITO Y, ONIMARU M, et al. Characterization and distribution of a new enterotoxin-relat ed superantigen produced by Staphylococcus aureus[J]. Microbiology and Immunology, 2000, 44(2): 79-88. DOI:10.1111/j.1348-0421.2000.tb01250.x.
[29] SU Y C, WONG A C. Identification and purification of a new sta phylococcal enterotoxin, H[J]. Applied and Environmental Microbiology, 1995, 61(4): 1438-1443.
[30] MUNSON S H, TREMAINE M T, BETLEY M J, et al. Identification and characterization of staphylococcal enterotoxin types G and I from Staphylococcus aureus[J]. Infection and Immunity, 1998, 66(7): 3337-3348.
[31] ORWIN P M, LEUNG D Y, DONAHUE H L, et al. Biochemical and biological properties of staphylococcal enterotoxin K[J]. Infection and Immunity, 2001, 69(1): 360-366. DOI:10.1128/IAI.69.1.360-366.2001.
[32] ORWIN P M, FITZGERALD J R, LEUNG D Y, et al. Characterization of Staphylococcus aureus entero toxin L[J]. Infection and Immunity, 2003, 71(5): 2916-2919. DOI:10.1128/IAI.71.5.2916-2919.2003.
[33] SCHELIN J, WALLIN-CARLQUIST N, COHN M T, et al. The formation of Staphylococcus aureus enterotoxin in food environments and advances in risk assessment[J]. Virulence, 2011, 2(6): 580-592. DOI:10.4161/viru.2. 6.18122.
[34] OMOE K, IMANISHI K, HU D L, et al. Characterization of novel staphylococcal enterotoxin-like toxin type P[J]. Infection and Immunity, 2005, 73(9): 5540-5546. DOI:10.1128/IAI.73.9.5540-5546.2005.
[35] LETERTRE C, PERELLE S, DILASSER F, et al. Identification of a new putative enterotoxin SEU encoded by the egc cluster of Staphylococcus aureus[J]. Journal of Applied Microbiology, 2003, 95(1): 38-43. DOI:10.1046/j.1365-2672.2003.0195 7.x.
[36] THOMAS D Y, JARR AUD S, LEMERCIER B, et al. Staphylococcal enterotoxin-like toxins U2 and V, two new staphylococcal superantigens arising from recombination within the enterotoxin gene cluster[J]. Infection and Immunity, 2006, 74(8): 4724-47 34. DOI:10.1128/IAI.00132-06.
[37] WILSON G J, SEO K S, CARTWRIGHT R A, et al. A novel core genome-encoded superantigen contributes to lethality of communityassociated MRSA necrotizing pneumonia[J]. PLoS Pathogens, 2008, 7(10): 1002271. DOI:10.1371/journal.ppa t.1002271.
[38] GOERKE C, WIRTZ C, FLÜCKIGER U, et al. Extensive phage dynamics in Staphylococcus aureus contributes to adaptation to the hum an host during infection[J]. Molecular Microbiology, 2006, 61(6): 1673-1685. DOI:10.1111/j.1365-2958.2006.05354.x.
[39] GOERKE C, PANTUCEK R, HOLTFRETER S, et al. Diversity of prophages in dominant Staphyloco ccus aureus clonal lineages[J]. Journal of Bact eriology, 2009, 191(11): 3462-3468. DOI:10.1128/ JB.01804-08.
[40] NOVICK R P, CHRISTIE G E, PENADES J R. The phage-related chromosomal islands of Gram-posit ive bacteria[J]. Nature Reviews Microbiology, 2010, 8(8): 541-551. DOI:10.1038/nrmicro2393.
[41] ALIBAYOV B, ZDENKOVA K, SYKOROVA H, et al. Molecular analysis of Staphylococcus aureus pathogenici ty islands (SaPI) and their superantigens combination of food samples[J]. Journal of Microbiological Methods, 2014, 107: 197-204. DOI:10.1016/ j.mimet.2014.10.014.
[42] NOVICK R. Mobile genetic elements and bacterial toxi noses: the superantigen-encoding pathogenicity islands of Staphylococcus aureus[J]. Plasmid, 2003, 49 (2): 93-105. DOI:10.1016/S0147-619X(02)00157-9.
[43] YARWOOD J M, MCCORMICK J K, PAUSTIAN M L, et al. Characterization and expression analysis of Staphylococcus aureus pathogenicity island 3. Implications for the evolution of staphylococcal pathogenicity islands[J]. The Journal of Biological Chemistry, 2002, 277(15): 13138-13147. DOI:10.10 74/jbc.M111661200.
[44] FITZGERALD J R, MONDAY S R, FOSTER T J, et al. Characterization of a putative pathogenicity island from bovine Staphylococcus aureus encoding multiple superantigens[J]. Journal of Bacteriology, 2001, 183(1): 63-70. DOI:10.1128/JB.183.1.63-70.2001.
[ 45] NOVICK R P, SUBEDI A. The SaPIs: mobile pathogenicity islands of Staphylococcus[J]. Chemical Immunology and Allergy, 2007, 93: 42-57. DOI:10.1159/000100857.
[46] ROSEC J P, GIGAUD O. Staphylococcal enterotoxin genes of classical and new t ypes detected by PCR in France[J]. International Journal of Food Microbiology, 2002, 77(1/2): 61-70. DOI:10.1016/ S0168-1605(02)00044-2.
[47] SMYTH D S, HARTIGAN P J, MEANEY W J, et al. Superantigen genes encoded by the egc cluster and SaPIbov are predominant among Staphylococcus aureus isolates from cows, goats, sheep, rabbits and poultry[J]. Journal of Medical Microbiology, 2005, 54(4): 401-411. DOI:10.1099/jmm.0.45863-0.
[48] COLLERY M M, SM YTH D, TWOHIG J M, et al. Molecular typing o f nasal carriage isolates of Staphylococcus aureus from an Irish university student population based on toxin gene PCR, agr locus types and mult iple locus, variable number tandem repeat analysis[J]. Journal of Medical Microbiology, 2008, 57(3): 348-358. DOI:10.1099/ jmm.0.47734-0.
[49] MUNSON S H, TREMAINE M T, BETLEY M J, et al. Identification and characterization of staphylococcal enterotoxin types G and I from Staphylococcus aureus[J]. Infection and Immunity, 1998, 66 (7): 3337-3348.
[50] CHEN T R, CHIOU C S, TSEN H Y. Use of novel PCR primers specific to the genes of staphylococcal enterotoxin G, H, I for th e survey of Staphylococcus aureus strains isolated from foodpoisoning cases and food samples in Taiwan[J]. International Journal of Food Microbiology, 2004, 92(2): 189-197. DOI:10.1016/ j.ijfoodmicro.2003.10.002.
[51] NOTO M J, ARCHER G L. A subset of Staphylococcus aureus strains harboring staphylococcal casse tte chromosome mec (SCCmec) type IV is deficient in CcrAB-mediated SCCmec excision[J]. Antimicrobial Agents and Chemotherapy, 2006, 50(8): 2782-2788. DOI:10.1128/ AAC.00032-06.
[52] ALIBAYOV B, BABA-MOUSSA L, SINA H, et al. Staphylococcus aureus mobile genetic elements[J]. Molecu lar Biology Reports, 2014, 41(8): 5005-5018. DOI:10.1007/s11033-014-3367-3.
[53] RAM G, CHEN J, KUMAR K, et al. Staphylococcal pathogen icity island interference with helper phage reproduction is a paradigm of molecular parasitism[J]. Proceedings of the National Academy of Sciences, 2012, 109(40): 16300-16305. DOI:10.1073/ pnas.1204615109.
[54] POCSFAL VI G, CACACE G, CUCCURULLO M, et al. Proteomic analysis of exoproteins expressed by enterotoxigenic Staphylococcus aureus strain[J]. Proteomics, 2008, 8(12): 2462-2476. DOI:10.1002/ pmic.200700965.
[55] TSENG C W, STEWART G C. Rot repression of enterotoxin B expression in Staphylococcus aure us[J]. Journal of Bacteriology, 2005, 187(15): 5301-5309. DOI:10.1128/JB.187.15.5301-5309.2005.
[56] TSENG C W, ZHANG S, STEWART G C. Accessory gene regulator control of staphyloccoccal enterotoxin D gene expression[J]. Journal of Bacteriology, 2004, 186(6): 1793-1801. DOI:10.1128/JB.186.6.1793-1801.2004.
[57] KUSCH K, HANKE K, HOLTFRETER S, e t al. The influence of SaeRS and sigma(B) on the expression of superantigens in different Staphylococcus aureus isolates[J]. International Journal of Medical Microbiology, 2011, 301(6): 488-499. DOI:10.1016/ j. ijmm.2011.01.003.
Progress in Staphylococcal Enterotoxins and the Mobile Genetic Elements Encoding Them
WANG Qiong, TANG Junni*
(College of Life Science and Technology, Southwest University for Nationalities, Chengdu 610041, China)
Staphylococcal enterotoxins (SEs) belong to the family of pyrogenic toxin superantigens. The consumption of foods containing SEs could cause food poisoning with the symptoms of nausea, vomiting, abdominal cramping, and diarrhea. This review summarizes the clas sification and physicochemical properties of different SEs, especially focusing on the mobile genetic elements encoding SEs, which would have a theoretical and practical significance to epidemiological investi gation and understanding of the virulence mechanism of Staphylococcus aureus enterotoxins.
Staphylococcus aureus; staphylococ c al enterotoxins (SEs); phy sicochemical properties; mobile genetic elements
10.7506/spkx1002-66 30-20160342
TS207.4
A
1002-6630(2016)03-0241-06
王琼, 唐俊妮. 金黄色葡萄球菌肠毒素及移动基因元件研究进展[J]. 食品科学, 2016, 37(3): 241-246. DOI:10.7506/ spkx1002-6630-201603042. http://www.spkx.net.cn
WANG Qiong, TANG Junni. Progress in staphylococcal enterotoxins and the mobile genetic elements encoding them[J]. Food Science, 2016, 37(3): 241-246. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201603042. http://www.spkx.net.cn
2015-03-16
国家自然科学基金 面上项目(31371781);四川省科技 厅 应用基础研究项目(14JC0702);教育部“新世纪人才支持计划”项目(NCET-11-0847)
王琼(1990—),女,硕士研究生,研究方向为畜产品加工与安全。E-mail:wangqiong528@163.com
*通信作者:唐俊妮(1971—),女,教授,博士,研究方向为食品安全与食品微生物。E-mail: junneytang@aliyun.com
