基于CyClus3D聚类算法的PPI网络模体研究
2015-04-18浦恩禄张孟娇张俊鹏
浦恩禄,张孟娇,张俊鹏
(大理大学工程学院,云南大理 671003)
随着生物信息学技术的发展,人们已经获得了大量生物分子之间的相互作用数据,并由此构成了庞大的生物网络,其中蛋白质相互作用网络是这些生物网络数据中很重要的一类数据。蛋白质相互作用(Protein-Protein Interaction,PPI)在生物体的生命活动中起核心作用,对PPI的研究将有利于更好地了解生命体中的生物过程〔1〕。
PPI网络的基本构造单元是网络模体。网络模体是一些在真实网络中重复出现的具有特定拓扑结构特征的小规模模式单元,它在真实网络中出现的频率要远高于随机网络〔2〕。网络模体挖掘有利于揭示复杂网络的更多细节和内容,将有助于深入理解生物体的内在作用机制。
1 PPI网络模体挖掘
网络模体是一些具有特定性质的小规模子图,是构建整个生物网络的基本单元,网络模体的识别对PPI组成的生物基础大网络的研究和探究具有非常重要的作用。
1.1 PPI数据源 大量的PPI数据构建了很多内容丰富并且日益更新的PPI数据库,这些数据库为进一步深入研究PPI网络提供了数据支持。对PPI关系的认识和研究可以更好地认识细胞生命活动的过程,揭示生物体的生理和病理现象。
目前,每个PPI数据库都有自己独有的特性,并且数据库之间的PPI重合率较低。由于PPI数据库的选择没有统一的标准,本文根据PPI数据库在其他文献中使用的频繁程度,选择 HPRD〔3〕,HIR〔4〕和BioGRID〔5〕3个数据库作为PPI数据源。3个数据库之间的PPI重合数只有7 631,整合个数为222 556,见图1。为了更加全面地分析PPI网络,本文整合3种PPI数据库作为PPI数据源,其PPI数据源大小为222 556。
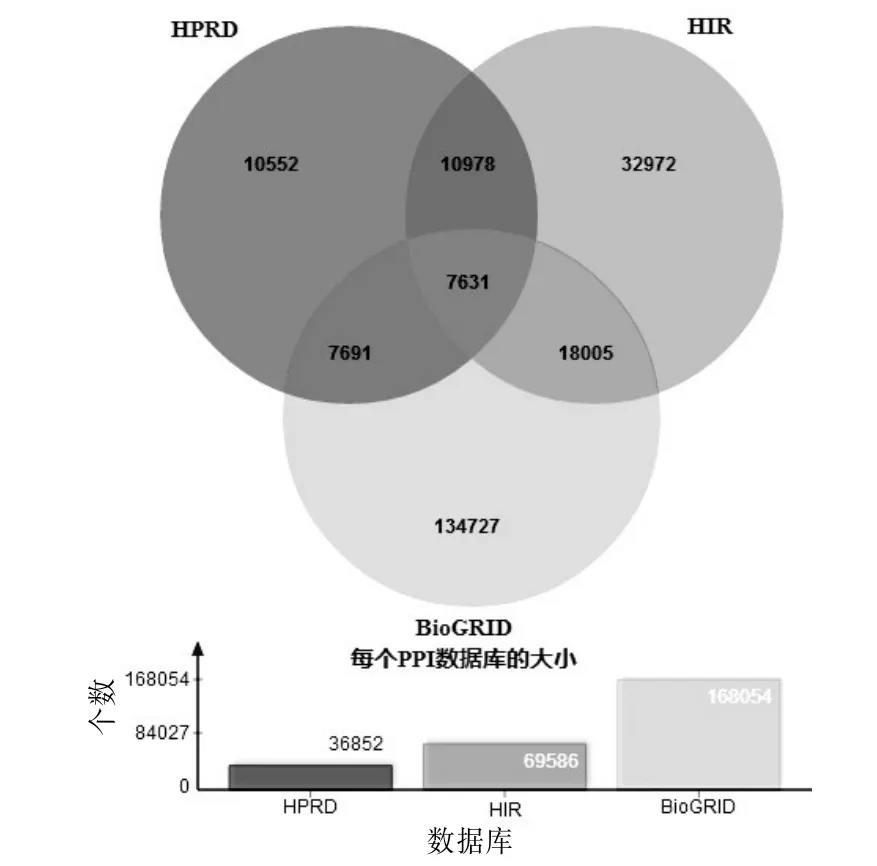
图1 HPRD、HIR和BioGRID 3种PPI数据库之间的维恩图
1.2 CyClus3D聚类算法 CyClus3D〔6〕是一种3-点网络模体挖掘算法。对于给定的3-点网络模体,其边可能是任何一种类型。假设所有模体的列表用一个3维数组T表示,如果节点(i,j,k)之间存在一个模体,那么Ti,j,k=1,否则Ti,j,k=0。由 3 个节点集(X1,X2,X3)组成的一个模体聚类可以用聚集分数来定义:
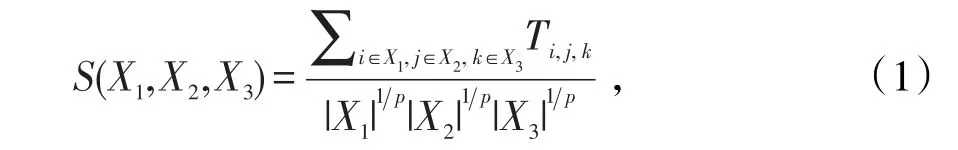
其中,|Xi|是Xi中的节点个数,如果p>1,它将扮演反精度参数的角色。为了最大化S,首先要确定T的最优秩-1逼近,即找到实数向量(x1,x2,x3)使下式最大化:
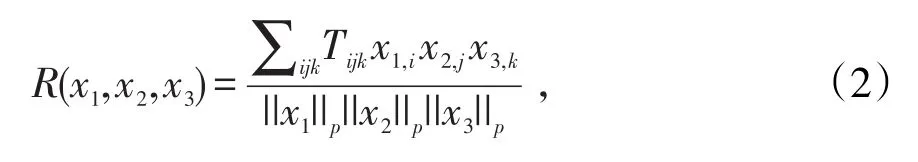

其中约束条件为||x1||p=1。
为了找到一个高分模体聚类,CyClus3D从模体集T中反复删除不存在的模体直至没有冗余的模体存在。由于模体数组的最优秩-1逼近等同于一个网络邻接矩阵的特征向量,因此CyClus3D聚类算法可以理解为二维频谱聚类算法〔7〕的泛化。
1.3 基于3-点网络模体的子网络 精度参数r=1/p,其值越小,高分模体聚类将大而稀疏。相反,其值越大,高分模体聚类将小而密集。为了折中处理,r设定为0.5。由于蛋白质相互作用关系为无向边,所使用的3-点模体边都为无向边。
CyClus3D聚类算法的3-点网络模体,见图2(a)。使用该模体挖掘的PPI子网络图,见图2(b),其节点数为374,边数为14 076。子网络的聚类效果较好(聚类系数=0.510),蛋白质与蛋白质之间的路径长度比较短(特征路径长度=1.835),并且子网络密度小而集中(网络密度=0.165,网络集中度=0.837),网络紧密程度高(网络直径=3),见表1。另外,同一家族的蛋白质往往有更多的相互作用,例如蛋白质组(RPL10,RPL11,…,RPL19),(PSMA1,PSMA2,…,PSMA8),(HDAC1,HDAC2,…,HDAC7)。
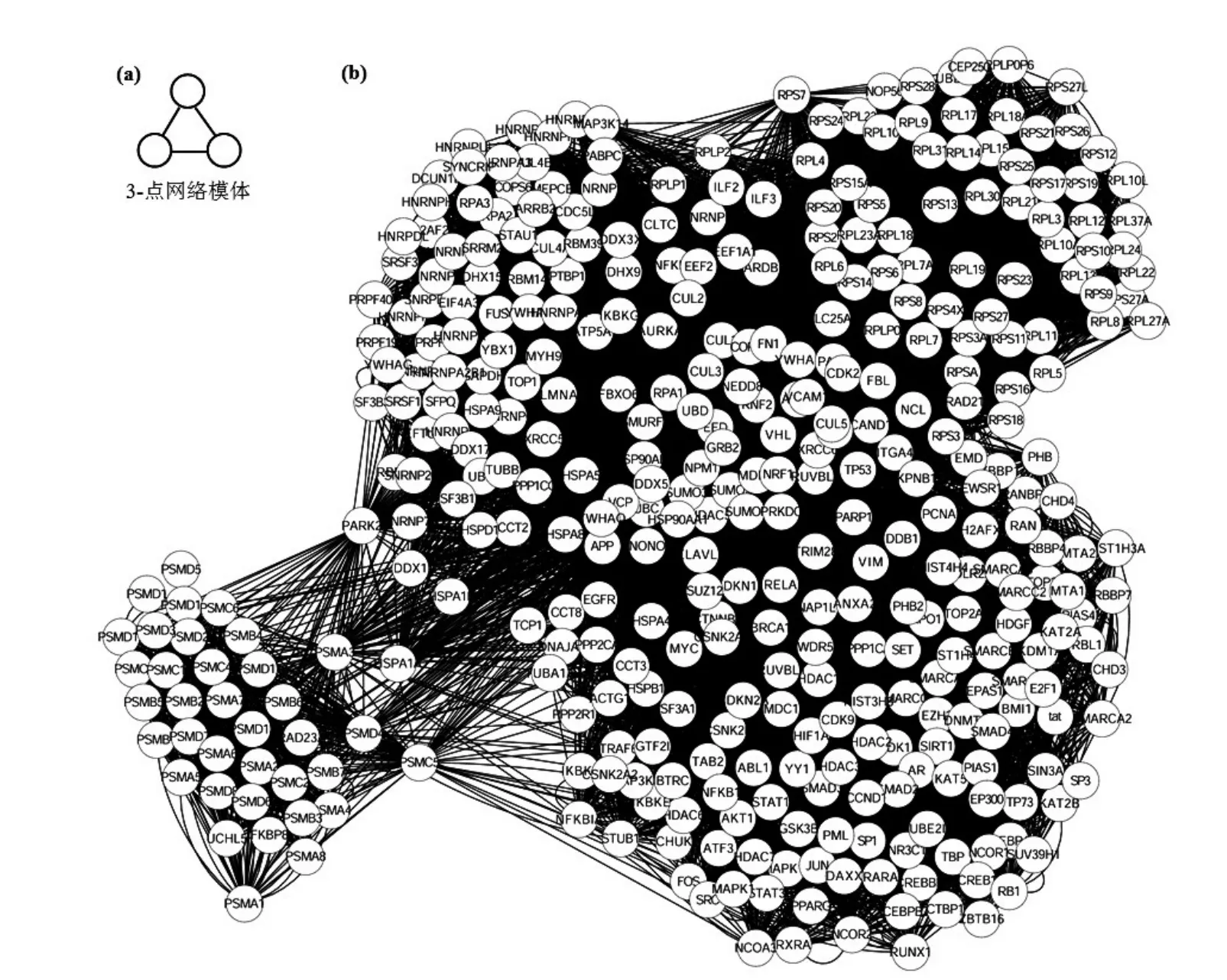
图2 CyClus3D聚类算法的3-点网络模体及其子网络图
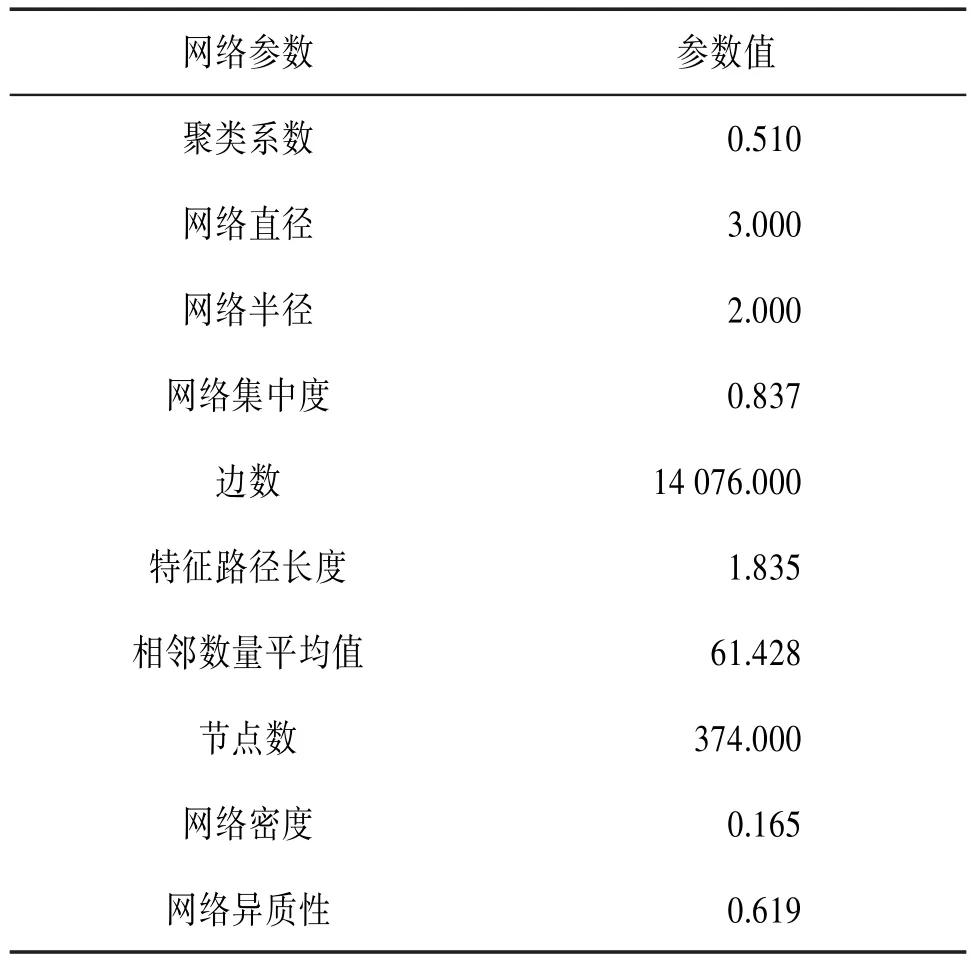
表1 基于3-点网络模体挖掘的子网络参数
2 网络富集分析
为了研究网络中的蛋白质与哪些生物过程和信号通道显著性相关,功能分析软件DAVID(http://david.abcc.ncifcrf.gov/)〔8〕用来对模体网络进行富集分析。DAVID是一个能为大规模的基因或蛋白质列表提供系统综合的生物功能注释信息的在线数据库。本文将从 GO(Gene Ontology)〔9〕生物过程和KEGG(Kyoto Encyclopedia of Genes and Genomes)〔10〕信号通道两个方面对模体网络进行富集分析。
2.1 GO生物过程富集分析 GO有3个结构化的网络,分别从生物过程、分子功能、细胞组成这3个方面对基因进行分类、定义和注释,本文重点挖掘蛋白质显著性相关的GO生物过程。
GO生物过程富集分析针对提交蛋白质列表采用超几何分布检测每一个GO生物过程条目的富集显著性。前5个GO生物过程如表2所示,其中3-点网络模体组成的子网络中有65个蛋白质(RPL18,RPL17,RPL19等)与生物过程:翻译延伸(GO:0006414~翻译延伸)显著性相关(p=3.65E-73)。
2.2 KEGG信号通道富集分析 KEGG信号通道富集分析针对提交的蛋白质列表采用超几何分布检测每一个KEGG信号通道的富集显著性,并进行KEGG信号通道图分析。前5个KEGG信号通道如表2所示,其中有53个蛋白质(E2F1,HSP90AB1,PPARG等)与癌症信号通道(KEGG:hsa05200~Pathways in cancer)显著性相关(p=7.88E-13)。
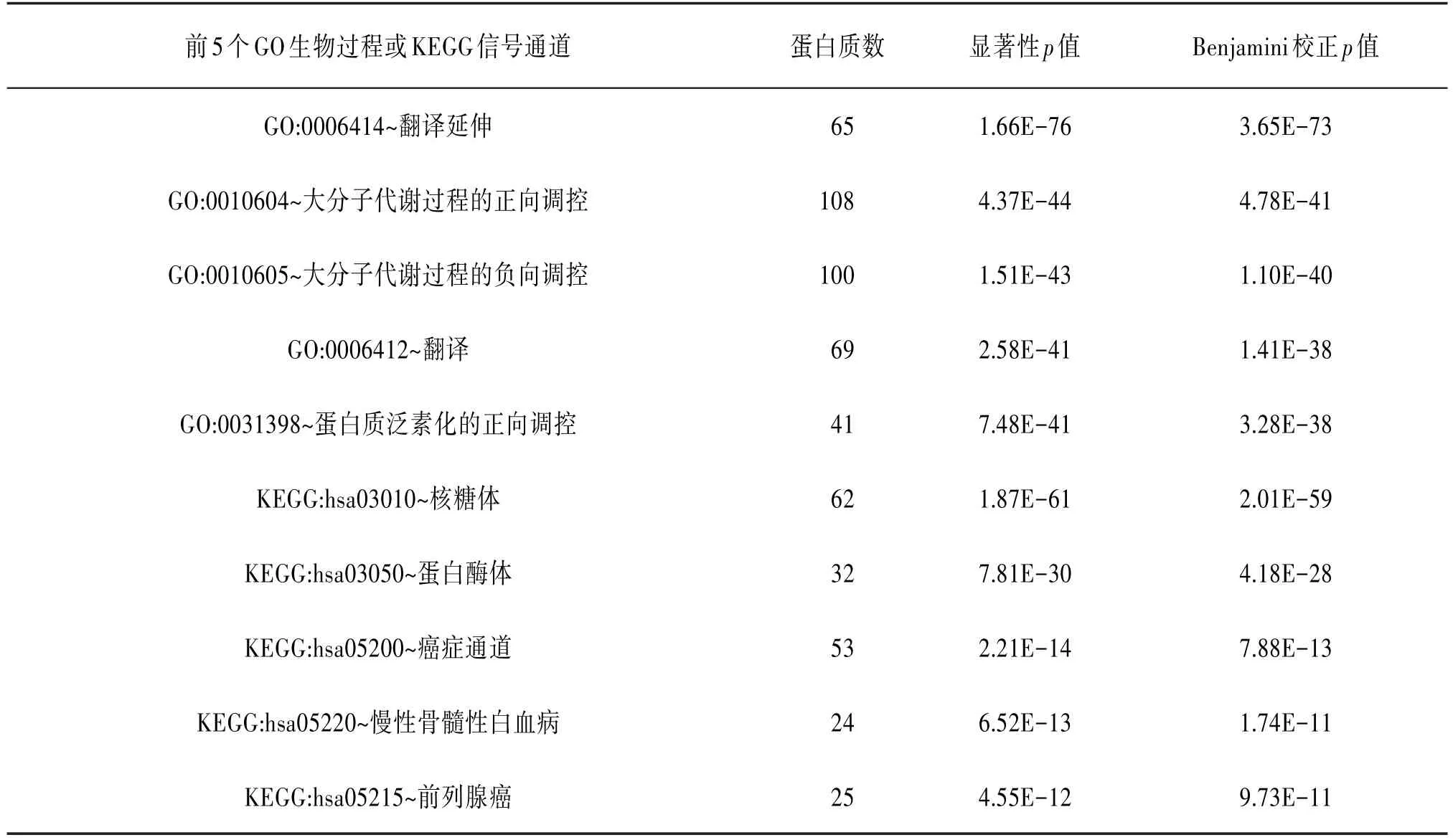
表2 前5个显著性GO生物过程和KEGG信号通道
3 结语
CyClus3D聚类算法对PPI网络进行模体挖掘,可以将222 556个PPI缩小到14 076个PPI来进行研究。这将大大简化PPI网络的分析,便于进一步研究蛋白质的相互关系。在线软件DAVID对网络模体组成的子网络进行GO生物过程和KEGG信号通道富集分析,能够挖掘出与蛋白质显著性相关的GO生物过程和KEGG信号通道。总之,PPI网络属于大型的复杂网络,从大量的PPI数据中挖掘出更多具有生物学意义的信息,有利于更好地了解生物体的生命活动中蛋白质之间的相互作用关系,从整体水平上理解某些疾病的细胞机制,预测一些蛋白质可能的生物功能。
〔1〕WAUGH D F.Protein-protein interactions〔J〕.Advances in Protein Chemistry,1954,9:325-437.
〔2〕SHEN-ORR S S,MILO R,MANGAN S,et al.Network motifs in the transcriptional regulation network of Escherichia coli〔J〕.Nature Genetics,2002,31(1):64-68.
〔3〕PRASAD T S K,GOEL R,KANDASAMY K,et al.Human protein reference database-2009 update〔J〕.Nucleic Acids Research,2009,37(S1):D767-D772.
〔4〕ZHOU X,CHEN P,WEI Q,et al.Human interactome resource and gene set linkage analysis for the functional interpretation of biologically meaningful gene sets〔J〕.Bioinformatics,2013,29(16):2024-2031.
〔5〕STARK C,BREITKREUTZ B J,REGULY T,et al.BioGRID:a general repository for interaction datasets〔J〕.Nucleic Acids Research,2006,34(S1):D535-D539.
〔6〕AUDENAERT P,VAN P T,BRONDEL F,et al.CyClus3D:a Cytoscape plugin for clustering network motifs in integrated networks〔J〕.Bioinformatics,2011,27(11):1587-1588.
〔7〕INOUE K,URAHAMA K.Sequential fuzzy cluster extraction by a graph spectral method〔J〕.Pattern Recognition Letters,1999,20(7):699-705.
〔8〕HUANG D W,SHERMAN B T,LEMPICKI R A.Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources〔J〕.Nature Protocols,2009,4(1):44-57.
〔9〕ASHBURNER M,BALL C A,BLAKE J A,et al.Gene Ontology:tool for the unification of biology〔J〕.Nature Genetics,2000,25(1):25-29.
〔10〕KANEHISA M,GOTO S.KEGG:kyoto encyclopedia of genes and genomes〔J〕.Nucleic Acids Rresearch,2000,28(1):27-30.
