核糖核酸开关用于微生物细胞工厂的智能与精细调控
2015-02-14赵雨佳张根林周晓宏李春
赵雨佳,张根林,周晓宏,李春
(北京理工大学生命学院生物工程系,北京 100081)
核糖核酸开关用于微生物细胞工厂的智能与精细调控
赵雨佳,张根林,周晓宏,李春
(北京理工大学生命学院生物工程系,北京 100081)
利用代谢工程与合成生物技术对细胞内复杂的代谢网络和调控网络进行重构和改造,以建立合成新化合物或提高目标产物产量的微生物细胞工厂是当今绿色化工技术发展的方向之一。微生物代谢途径的调控受环境和遗传的双重影响,细胞通过全局转录因子、信使分子和反馈抑制等方式响应环境变化来维持细胞的内稳态;同时细胞还受自身遗传基因线路的调控,在转录、翻译以及翻译后修饰过程中调控特定基因的表达。核糖核酸开关是一类调控基因线路表达的RNA元件,通过与金属离子、糖类衍生物、氨基酸、核酸衍生物以及辅酶等特异性配体结合发生的构象变化,从而启动或阻断mRNA的转录、翻译、拼接等过程来调控基因的表达。核糖核酸开关作为天然的生物感受器和效应器通过人工设计可成为微生物细胞工厂智能化和精细化调控的分子工具,并在化工、医药、环保、食品等领域得到广泛应用。
核糖核酸开关;微生物细胞工厂;精细调控;生物转化;优化;代谢工程;合成生物学
Key words:riboswitch;microbial cell factory;fine regulation;biotransformation;optimization;metabolic engineering;synthetic biology
引 言
微生物由于其种属多样性和遗传多样性,在38亿年的进化历程中,形成了复杂的代谢途径,可以生产包括大宗化学品、医药、燃料、塑料在内的多种重要的医药和化工产品,现已利用谷氨酸棒状杆菌成功生产丝氨酸[1]、赖氨酸[2]、谷氨酸[3]以及丁二酸[4]等大宗化学品;利用酿酒酵母生产青蒿素[5-6]、紫杉醇[7]、丹参酮[8]等多种名贵药物;通过蓝细菌生产乙醇[9]、正丁醇[10]等生物燃料以及通过大肠杆菌生产聚羟基脂肪酸酯(polyhydroxyalkanoate,PHA)[11]、聚羟基丁酸酯(polyhydroxybutyrate, PHB)[12]等可降解的生物塑料。利用微生物生产化工产品为解决能源危机和环境污染提供了新思路。然而天然的微生物因为产量低或者底物谱窄等原因往往不能满足工业化生产的需要,因此,需要人为地对其代谢途径进行改造,通过重组和优化,使微生物细胞内的代谢流和能量流重新分配,成为服务于工业需求的微生物细胞工厂。
利用微生物细胞工厂生产医药和化工产品具有清洁、高效、经济附加值高等优点,已成为发展绿色工业的核心部分。然而长期的研究发现,微生物细胞暴露在环境中会受到来自外界和内部的多种信号刺激,为了维持细胞的内稳态,微生物进化出了复杂的调控网络来感受信号,进而调控相应的代谢途径。因此单纯的引入外源代谢途径或者酶往往会导致代谢失衡,不能得到目标产物或者产量难以满足工业需求。为了得到能满足工业需求的微生物细胞工厂不仅要对代谢途径进行改造还要对相应的调控系统进行修饰。但由于其调控的复杂性、人们缺乏在分子水平上对细胞调控机制的了解,难以开发智能的表达调控工具,严重制约了微生物细胞工厂的发展。
细胞的调控网络受环境和遗传的双重影响。由于微生物细胞生活在不断变化的环境中,温度、pH、营养等环境因素都会对细胞的生理代谢产生影响,细胞通过各种全局转录因子以及信使分子感受环境因素的变化从而调控一系列生理过程并通过产物对代谢途径的反馈抑制等方式使细胞适应环境变化,维持内稳态。同时,细胞可以通过转录、翻译以及翻译后修饰等方式调控特定基因的表达。核糖核酸开关是细胞内天然存在的表达调控工具,能够响应单磷酸腺苷环二聚体(c-di-AMP)、单磷酸鸟苷环二聚体(c-di-GMP)等第二信使进而调控微生物细胞产芽孢、维持渗透压等多种生理过程,还能感受细胞内氨基酸的浓度,在浓度较高时通过使转录提前终止或抑制翻译起始的方式对相应的代谢途径进行反馈抑制[13-15],自2002年首次发现就引起了轰动。作为调控基因表达的RNA元件,核糖核酸开关由两部分组成,适体区(aptamer domain,AD)和基因表达区(expression domain,ED),其中AD能识别特异性的小分子代谢物或金属离子,与之结合后使ED变构,从而能够在转录水平或翻译水平上调控相关基因的表达(图1)[16]。由于核糖核酸开关不仅能感受信号还能使细胞针对信号变化做出相应的反应,并且不需要蛋白质的参与,具有结构简单、反应迅速等优点。自发现其原理以来,人们通过模拟天然核糖核酸开关的调控机理设计出一系列人工核糖核酸开关,并将这些核糖核酸开关成功应用于调控细菌的趋化性、构建电子基因线路、开发生物传感器等方面,近年来也致力于将其作为微生物细胞工厂的智能化精细调控工具而开发应用。因此本文将对核糖核酸开关的作用机制及其应用前景进行综述。

图1 核糖核酸开关的结构Fig.1 Architecture of riboswitch
1 核糖核酸开关的种类及作用机制
随着对核糖核酸开关研究的逐步深入,现已发现至少有20种天然的核糖核酸开关(表1)。这些天然的核糖核酸开关可以按照配体的不同分为5类,分别是响应离子、糖衍生物、氨基酸、核酸衍生物以及响应辅酶的核糖核酸开关,下面将对这些天然的核糖核酸开关的种类及作用机制进行简要综述。
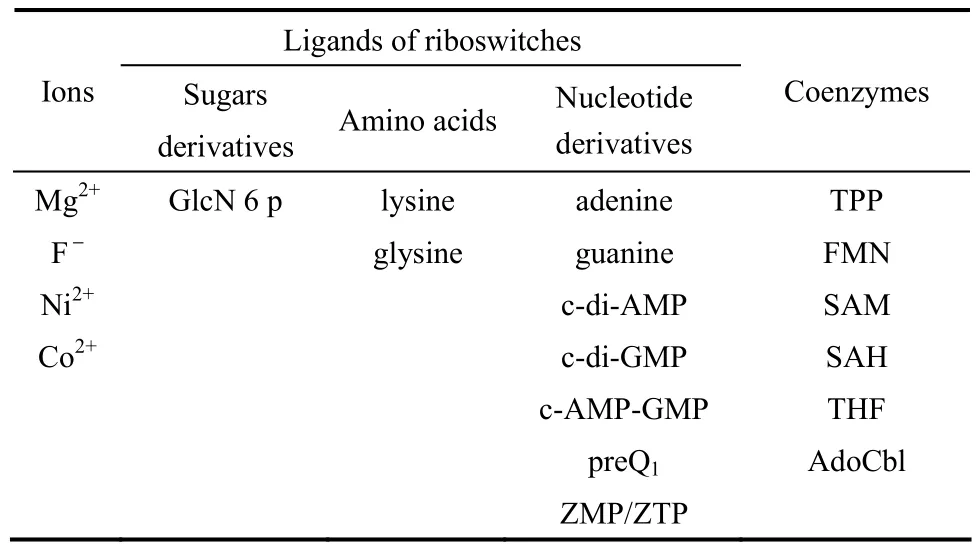
表1 核糖核酸开关的种类Table 1 Class of riboswitches
1.1 离子配体的核糖核酸开关
微生物生活在包含各种离子的环境中,这些离子参与生物体内很多重要的催化反应,如Mg2+是DNA聚合酶的辅酶,如果缺少Mg2+,DNA聚合酶的效率会显著降低;但是离子过多则会对细胞造成毒害,使细胞死亡。因此维持细胞内的离子内稳态至关重要。早先的研究表明微生物主要通过蛋白质来调控离子转运蛋白的表达从而维持细胞内的离子内稳态。近年来,许多研究表明一些位于离子转运蛋白5′-UTR的核糖核酸开关也能感受离子浓度并通过变构效应调控离子转运蛋白的表达。
迄今为止,发现的能识别离子的核糖核酸开关有Mg2+核糖核酸开关[17-18]、F−核糖核酸开关[19]、Ni2+、Co2+核糖核酸开关[20]。这些识别离子的核糖核酸开关能够感受细胞内离子的浓度,高浓度状态下核糖核酸开关打开,使下游编码的转运蛋白的基因表达;低浓度状态下核糖核酸开关关闭,抑制下游转运蛋白的表达[图2(a)],从而维持细胞内离子的内稳态,维持细胞正常生理过程。
1.2 糖衍生物配体的核糖核酸开关
目前只发现一种能识别葡糖衍生物——葡糖胺-6-磷酸(glucosamine-6-phosphate,GlcN6p)的核糖核酸开关,这种核糖核酸开关被命名为glmS。glmS主要存在于各种革兰阳性菌中,其中很多都是人类的病原菌。
glmS是现已发现的天然核糖核酸开关中一种非常独特的核糖核酸开关,其作用机制不同于一般的调控转录的提前终止或者是抑制核糖体与核糖体结合位点(ribosome binding site,RBS)的结合。glmS是一种自剪切核酶,其位于基因glmS(编码谷氨酰胺-果糖-6-磷酸酰胺转移酶,可利用果糖-6-磷酸和谷氨肽合成GlcN6p参与糖代谢和细胞壁的生物合成)的5′-UTR,通过变构效应对GlcN6p的合成起反馈抑制作用[21]。当细胞内GlcN6p浓度较高时,GlcN6p与glmS结合,glmS的酶活增强,降解谷氨酰胺-果糖-6-磷酸酰胺转移酶的前导mRNA,减少GlcN6p的合成量[图2(b)]。GlcN6p是细胞壁成分N-乙酰葡糖胺的前体物质,GlcN6p量的减少会导致细胞壁合成障碍,因此可以利用glmS作为开发新药的靶位点,用来抵抗细菌的多重耐药性机制,研制新型抗生素[22]。

图2 天然核糖核酸开关调控机制Fig.2 Regulatory mechanism of natural riboswitches
1.3 氨基酸配体的核糖核酸开关
现已发现两种受核糖核酸开关调控表达的氨基酸,分别是甘氨酸和赖氨酸。
甘氨酸核糖核酸开关的结构非常特别,它是发现的天然核糖核酸开关中唯一一个串联了两个识别同一配体的AD和一个ED构成的核糖核酸开关。并且两个AD表现出与甘氨酸结合的协同性,推测这种机制能够使核糖核酸开关对配体的亲和性变强,提高调控系统的灵活性[23]。当细胞内甘氨酸浓度较高时,甘氨酸与AD结合,使gcvT基因的5′-UTR区原本存在的前终止子结构被破坏,转录出完整的mRNA,使gcvT基因表达[图2(c)]。因此,甘氨酸核糖核酸开关还是一种罕见的与配体结合后使下游基因表达的“ON”开关。
赖氨酸核糖核酸开关通常位于基因lysC的5′-UTR区,由能识别赖氨酸的AD和包含有RNAaseE识别位点的ED组成。当细胞内赖氨酸浓度高时,赖氨酸与AD结合促使ED变构暴露出RNAaseE识别位点,从而降解新生的mRNA[图2(d)],实现对赖氨酸的反馈抑制[24-25]。
1.4 核酸衍生物配体的核糖核酸开关
c-di-GMP、c-di-AMP和7-氨甲基-7-去氮杂鸟嘌呤(7-aminomethyl-7-deazaguanine,preQ1)等核酸衍生物均有相应的核糖核酸开关,通过与核糖核酸开关结合后的变构效应来调控基因的表达。以c-di-GMP核糖核酸开关为例介绍其作用机制。
c-di-GMP是细胞内最主要的第二信使分子,能调控多种重要的生理过程,如调控病原菌的毒力、鞭毛的运动以及生物膜的形成等[26]。2008年耶鲁大学的Breaker课题组首次通过比较基因组学发现多种微生物基因组中普遍存在保守的GEMM motif,GEMM motif能识别c-di-GMP并能影响相关下游基因的表达。经过实验验证GEMM是一类能识别c-di-GMP的核糖核酸开关[27]。研究发现,病原菌梭状芽胞杆菌(Clostridium difficile)中调控其毒力基因的5′-UTR区存在一类c-di-GMP核糖核酸开关,这类核糖核酸开关是一种自剪切核酶,被Breaker命名为I型自剪切核酶(Group I self-splicing ribozyme)。这类核酶通过与c-di-GMP的结合暴露或者覆盖住其自身的切割位点,从而阻止或者开始下游基因的表达[28]。
1.5 辅酶配体的核糖核酸开关
硫胺素焦磷酸(thiamine pyrophosphate,TPP)、黄素单核苷酸(flavin monoucleotide , FMN)、腺苷蛋氨酸(S-adenosyl methionine,SAM)等都是重要的辅酶,分别参与细胞内的脱羧反应、氧化还原反应以及甲基转移过程,对于维持细胞的内稳态至关重要。通过比较基因组学分析发现,多种细菌基因组中普遍存在保守的响应这些辅酶的核糖核酸开关,与相应的辅酶结合后发生变构从转录水平或翻译水平调控相关基因的表达。基于这些辅酶的重要生理作用和独特的调控机制,可以认为这些核糖核酸开关是RNA世界的遗迹。
2 人工核糖核酸开关的设计
天然的核糖核酸开关可以同时作为生物感受器和效应器——感受细胞内小分子代谢物或金属离子的浓度并根据相应的浓度调控相关基因的表达从而使细胞维持内稳态以适应周围的环境。核糖核酸开关的这种特性,可用于开发智能化的精细调控工具,优化细胞工厂的调控网络。然而天然核糖核酸开关的配体多为细胞的初级代谢产物,存在调控的背景干扰过强的问题,因此设计识别细胞内原本不存在的配体的人工核糖核酸开关将更有利于实现细胞工厂的智能化调控。
2.1 基于控制转录提前终止原理设计核糖核酸开关
某些基因的5′-UTR存在一些不依赖于Rho的终止子结构,天然的核糖核酸开关AD与配体结合后会使ED变构导致ED的部分序列与终止子序列形成W-C配对,从而破坏5′-UTR天然的终止子结构,使基因能够转录。基于这一原理,Mörl课题组设计了一系列在转录水平调控基因表达的人工核糖核酸开关[29]。他们的设计策略是在茶碱适体的3′端和终止子结构的聚尿嘧啶尾巴之间随机插入不同长度的核苷酸,利用软件RNA fold模拟插入序列能否与聚尿嘧啶尾巴形成类似终止子的茎环结构,再通过报告基因的颜色变化进行筛选,最后得到一个人工设计的能阻止转录提前终止的核糖核酸开关。当没有茶碱时,ED形成终止子结构转录不能进行,添加茶碱后,茶碱与AD结合,终止子结构被破坏,转录开始,目标基因表达[图3(a)]。
2.2 基于控制RBS与核糖体结合原理设计核糖核酸开关
通过控制RBS与核糖体的结合来调控翻译的起始,是天然核糖核酸开关最常用的两种调控策略之一,天然的核糖核酸开关AD与配体结合后会导致RBS被遮盖或者暴露,从而抑制或起始目标基因的翻译过程。利用这一机制,Gallivan等[30]设计了一系列人工核糖核酸开关。其设计思想是在茶碱适体和RBS之间随机插入不同长度的核苷酸,通过报告基因cat对四环素的抗性筛选,分别得到响应茶碱后表达cat基因的“ON”核糖核酸开关和响应茶碱后抑制cat基因表达的“OFF”核糖核酸开关[图3(b)]。
2.3 基于控制mRNA提前降解原理设计核糖核酸开关
有些天然核糖核酸开关本身是自剪切核酶,如GlcN6p核糖核酸开关,与配体结合后暴露出酶切位点,降解目标基因的mRNA。基于这一原理,耶鲁大学的Breaker课题组将一个ATP适体与锤头状核酶通过一段随机序列的交流模块连接起来,通过外加ATP,观察锤头状核酶能否自剪切,最终得到两种具有自剪切核酶功能的人工核糖核酸开关,一种加入ATP后使核酶自剪切,一种加入ATP后能够抑制核酶的自剪切[31][图3(c)]。
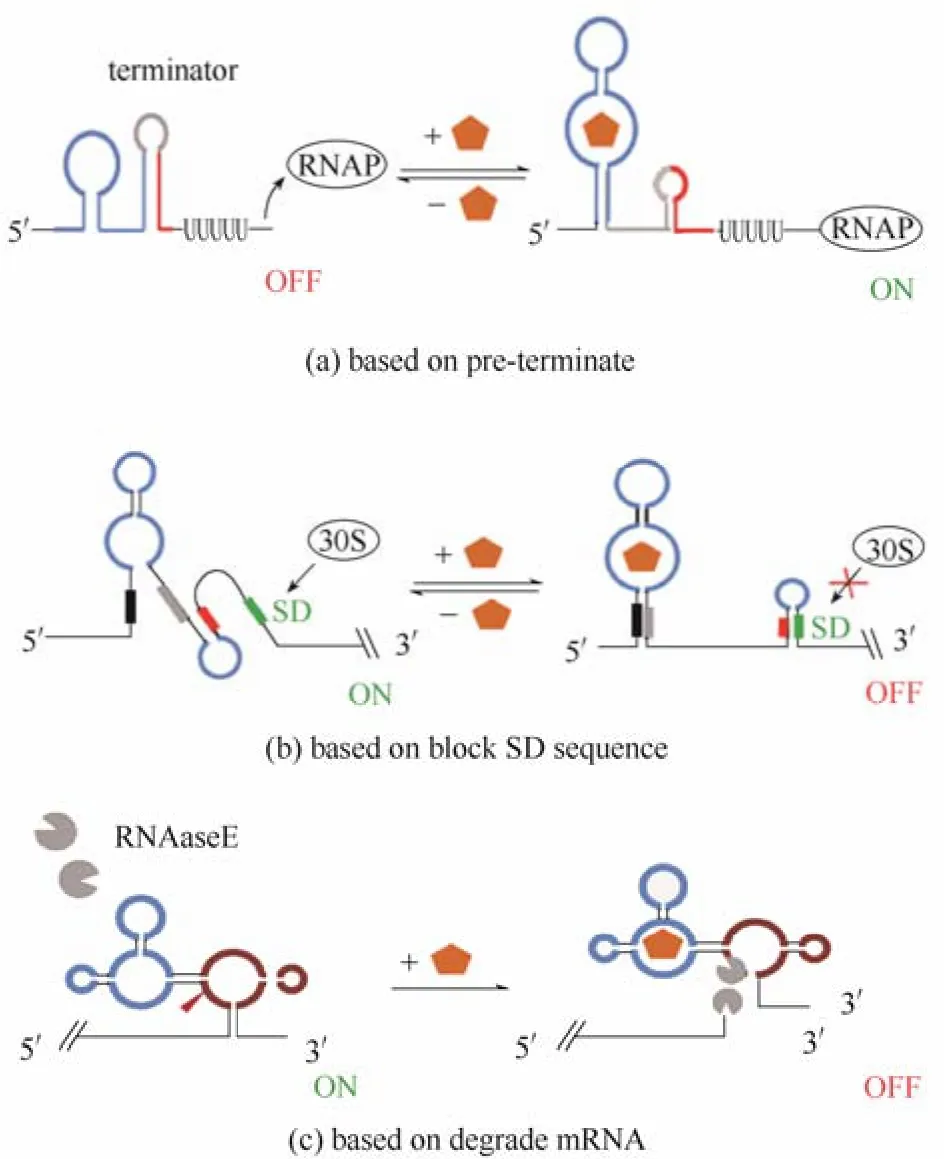
图3 人工核糖核酸开关设计原理Fig.3 Principle of design artificial riboswitch
3 核糖核酸开关在细胞工厂中的应用
3.1 应用于工业菌株的筛选和途径的精细调控
性状优良的菌株是发酵工业的灵魂,传统方法往往是对出发菌株进行物理或化学诱变,再通过对突变文库进行人工筛选来检验有没有理想的高产菌株。但这种方法对环境和实验人员的伤害较大,而且诱变不能针对某一特定性状,基因的随机突变导致突变文库较大,很难筛选到理想的工业菌株。天然核糖核酸开关的效应物大多是一些初级代谢产物,这些核糖核酸开关可以特异性地针对某种代谢产物在细胞内行使反馈调节的作用。因此可以利用核糖核酸开关对代谢途径进行精细调控,提高目标产物产量。例如Zeng课题组在生产赖氨酸的工业菌株谷氨酸棒状杆菌LP917(Corynebacterium glutamicumLP917)基因gltA(编码柠檬酸合酶)的5′-UTR区插入响应赖氨酸的核糖核酸开关,通过外加赖氨酸抑制柠檬酸合酶的转录,使菌体积累草酰乙酸。草酰乙酸作为合成赖氨酸的前体物质,它的积累能提高赖氨酸的产量,进一步抑制柠檬酸合酶的产生,形成一个正循环,提高工业菌株中赖氨酸的产量[32]。利用这种方法,他们成功使赖氨酸的产量提高了63%。
Jung课题组[33]采用另一种策略构建了包含赖氨酸核糖核酸开关和tetA基因[34](编码四环素/H+转运蛋白,少量表达能够抗四环素,大量表达会使细菌对Ni2+类金属离子敏感)的筛选系统。在培养基中添加NiCl2作为筛选压力,菌体内赖氨酸浓度高,抑制tetA基因的表达,使菌体对NiCl2不敏感,能够存活;而当菌体内赖氨酸浓度低,tetA基因表达,菌体对NiCl2敏感,死亡。因此经过几轮的筛选和富集,最终得到的就是高产赖氨酸的菌株。Jung课题组构建的这种基于核糖核酸开关来筛选高产菌株的平台,只要能找到响应效应物的核糖核酸开关就能定向地筛选到高产菌株,具有针对性强、适用范围广、操作简单等优点。
此外,工业发酵过程会产生大量的发酵热,不仅影响微生物的生长和生产,还因冷却控温增加了生产成本,因此发酵工业迫切需要耐热的底盘宿主。基于温度变构原理,人工设计响应不同温度的核糖核酸开关[35],使热激蛋白(heat shock protein,HSP)、超氧化物歧化酶(superoxide dismutase, SOD)等能提高微生物耐热性的耐热元件在不同温度下依次表达,不仅能提高微生物的耐热性还能减少能耗,保护环境。
综上所述,利用核糖核酸开关来构建的工业菌株筛选方法具有特异性强、灵活度高、对环境和实验人员危害小等优点,是一种简便高效的筛选工业菌株的方法,在发酵工业上有巨大的应用价值。
3.2 应用于新药开发
随着抗生素类药物的广泛使用,细菌的多耐药现象引起了人们的关注,近30年来研发的新型抗生素药物屈指可数,甚至出现了“抗生素悖论”效应。传统抗生素的作用机制仅限于阻碍核酸的合成、干扰蛋白质的合成、影响细胞膜和细胞壁的功能,因此亟需找到新型药物作用靶点,利用特异性的调控作用来抑制致病基因的表达。核糖核酸开关在细菌中普遍存在并参与细菌基本生命代谢的重要过程,将核糖核酸开关作为研发新药的作用靶点[36],大大拓展了传统抗生素的作用机制,为解决多重耐药性问题提供了崭新的思路。
氟化物在ATP或GTP结合二价金属离子的过程中能模拟ATP或GTP的γ磷酸基团[37],从而阻断很多酶的活性[38],对各种有机体都有毒性。病原菌中存在天然的响应氟离子的核糖核酸开关,当细胞内氟离子过多时,开关打开,表达氟离子转运蛋白,外排出过多的氟化物从而维持细胞内氟离子的内稳态。开发新药使氟离子更多地进入细胞,打破病原菌内的氟离子内稳态,则能有效杀死病原菌。基于这一思想,Breaker课题组在病原菌中构建了一个高效筛选系统,这个系统包含响应氟离子的核糖核酸开关和报告基因lacZ。将含有高效筛选系统的病原菌涂在含有X-gal和氟化物以及不同药物的培养基上,通过颜色的变化判断哪种新药能使更多的氟离子进入细胞,通过一系列实验发现短杆菌肽作用于细胞膜上的氟离子通道,使更多的氟离子进入细胞,从而杀死病原菌[39]。通过短杆菌肽辅助氟离子杀菌的方式能有效抑制口腔病原菌的生长,对治疗龋齿有良好的效果。
glmS核糖核酸开关与配体GlcN6p结合后抑制谷氨酰胺-果糖-6-磷酸酰胺转移酶的表达,导致产物GlcN6p的量减少,而GlcN6p是细菌细胞壁N-乙酰葡糖胺的前体,减少GlcN6p将抑制细菌细胞壁的合成。利用这一思路,Soukup课题组设计了一系列GlcN6p的结构类似物,其中有两个能有效与glmS核糖核酸开关结合发挥抑菌作用[40]。这两种GlcN6p的结构类似物可以作为新药作用于含有glmS核糖核酸开关的多种病原菌。
Gallivan课题组在大肠杆菌中构建了一个调控大肠杆菌趋化性[41]的系统,这个系统由茶碱核糖核酸开关和调控鞭毛旋转的cheZ基因组成。通过外源添加茶碱,使CheZ表达,控制细菌向茶碱方向移动[42]。利用这种改变细菌趋化性的策略,可以使携带药物工程菌向病灶聚集增强治病能力。
3.3 作为酶分子定向进化的高通量筛选方法
细胞工厂通过酶的催化作用生产各种满足人类需要的化合物,可以说酶的催化作用是工业发酵的核心。然而,许多天然酶具有与底物识别的专一性差、催化效率低以及热稳定性差等问题,需要对其进行改性才能满足工业生产的需求。
定向进化是一种有效的酶改性策略,可以通过易错PCR等方法对编码基因进行随机突变或重组,使其原有活性提高或者产生新的功能。由于随机突变后产生的文库较大,筛选难度高,定向进化成功的关键在于能否针对目标特性开发出高效的筛选方法。核糖核酸开关在微生物中普遍存在并且能响应特定的效应物调节目标基因的表达,因此可作为一个有效的筛选模块,为开发高效筛选方法提供崭新的思路。Smolke课题组将响应茶碱的适体与自剪切核酶融合后构建了能响应茶碱的核酶开关,当没有茶碱时该核糖核酸开关结构不稳定会降解目标基因的mRNA,抑制目标基因的表达;只有当茶碱与适体结合后核酶结构稳定,才能表达目标基因。将这个人工设计的核糖核酸开关构建到咖啡因脱甲基酶与绿色荧光蛋白(green fluorescent protein,GFP)融合的蛋白质的3′-UTR区,可以用于咖啡因脱甲基酶的定向进化。因为咖啡因脱甲基酶能够将咖啡因转化成茶碱,而茶碱是该核糖核酸开关的配体,通过荧光强度的不同利用流式细胞仪(fluorescence activated cell sorter,FACS)筛选到酶活提高了32倍的咖啡因脱甲基酶[43]。
3.4 构建新型生物传感器
生物传感器是将生物识别元件和信号转换元件紧密结合,从而检测目标化合物的分析装置。将核糖核酸开关与报告基因(beta-半乳糖苷酶、荧光蛋白、荧光素酶)串联在一起,通过报告基因的表达强度来判断相应的配体浓度,开发出各种新型生物传感器。这些新型生物传感器与传统的生物传感器相比,不需要借助细胞外的各种光学元件或者压电装置,可以直接将细胞内部某一代谢物的浓度信号转化为颜色变化等输出信号。具有结构简单、反应灵敏、价格低廉、生物安全性高等优点,可以广泛应用于食品药品的检测等方面。如Gu等[44]将天然的响应腺苷钴胺素(adenosyl-cobalamin, AdoCbl)的核糖核酸开关构建到GFP的5′-UTR区,通过荧光强度的变化来检测B12的浓度,利用这种方法对AdoCbl的检测限达到10 ng·ml−1,相当于高效液相色谱(high performance liquid chromatography,HPLC)的水平。
融合适体与核酶构建成的人工适体酶(aptazyme)能将与靶分子结合的信号稳定转换成更清晰的酶学信号,如自切割活性,通过在适体酶上标记放射性核素或荧光基团,能够检测到微量浓度的配体[45]。
2011年Jaffrey等[46]利用RNA成功模拟了绿色荧光蛋白,设计出一种名为Spinach的核糖核酸开关,结合底物DMHBI之后会发出绿色荧光。通过将Spinach与相应不同的适体融合,开发出能检测c-AMP- GMP[47]、腺苷酸、ADP、SAM、谷氨酰胺和GFP[48]的核糖核酸开关。综上所述基于核糖核酸开关开发的生物传感器是一种检测发酵工业中某种特定产物的有效手段。
4 结论与展望
自2002年Breaker首次提出核糖核酸开关这个术语[49],对于核糖核酸开关的研究已经整整13年,工程化的核糖核酸开关已经不再仅仅是一个构想,而被用于在各种工业微生物中构建具有鲁棒性和正交性的调控系统,利用不同的调控机制对细胞工厂进行智能调控。目前,工程化的核糖核酸开关已经成功应用于筛选工业菌株、研发新药、构建酶分子的定向进化的筛选方法以及开发新型生物传感器等方面。
与基因表达的蛋白调控系统比较,核糖核酸开关调控基因的表达不需要蛋白质的参与,具有免疫原性小、结构简单、反应迅速等优势。然而天然核糖核酸开关响应的效应物大多是细胞内的初级代谢产物,利用天然核糖核酸开关开发的细胞内调控系统存在背景干扰过强的问题。针对这一现象,可以以细胞内不存在的物质作为配体设计人工核糖核酸开关,能有效区分核糖核酸开关调控的基因是受细胞内源配体影响还是外加配体影响,解决细胞内调控背景干扰过强的问题。除此之外,核糖核酸开关的工业化应用还存在一个问题——难以筛选到合适的响应非细胞内天然产物的适体。虽然理论上任何一种物质都能在体外筛选到相应的适体,然而大量实验证明体外筛选到的适体大多不能在体内起作用,这可能是因为体内的环境与体外不同所导致的。而这一问题大大限制了核糖核酸开关的应用,因此,如何开发出更多的能在体内响应不同配体的适体是核糖核酸开关在工业化应用进程中的一大挑战。
符 号 说 明
AD——核糖核酸开关的适体区
AdoCbl——腺苷钴胺素
c-di-AMP——单磷酸腺苷环二聚体
c-di-GMP——单磷酸鸟苷环二聚体
FACS——流式细胞仪
FMN——黄素单核苷酸
GFP——绿色荧光蛋白
GlcN6p——葡糖胺-6-磷酸
HPLC——高效液相色谱
HSP——热激蛋白
ORF——开放阅读框
PHA——聚羟基脂肪酸酯
PHB——聚羟基丁酸酯
preQ1——7-氨甲基-7-去氮杂鸟嘌呤
RBS——核糖体结合位点
SAM——腺苷蛋氨酸
SOD——超氧化物歧化酶
TPP——硫胺素焦磷酸
UTR——非翻译区
[1] Lai Shujuan, Zhang Yun, Liu Shuwen, Liang Yong, Shang Xiuling, Chai Xin, Wen Tingyi. Metabolic engineering and flux analysis ofCorynebacterium glutamicumfor L-serine production [J].Science China Life Sciences, 2012, 55(4): 283-290.
[2] Wu Y, Li P, Zheng P, Zhou W, Chen N, Sun J. Complete genome sequence ofCorynebacterium glutamicumB253, a Chinese lysine-producing strain [J].J. Biotechnol., 2015, 4(207): 10-11.
[3] Wang N, Ni Y, Shi F. Deletion of odhA or pyc improves production of γ-aminobutyric acid and its precursor L-glutamate in recombinantCorynebacterium glutamicum[J].Biotechnol. Lett., 2015, 37(7): 1473-1481.
[4] Shi X, Chen Y, Ren H, Liu D, Zhao T, Zhao N, Ying H. Economically enhanced succinic acid fermentation from cassava bagasse hydrolysate usingCorynebacterium glutamicumimmobilized in porous polyurethane filler [J].Bioresour. Technol., 2014, 174: 190-197.
[5] Dae-Kyun Ro, Eric M Paradise, Jay D Keasling,et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast [J].Nature, 2005, 440(7086): 940-943.
[6] Patrick J Westfall, Douglas J Pitera, Jay D Keasling,et al. Production of amorphadiene in yeast, and its conversion to dihydroartemisinic acid, precursor to the antimalarial agent artemisinin [J].Proc. Natl. Acad. Sci. USA, 2012, 109(3): E111-E118.
[7] Jennewein S, Park H, DeJong J M, Long R M, Bollon A P, Croteau R B. Coexpression in yeast of Taxus cytochrome P450 reductase with cytochrome P450 oxygenases involved in Taxol biosynthesis [J].Biotechnol. Bioeng., 2005, 89(5): 588-598.
[8] Guo J, Zhou Y J, Hillwig M L, Huang L,et al. CYP76AH1 catalyzes turnover of miltiradiene in tanshinones biosynthesis and enables heterologous production of ferruginol in yeasts [J].Proc. Natl. Acad.Sci.USA, 2013, 110(29): 12108-12113.
[9] Gao Z X, Zhao H, Li Z M, Tan X M, Lu X F. Photosynthetic production of ethanol from carbon dioxide in genetically engineered cyanobacteria [J].Energy & Environmental Science,2012, 5(12): 9857-9865.
[10] Lan E I, Liao J C. Metabolic engineering of cyanobacteria for 1-butanol production from carbon dioxide [J].Metabolic Engineering, 2011, 13(4): 353-363.
[11] Meng D C, Wang Y, Wu L P, Shen R, Chen J C, Wu Q, Chen G Q. Production of poly (3-hydroxypropionate) and poly (3-hydroxybutyrate-co-3-hydroxypropionate) from glucose by engineeringEscherichia coli[J].Metab. Eng., 2015, 29: 189-195.
[12] Jari M, Khatami S R, Galehdari H, Shafiei M. Cloning and expression of poly 3-hydroxybutyrate operon intoEscherichia coli[J].Jundishapur Journal of Microbiology, 2015, 8(2): e16318.
[13] Nahvi A, Sudarsan N, Ebert M S, Zou X, Brown K L, Breaker R R. Genetic control by a metabolite binding mRNA [J].ChemBiol., 2002, 9(9): 1043.
[14] Winkler W C, Cohen-Chalamish S, Breaker R R. An mRNA structure that controls gene expression by binding FMN [J].Proc. Natl. Acad. Sci. USA, 2002, 99: 15908-15913.
[15] Mironov A S, Gusarov I, Rafikov R, Lopez L E, Shatalin K, Kreneva R A, Perumov D A, Nudler E. Sensing small molecules by nascent RNA: a mechanism to control transcription in bacteria [J].Cell, 2002, 111: 747-756.
[16] Breaker R R. Prospects for riboswitch discovery and analysis [J].Molecular Cell, 2011, 43(6): 867-879.
[17] Dann C E 3rd, Wakeman C A, Sieling C L, Baker S C, Irnov I, Winkler W C. Structure and mechanism of a metal-sensing regulatory RNA [J].Cell, 2007, 130(5): 878-892.
[18] Ramesh A, Winkler W C. Magnesium-sensing riboswitches in bacteria [J].Journal of RNA Biol., 2010, 7: 77-83.
[19] Chawla M, Credendino R, Poater A, Oliva R, Cavallo L. Structural stability, acidity, and halide selectivity of the fluoride riboswitch recognition site [J].J. Am. Chem. Soc.,2015, 137(1): 299-306.
[20] Furukawa K, Ramesh A, Zhou Z Y, Weinberg Z, Vallery T, Wade W C, Breaker R R. Bacterial riboswitches cooperatively bind Ni2+or Co2+ions and control expression of heavy metal transporters [J].Molecular Cell, 2015, 57(6): 1088-1098.
[21] McCown P J, Winkler W C, Breaker R R. Mechanism and distribution of glmS ribozymes [J].Methods. Mol. Biol., 2012, 848: 113-129.
[22] Soukup J K. The structural and functional uniqueness of the glmS ribozyme [J].Catalytic Rna., 2013, 120: 173-193.
[23] Mandal M, Lee M, Barrick J E, Weinberg Z, Emilsson G M, Ruzzo W L, Breaker R R. A glycine-dependent riboswitch that uses cooperative binding to control gene expression. [J].Science, 2004, 306(5694): 275-279.
[24] Mellin J R, Pascale Cossart. Unexpected versatility in bacterial riboswitches [J].Trends in Genetics, 2015, 31(3): 150-156.
[25] Wilson-Mitchell S N, Grundy F J, Henkin T M. Analysis of lysine recognition and specificity of theBacillus subtilisL box riboswitch [J].Nucleic Acids Res., 2012, 40(12): 5706-5717.
[26] Furukawa K, Gu H, Sudarsan N, Hayakawa Y, Hyodo M, Breaker R R. Identification of ligand analogues that control c-di-GMP riboswitches [J].ACS Chem. Biol., 2012, 7(8): 1436-1443.
[27] Sudarsan N, Lee E R, Weinberg Z, Moy R H, Kim J N, Link K H, Breaker R R. Riboswitches in eubacteria sense the second messenger cyclic di-GMP [J].Science, 2008, 321: 411-413.
[28] Lee E R, Baker J L, Weinberg Z, Sudarsan N, Breaker R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger [J].Science, 2010, 329(5993): 845-848.
[29] Wachsmuth M, Findeiss S, Weissheimer N, Stadler P F, Mörl M. Denovodesign of a synthetic riboswitch that regulates transcription termination [J].Nucleic Acids Res., 2013, 41(4): 2541-2551.
[30] Desai S K, Gallivan J P. Genetic screens and selections for small molecules based on a synthetic riboswitch that activates protein translation [J].Journal of the American Chemical Society, 2004, 126(41): 13247-13254.
[31] Tang J, Breaker R R. Rational design of allosteric ribozymes [J].Chemistry & Biology, 1997, 4(6): 453-459.
[32] Zhou L B, Zeng A P. Exploring lysine riboswitch for metabolic flux control and improvement of L-lysine synthesis inCorynebacterium glutamicum[J].ACS Synth. Biol., 2015, 4(6): 729-734.
[33] Yang J, Seo S W, Jang S, Shin S I, Lim C H, Roh T Y, Jung G Y. Synthetic RNA devices to expedite the evolution of metabolite-producing microbes [J].Nat. Commun., 2013, 4: 1413.
[34] Muranaka N, Sharma V, Nomura Y, Yokobayashi Y. An efficient platform for genetic selection and screening of gene switches inEscherichia coli[J].Nucleic Acids Res., 2009, 37(5): e39.
[35] Cimdins A, Klinkert B, Aschke-Sonnenborn U, Kaiser F M, Kortmann J, Narberhaus F. Translational control of small heat shock genes in mesophilic andThermophilic cyanobacteriaby RNA thermometers [J].RNA Biol., 2014, 11(5): 594-608.
[36] Deigan K E, Ferré-D'Amaré A R. Riboswitches: discovery of drugs that target bacterial gene-regulatory RNAs [J].Accounts of Chemical Research, 2011, 44(12): 1329-1338.
[37] Li L. The biochemistry and physiology of metallic fluoride: action, mechanism, and implications [J].Critical Reviews in Oral Biology & Medicine, 2003, 14(2): 100-114.
[38] Barbier O, Arreola-Mendoza L, Del Razo L M. Molecular mechanisms of fluoride toxicity [J].Chem. Biol. Interact., 2010, 188(2): 319-333.
[39] Nelson J W, Zhou Z, Breaker R R. Gramicidin D enhances the antibacterial activity of fluoride [J].Bioorg. Med. Chem. Lett., 2014, 24(13): 2969-2971.
[40] Fei X, Holmes T, Diddle J, Hintz L, Delaney D, Stock A, Renner D, McDevitt M, Berkowitz D B, Soukup J K. Phosphatase-inert glucosamine 6-phosphate mimics serve as actuators of the glmS riboswitch [J].ACS Chem. Biol., 2014, 9(12): 2875-2882.
[41] Bren A, Eisenbach M. How signals are heard during bacterial chemo taxis: protein-protein interactions in sensory signal propagation [J].J. Bacteriol.,2000, 182(24): 6865-6873.
[42] Topp S, Gallivan J P. Guiding bacteria with small molecules and RNA [J].J. Am. Chem. Soc., 2007, 129(21): 6807-6811.
[43] Joshua K Michener, Christina D Smolke. High-throughput enzyme evolution inSaccharomyces cerevisiaeusing a synthetic RNA switch [J].Metabolic Engineering,2012, 14(4): 306-316.
[44] Zhu X, Wang X, Zhang C, Wang X, Gu Q. A riboswitch sensor to determine vitamin B12in fermented foods [J].Journal of Food Chem.,2015, 15(175): 523-528.
[45] Knudsen S M, Lee J, Ellington A D, Savran C A. Ribozyme-mediated signal augmentation on a aass-sensitive biosenser [J].J. Am. Chem.Soc., 2006, 128(50): 15936-15937.
[46] Paige J S, Wu K Y, Jaffrey S R. RNA mimics of green fluorescent protein [J].Science,2011, 333(6042): 642-646.
[47] Kellenberger C A, Wilson S C, Sales-Lee J, Hammond M C. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP [J].J. Am. Chem. Soc.,2013, 135(13): 4906-4909.
[48] Strack R L, Jaffrey S R. New approaches for sensing metabolites and proteins in live cells using RNA [J].Curr. Opin. Chem. Biol., 2013, 17(4): 651-655.
[49] Nahvi A, Sudarsan N, Ebert M S, Zou X, Brown K L, Breaker R R. Genetic control by a metabolite binding mRNA [J].Chem. Biol., 2002, 9(9): 1043-1049.
Intelligent and fine regulation of microbial cell factory based on riboswitches
ZHAO Yujia,ZHANG Genlin,ZHOU Xiaohong, LI Chun
(Department of Bioengineering,College of Life Science,Beijing Institute of Technology,Beijing100081,China)
Construction of the microbial cell factory is one of the developmental directions of current green chemical industry. The microbial cell factory is a kind of recombined microorganism and its metabolic and regulatory pathways have been reconstructed by metabolic engineering and synthetic biology to synthetic new compounds or to improve the yield of target production. The microbial metabolic pathway is regulated by two points: environment and genetic information. The cell maintains its homeostasis by global transcription factors, messenger molecules and feedback inhibition when the circumstance is changed. Meanwhile, the cell is affected by its own genetic circulate through transcription, translation and post-translational modification to regulate the expression of target gene. The riboswitches are RNA elements which change their conformation when bind to specific ligands such as ions, sugar derivatives, amino acids, nucleic acid derivatives and coenzymes to regulate the process of transcription, translation and splicing of mRNA. The riboswitches are natural biosensors and bioeffectors which can be designed as the intelligent molecular tools to fine regulate microbial cell factories. Using riboswitches in the microbial cell factory can extend the application in the field of chemical, pharmaceutical, environmental protection and food production.
Prof. LI Chun, lichun@bit.edu.cn
10.11949/j.issn.0438-1157.20150935
TQ 936.2
:A
:0438—1157(2015)10—3811—09
2015-06-16收到初稿,2015-07-16收到修改稿。
联系人:李春。
:赵雨佳(1989—),女,博士研究生。
国家杰出青年科学基金项目(21425624);国家自然科学基金面上项目(21376028,21476026)。
Received date: 2015-06-16.
Foundation item: supported by the National Science Fund for Distinguished Young Scholars(21425624)and the National Natural Science Foundation of China(21376028, 21476026).
