肠球菌万古霉素耐药基因簇遗传特性
2015-02-04陈春辉徐晓刚
陈春辉,徐晓刚
肠球菌万古霉素耐药基因簇遗传特性
陈春辉,徐晓刚
复旦大学附属华山医院抗生素研究所,卫生部抗生素临床药理重点实验室,上海 200040
万古霉素耐药肠球菌自20世纪80年代后期被发现以来,已逐渐发展成为重要的医院感染病原菌。此类耐药肠球菌携带的万古霉素耐药基因簇编码产物可催化合成与万古霉素、替考拉宁等糖肽类抗生素亲和力极低的细胞壁前体导致耐药。目前已在肠球菌中发现的万古霉素耐药基因簇根据基因序列及构成不同分为9个型别;依据它们编码的连接酶合成产物不同又可分为D-Ala:D-Lac连接酶基因簇(VanA、VanB、VanD及VanM型)和D-Ala:D-Ser连接酶基因簇(VanC、VanE、VanG、VanL和VanN型)。这些耐药基因簇介导的耐药水平及其传播模式各有特点。文章综述了肠球菌中万古霉素耐药基因簇的类型、基因构成及传播特性。
肠球菌属;万古霉素;耐药;基因簇
肠球菌属(spp)是人类肠道以及泌尿生殖道正常菌群,也可作为条件致病菌引起尿路、腹腔、伤口感染,以及血流感染和心内膜炎等严重感染[1]。肠球菌属细菌中引起感染最多见的是粪肠球菌(),其次为屎肠球菌(),其余尚可见少量鹑鸡肠球菌()、铅黄肠球菌()、黄色肠球菌()等[1]。万古霉素是一种高分子量的糖肽类抗菌药物,目前临床使用的同类药物还有替考拉宁,它们通过与革兰氏阳性菌细胞壁肽聚糖前体结合,抑制细菌细胞壁合成发挥抗菌作用,临床上大多数革兰氏阳性菌对此类抗生素敏感[2]。万古霉素耐药肠球菌(Vancomycin-resistant enterococcus, VRE)自1988年在欧洲被首次报道以来,此类细菌的检出呈增长趋势[3]。VRE的耐药机制主要与万古霉素等糖肽类药物作用靶位改变有关,万古霉素耐药基因簇是介导此类靶位改变的遗传物质[4]。本文对肠球菌中万古霉素耐药基因簇的类型、基因构成及传播特性进行了综述。
1 万古霉素耐药基因簇种类
万古霉素等糖肽类抗菌药物通过与革兰氏阳性菌细胞壁肽聚糖前体中的D-丙氨酸-D-丙氨酸(D-Ala-D-Ala)末端结合,抑制细菌细胞壁合成而发挥抗菌作用。VRE携带的万古霉素耐药基因簇(gene cluster)可编码多种蛋白,合成新的细胞壁前体,并清除原有含D-Ala-D-Ala的细胞壁前体,致使糖肽类药物与细胞壁前体的亲和力下降,进而产生耐药性,是已知细菌耐药机制中最复杂的机制。根据这些基因簇的基因序列以及基因结构不同可分为9种类型,并根据它们携带的连接酶分别命名为VanA、VanB、VanC、VanD、VanE、VanG、VanL、VanM和VanN型万古霉素耐药基因簇[4~7]。
根据基因簇编码蛋白催化产生的细胞壁前体不同又可分为2类:一类是D-Ala:D-Lac连接酶(D-Ala: D-Lac ligase)基因簇,包括VanA、VanB、VanD和VanM,介导产生的细胞壁前体含D-丙氨酸-D-乳酸(D-Ala-D-Lac)末端,常介导宿主菌对万古霉素高度耐药;另一类为D-Ala:D-Ser连接酶(D-Ala:D-Ser ligase)基因簇,包括VanC、VanE、VanG、VanL和VanN,介导产生的细胞壁前体含D-丙氨酸-D-丝氨酸(D-Ala-D-Ser)末端,携带此类基因簇的菌株表现为对万古霉素低水平耐药,对替考拉宁敏感[4]。系统进化分析结果显示,这些基因簇的连接酶基因具有共同起源,但基因较为特殊,编码蛋白具有D-Ala:D-Ser连接酶活性,但起源与VanA、VanB等D-Ala:D-Lac连接酶的基因相近[7,8]。
1.1 D-Ala:D-Lac连接酶基因簇
最早发现的D-Ala:D-Lac耐药基因簇是VanA型耐药基因簇,含7个基因,可编码产生7种功能蛋白,包括VanS、VanR、VanA、VanH、VanX、VanY以及VanZ(图1)。根据它们的作用可分为3类。(1)VanS和VanR构成的两元件调控系统(Two-component regulatory system),VanS是一种膜蛋白,当环境中有万古霉素存在时,VanS被激活产生组氨酸激酶活性,致使调控蛋白VanR磷酸化,进而激活下游与耐药相关基因表达。近期的蛋白质组学研究也证实,VRE菌株在万古霉素作用下,可出现VanA、VanX等耐药相关蛋白表达上调[9]。(2)VanH、VanA、VanX构成的耐药相关蛋白,其中VanH是细菌合成D-乳酸(D-Lac)所需的丙酮酸脱氢酶;VanA蛋白是D-Ala:D-Lac连接酶,是介导细菌耐药的关键蛋白,也是基因簇分型的主要依据,该蛋白可催化二肽D-Ala-D-Lac的生成,替代正常肽聚糖五肽前体中的D-Ala-D-Ala,生成与万古霉素亲和力极低的新UDP-NAM五肽前体;VanX是D,D二肽酶(D,D- dipeptidase),可降解细菌合成的D-Ala-D-Ala。(3)辅助蛋白VanY为D,D羧肽酶(D,D-carboxypeptidase),可破坏细菌已经生成的正常肽聚糖五肽前体末端的D-Ala-D-Ala残基,导致万古霉素无法与靶位结合产生耐药;VanZ蛋白可引起细菌对替考拉宁产生低水平耐药,其机制尚未明确[4,10]。
VanB、VanD和VanM型耐药基因簇的结构和功能与VanA型相似,均含两元件调控系统、丙酮酸脱氢酶、D-Ala:D-Lac连接酶、D,D二肽酶及D,D羧肽酶等蛋白的编码基因,并具有较高的序列一致性;但VanB型没有,但有一尚未明确功能的基因,VanD型和VanM型耐药基因簇中则与均不存在(图1)。此外,VanB、VanD和VanM型基因簇与VanA型基因簇的两元件调控系统间的序列不完全相同,介导下游基因表达的功能也存在差异,VanA型的VanS蛋白的组氨酸激酶活性可被万古霉素及替考拉宁激活,而VanB型的VanS蛋白仅能被万古霉素激活,故VanA型VRE对万古霉素及替考拉宁均耐药,VanB型通常仅对万古霉素耐药。VanD型基因簇的两元件调控系统常因突变导致下游耐药相关基因持续表达,致使VanD型VRE可对万古霉素和替考拉宁均耐药,但部分VanD型VRE菌株对替考拉宁敏感,其机制不明;VanM型VRE在万古霉素和替考拉宁诱导下可产生含D-Ala-D-Lac的细胞壁前体,但部分菌株对万古霉素的耐药呈异质性,还有部分菌株对替考拉宁敏感,其机制有待进一步研究[4,6,11,12]。
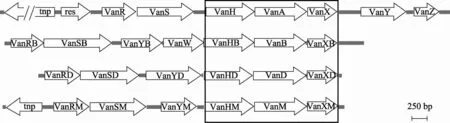
图1 D-Ala:D-Lac连接酶基因簇结构示意图
VanA、VanB、VanD及VanM型基因簇分别根据GenBank登录号为M97297(屎肠球菌BM4147)、EFU35369(粪肠球菌V583)、AF130997(屎肠球菌BM4339)及FJ349556(屎肠球菌Efm-HS0661)的序列及注释绘制。方框内的丙酮酸脱氢酶(VanH、VanHB、VanHD及VanHM),D-Ala: D-Lac连接酶(VanA、VanB、VanD及VanM),D, D二肽酶(VanX、VanXB、VanXD及VanXM)的基因编码肠球菌万古霉素耐药的关键蛋白。
1.2 D-Ala:D-Ser连接酶基因簇
最早发现的D-Ala:D-Ser连接酶基因簇是VanC型基因簇,由5个基因构成(图2),分别编码3种耐药相关蛋白(VanC、VanXYc、VanT)和两元件调控系统(VanSc、VanRc),其中VanC为D-Ala:D-Ser连接酶,VanXYc为二肽酶兼具VanX和VanY两种功能,VanT具有消旋酶活性,可催化D-Ser的合成,3种蛋白共同作用合成D-Ala-D-Ser,替代正常肽聚糖前体中的D-Ala-D-Ala[4,13]。VanC型两元件调控系统的VanSc仅可被万古霉素激活,故VanC型VRE对万古霉素天然低水平耐药,对替考拉宁敏感。VanC型耐药基因簇根据序列不同可分为3个亚型:VanC-1、VanC-2及VanC-3,分别发现于鹑鸡肠球菌、铅黄肠球菌和黄色肠球菌中,其中VanC-2以及VanC-3具有很高的序列一致性,不易区分[4]。
VanE及VanN型耐药基因簇的结构与VanC型基本相同,均含有5种基因(图2),分别编码耐药相关的D-Ala:D-Ser连接酶、二肽酶、消旋酶以及两元件调控系统[4,7,14]。VanL型基因簇的结构与VanC型相似,但其含有vanTm和vanTr两个消旋酶基因,编码蛋白分别与VanTc的氨基末端及羧基末端同源,且具有相似的功能[4,5]。VanG型基因簇不但所含的连接酶基因在进化上与众不同,而且其基因结构也相对特殊,由8个基因构成(图2),两元件调控系统位于基因簇的前段,在其上游有一功能未明的vanU基因,推测vanU、vanS及vanR共同构成三元件调控系统,它们的下游依次为vanY、vanW、、vanXY及vanT,表达与耐药相关的二肽酶、连接酶及消旋酶,vanW与VanB型基因簇的vanW编码氨基酸序列一致率达49%,但功能亦未明确[4,8]。携带VanE、VanG、VanL及VanN型基因簇的VRE耐药表型与VanC型相似,均表现为对万古霉素低水平耐药,对替考拉宁敏感[4,5,7]。
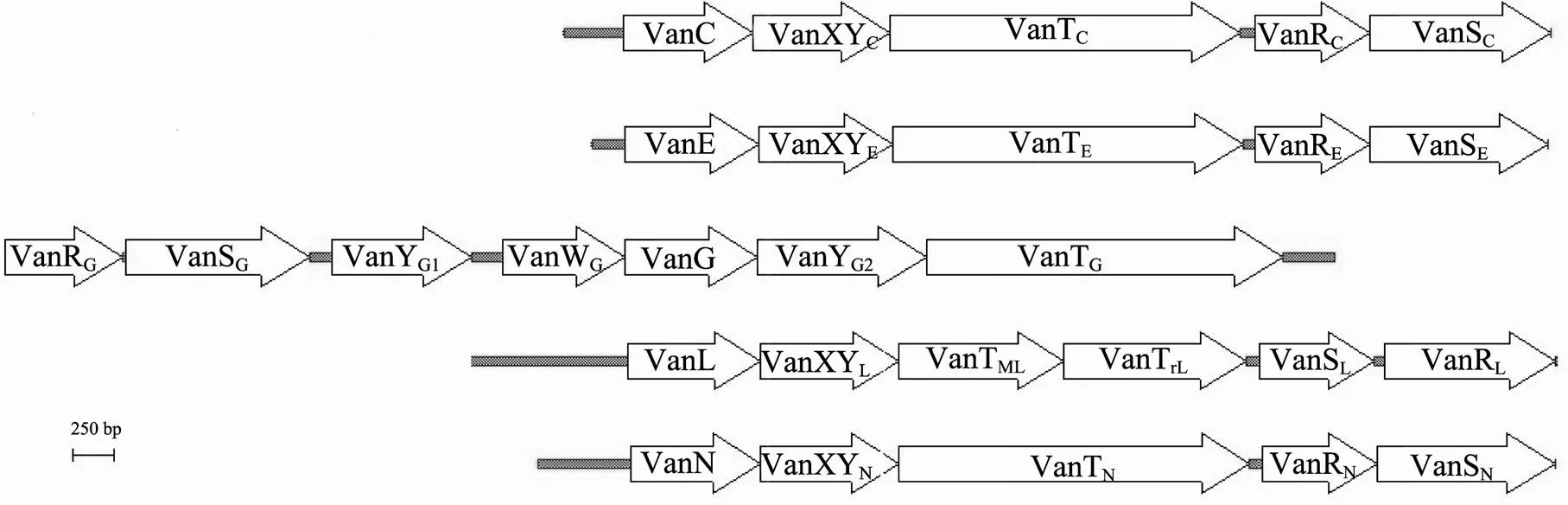
图2 D-Ala:D-Ser连接酶基因簇结构示意图
VanC、VanE、VanG、VanL及VanN型基因簇分别根据GenBank登录号为AF162694(鹑鸡肠球菌BM4174)、FJ872411(粪肠球菌N00-0410)、AF253562(粪肠球菌WCH9)、EU250284(粪肠球菌N06-0364)及JF802084(屎肠球菌UCN71)的序列及注释绘制。其中VanC、VanE及VanN型基因簇的基因结构相同,VanL型略有差异,含VanTmL和VanTrL两个消旋酶基因,但编码蛋白功能与VanC、VanE及VanN型的消旋酶相似;VanG型与其他各型差异较大,推测由多种万古霉素耐药基因簇重组形成[4]。
2 万古霉素耐药基因簇的传播模式
VRE自发现以来已在全球广泛传播,不同来源的VRE菌株常呈现不同的耐药表型。研究显示,耐药表型的差异主要因万古霉素耐药基因簇的不同所致;同一菌种可携带不同的万古霉素耐药基因簇;相同的耐药基因簇也可在不同菌株及菌种中出现,表明万古霉素耐药基因簇可在不同菌株及菌种间传播[4]。
2.1 D-Ala:D-Lac连接酶基因簇的传播
VanA型基因簇在万古霉素耐药的粪肠球菌、屎肠球菌中多见,在鹑鸡肠球菌、铅黄肠球菌及鸟肠球菌()中亦有报道[4]。最初发现该基因簇位于非接合Tn1546转座子[15]。临床分离的VanA型VRE中耐药基因簇主要位于一些可接合转移质粒携带的Tn1546转座子样遗传元件(genetic element),这些携带耐药基因簇的遗传元件偶而也存在于宿主菌染色体[4,16,17]。因此,VanA型耐药基因簇的传播既可通过转座形式获得Tn1546转座子样遗传元件,或通过质粒接合转移来实现。万古霉素耐药金黄色葡萄球菌的产生就是细菌获得了VanA型基因簇的质粒所致[4]。
VanB型耐药基因簇多见于对万古霉素耐药的粪肠球菌及屎肠球菌。通常位于宿主菌染色体携带的一些遗传元件,它们可在染色体间接合转移;临床分离的VanB型VRE菌株中,耐药基因簇也可由质粒携带。分子流行病学研究显示,VanB型耐药基因簇的传播主要由可接合转移的Tn916样转座子介导,但由Tn1549和Tn5382介导的耐药基因簇传播在欧美地区也十分常见[4,18,19]。
VanD型耐药基因簇见于少数万古霉素耐药的粪肠球菌及屎肠球菌。研究显示,该基因簇位于宿主菌染色体,无法通过结合转移,亦无质粒携带及传播的证据,故VanD型基因簇的传播主要通过VanD型VRE菌株的克隆传播来实现[4,12]。VanM型基因簇目前仅在屎肠球菌中检出,序列分析结果显示,该基因簇位于一含转座酶的IS1216样遗传元件,可通过接合转移。临床分离VanM型VRE中,虽然ST78型居多,但有多种不同的MLST型别,亦提示此基因簇不但能通过克隆传播,还能在不同菌株间传播,具体传播机制尚待进一步明确[6]。
2.2 D-Ala:D-Ser连接酶基因簇的传播
VanC型耐药基因簇主要见于万古霉素天然耐药的鹑鸡肠球菌及铅黄肠球菌,存在于这些耐药菌的染色体,通常以克隆播散形式传播[4],近年在粪肠球菌的染色体及质粒中也检测出VanC型耐药基因簇[20],提示该基因簇也可通过相关的遗传元件或质粒传播。虽然VanE型耐药基因簇的基因结构与VanC型基本相同,但VanE型基因簇仅在对万古霉素低水平耐药的粪肠球菌中检出,早期研究结果显示该基因簇不能通过接合转移,但近年研究发现,有些VanE型耐药基因簇由一可接合或整合的遗传元件Tn6202携带,其传播模式也存在多样性[4, 21]。
VanG和VanL型耐药基因簇主要见于对万古霉素低水平耐药的粪肠球菌,由染色体携带[4,5,8]。其中VanG型基因簇可通过接合,在不同染色体间转移,序列分析显示此类转移由一大小约240 kb遗传元件介导[4,22];VanG样基因簇还在其他种属细菌中检出[23,24],它们与肠球菌中VanG型基因簇间的相关性尚不明确。VanL型基因簇由一种含插入序列的遗传元件携带,但尚未证实其能通过接合转移;VanL型基因簇及粪肠球菌基因组的G+C含量分别为32%和38%,此差异提示VanL型基因簇可能源自G+C含量更高的物种[5]。VanN型耐药基因簇目前仅在2株屎肠球菌中检出,也是首个在屎肠球菌中检出的D-Ala:D-Lac连接酶基因簇,可通过接合实验转移至屎肠球菌(BM4107),但不能转移至粪肠球菌(JH2-2),其具体传播模式还有待进一步研究[7]。
3 结 语
万古霉素耐药基因簇是肠球菌对万古霉素耐药的遗传基础。随着万古霉素等糖肽类抗生素的广泛应用,在其形成的抗生素选择压力作用下,具有万古霉素耐药基因簇的菌株具有生存优势。肠球菌的耐药基因簇既可天然携带,亦可从外界获取,并且可通过特定传播模式,在不同菌株、甚至菌种间传播。近年出现的万古霉素高水平耐药金黄色葡萄球菌主要为VanA型基因簇传播所致[25],但金黄色葡萄球菌万古霉素耐药机制不仅限于此,诸如sRNA(SprX)介导的SpoVG蛋白表达调控机制也参与其中[26],肠球菌耐药基因簇的表达是否存在类似机制有待进一步研究。
总之,通过对耐药基因簇分类、表达调控机制、介导耐药特性及传播模式的分析,有助于了解肠球菌万古霉素耐药性的形成及传播机制,为此类耐药菌感染的防治积累理论依据。
[1] Fisher K, Phillips C. The ecology, epidemiology and virulence of., 2009, 155(Pt 6): 1749–1757.
[2] Levine DP. Vancomycin: a history., 2006, 42(Suppl 1): S5–S12.
[3] Bonten MJ, Willems R, Weinstein RA. Vancomycin-resistant enterococci: why are they here, and where do they come from?, 2001, 1(5): 314–325.
[4] Courvalin P. Vancomycin resistance in gram-positive cocci., 2006, 42(Suppl. 1): S25–S34.
[5] Boyd DA, Willey BM, Fawcett D, Gillani N, Mulvey MR. Molecular characterization ofN06–0364 with low-level vancomycin resistance harboring a novel D-Ala-D-Ser gene cluster,., 2008, 52(7): 2667–2672.
[6] Xu XG, Lin DF, Yan GQ, Ye XY, Wu S, Guo Y, Zhu DM, Hu FP, Zhang YY, Wang F, Jacoby GA, Wang MG., a new glycopeptide resistance gene cluster found in., 2010,54(11): 4643–4647.
[7] Lebreton F, Depardieu F, Bourdon N, Fines-Guyon M, Berger P, Camiade S, Leclercq R, Courvalin P, Cattoir V. D-Ala-d-Ser VanN-type transferable vancomycin resistance in., 2011, 55(10): 4606–4612.
[8] McKessar SJ, Berry AM, Bell JM, Turnidge JD, Paton JC. Genetic characterization of, a novel vancomycin resistance locus of., 2000, 44(11): 3224–3228.
[9] Ramos S, Chafsey I, Silva N, Hébraud M, Santos H, Capelo-Martinez JL, Poeta P, Igrejas G. Effect of vancomycin on the proteome of the multiresistantSU18 strain., 2015, 113: 378–387.
[10] Bugg TDH, Wright GD, Dutka-Malen S, Arthur M, Courvalin P, Walsh CT. Molecular basis for vancomycin resistance inBM4147: biosynthesis of a depsipeptide peptidoglycan precursor by vancomycin resistance proteins VanH and VanA., 1991, 30(43): 10408–10415.
[11] Evers S, Reynolds PE, Courvalin P. Sequence of theandgenes encoding D-alanine: D-lactate and D-alanine: D-alanine ligases in vancomycin-resistantV583.1994, 140(1): 97–102.
[12] Depardieu F, Reynolds PE, Courvalin P. VanD-type vancomycin-resistant10/96A., 2003, 47(1): 7–18.
[13] Dutka-Malen S, Molinas C, Arthur M, Courvalin P. Sequence of thegene ofBM4174 encoding a D-alanine: D-alanine ligase-related protein necessary for vancomycin resistance., 1992, 112(1): 53–58.
[14] Patiño LA, Courvalin P, Perichon B.gene cluster of vancomycin-resistantBM4405., 2002, 184(23): 6457–6464.
[15] Arthur M, Molinas C, Depardieu F, Courvalin P. Characterization of Tn1546, a Tn3-related transposon conferring glycopeptide resistance by synthesis of depsipeptide peptidoglycan precursors inBM4147., 1993, 175(1): 117–127.
[16] Leclercq R, Derlot E, Duval J, Courvalin P. Plasmid-mediated resistance to vancomycin and teicoplanin in., 1988, 319(3): 157–161.
[17] Handwerger S, Skoble J. Identification of chromosomal mobile element conferring high-level vancomycin resistance in., 1995, 39(11): 2446–2453.
[18] Quintiliani R Jr, Courvalin P. Conjugal transfer of the vancomycin resistance determinantbetween enterococci involves the movement of large genetic elements from chromosome to chromosome., 1994, 119(3): 359–363.
[19] Rice LB, Carias LL, Donskey CL, Rudin SD. Transferable, plasmid-mediated-type glycopeptide resistance in., 1998, 42(4): 963–964.
[20] Moura TM, Cassenego AP, Campos FS, Ribeiro AM, Franco AC, d'Azevedo PA, Frazzon J, Frazzon AP. Detection ofgene transcription in vancomycin-susceptible., 2013, 108(4): 453–456.
[21] Boyd DA, Mulvey MR. The VanE operon inN00–410 is found on a putative integrative and conjugative element, Tn6202., 2013, 68(2): 294–299.
[22] Depardieu F, Bonora MG, Reynolds PE, Courvalin P. Theglycopeptides resistance operon fromrevisited., 2003, 50(3): 931–948.
[23] Srinivasan V, Metcalf BJ, Knipe KM, Ouattara M, McGee L, Shewmaker PL, Glennen A, Nichols M, Harris C, Brimmage M, Ostrowsky B, Park CJ, Schrag SJ, Frace MA, Sammons SA, Beall B.element insertions within a conserved chromosomal site conferring vancomycin resistance toand., 2014, 5(4): e01386–14.
[24] Peltier J, Courtin P, El Meouche I, Catel-Ferreira M, Chapot-Chartier MP, Lemée L, Pons JL. Genomic and expression analysis of the-like gene cluster of., 2013, 159(Pt 7): 1510– 1520.
[25] Gardete S, Tomasz A. Mechanisms of vancomycin resistance in., 2014, 124(7): 2836–2840.
[26] Eyraud A, Tattevin P, Chabelskaya S, Felden B. A small RNA controls a protein regulator involved in antibiotic resistance in., 2014, 42(8): 4892–4905.
(责任编委: 刘钢)
Genetic characteristics of vancomycin resistance gene cluster inspp
Chunhui Chen, Xiaogang Xu
Vancomycin resistant enterococci has become an important nosocomial pathogen since it is discovered in late 1980s. The products, encoded by vancomycin resistant gene cluster in enterococci, catalyze the synthesis of peptidoglycan precursors with low affinity with glycopeptide antibiotics including vancomycin and teicoplanin and lead to resistance. These vancomycin resistant gene clusters are classified into nine types according to their gene sequences and organization, or D-Ala:D-Lac (VanA, VanB, VanD and VanM) and D-Ala:D-Ser (VanC, VanE, VanG, VanL and VanN) ligase gene clusters based on the differences of their encoded ligases. Moreover, these gene clusters are characterized by their different resistance levels and infection models. In this review, we summarize the classification, gene organization and infection model of vancomycin resistant gene cluster inspp.
spp; vancomycin; resistance; gene cluster
2014-11-29;
2014-12-15
国家自然科学基金项目(编号:81171613)资助
陈春辉,博士研究生,研究方向:病原菌耐药机制。E-mail:12111220002@fudan.edu.cn
徐晓刚,博士,副主任医师,研究方向:病原菌耐药机制。E-mail:xuxiaogang@fudan.edu.cn
10.16288/j.yczz.14-420
2015-2-9 17:08:19
http://www.cnki.net/kcms/detail/11.1913.R.20150209.1708.002.html
