锯齿状病变组织中Runx3基因的甲基化及蛋白表达
2014-11-27张玉萍王鲁平许春伟
张玉萍,王鲁平,方 园,许春伟,葛 畅
(北京军区总医院 病理科,北京 100700)
锯齿状病变组织中Runx3基因的甲基化及蛋白表达
张玉萍,王鲁平*,方 园,许春伟,葛 畅
(北京军区总医院 病理科,北京 100700)
目的探讨Runx3基因启动子区CpG岛甲基化状态和蛋白表达在锯齿状病变发生与癌变通路中的作用及意义。方法用TaqMan 探针为基础的实时定量PCR(Methylight)法检测77例锯齿状病变(29例HP、29例SSA/P和19例TSA)、16例正常结直肠组织和14例结直肠癌中Runx3基因CpG岛甲基化状态,同时用免疫组化法检测相应组织中Runx3蛋白的表达;并分析两者之间的关系。结果Runx3启动子CpG岛甲基化率在正常和HP、SSA/P、TSA、CRC分别为12.5%(2/16)和17.2%(5/29)、51.7%(15/29)、63.2%(12/19)和78.6%(11/14)。Runx3蛋白阳性表达率在正常和HP、SSA/P、TSA、CRC分别为81.3%(13/16)和72.2%(21/29)、48.3%(14/29)、31.6%(6/19)、21.4%(3/14)。SSA/P、TSA和CRC 3组中Runx3甲基化与Runx3蛋白表达结果具有相关性(Plt;0.05),且为负相关。结论Runx3基因启动子区域甲基化是诱导Runx3蛋白表达下调或缺失的主要原因,在锯齿状病变尤其是锯齿状腺瘤的发生及癌变通路中起重要作用。
DNA甲基化;Runx3基因;锯齿状病变;免疫组化
锯齿状病变(Serrated Lesions)是一组以上皮呈锯齿状结构为特征的结直肠息肉/腺瘤,包括增生性息肉(hyperplastic polyp,HP)、传统锯齿状腺瘤(traditional serrated adenoma,TSA)、广基锯齿状腺瘤/息肉(sessile serrated adenoma/polyp,SSA/P);具有恶变为结直肠癌(colorectal cancer, CRC)的潜能,近年来受到临床病理及分子遗传学工作者的广泛关注。据最新报告,60%的CRC来自普通腺瘤,35%来自锯齿状通路[1],尤其是来自CpG岛甲基化表型(CIMP)的锯齿状病变。锯齿状通路是与经典“腺瘤-癌”并存的一种新的致癌途径,该通路涉及一系列的表观遗传学和基因学改变[2]。DNA甲基化是表观遗传学的一种主要调控机制,异常甲基化将引起抑癌基因表达下调甚至沉默,进而导致肿瘤的发生。
Runx3(runt-related transcription factor 3)基因是近年来新发现的一种抑癌基因,基因定位于人染色体1p36.1上,Runx3蛋白在TGF-β(transforming growth factor beta)信号通路中发挥重要作用。Runx3基因的表达下调或沉默在多种人类肿瘤的发生发展过程中起着重要作用,启动子区CpG岛高甲基化是Runx3基因失活的主要机制[3]。研究发现,Runx3基因指导TGF-β信号传导过程而抑制细胞增生并促进凋亡,在结直肠癌的发病机制中扮演重要角色[4-5],而Runx3基因在锯齿状通路中作用尚不明确。目前国内、外有关Runx3在锯齿状病变中作用的研究报告很少,更没有相关表观遗传学方面的研究。本研究通过Taqman探针实时荧光定量PCR法来分析锯齿状病变中Runx3基因启动子区CpG岛甲基化状态,并观察相应组织中Runx3蛋白的表达情况,从而对Runx3基因在锯齿状通路中的作用进行初步探讨。
1 材料与方法
1.1 材料
1.1.1 标本: 收集2007至2012年间北京军区总医院病理诊断为锯齿状病变291例,进行回顾性阅片。 按WHO(2010)消化系统肿瘤分类[6]及第2版《诊断病理学》标准,由3名病理医师4~5轮阅片,从中筛选出77例锯齿状病变(29例HP、29例SSA/P和19例TSA)作为实验组,并收集16例正常结直肠组织和14例结直肠癌作为对照组,所有实验标本均经患者知情同意。
1.1.2 主要试剂和仪器: DNA提取试剂盒(QIAGEN公司),甲基化修饰试剂盒(ZYMO公司),核酸蛋白质浓度测量仪B-500(上海创萌生物科技有限公司),甲基化阳性/阴性对照(ZYMO公司),实时荧光定量PCR反应试剂盒(TaKaRa公司),Runx3抗体[日本MBL公司(1∶500稀释)],PCR引物和探针参照文献[7]设计,内参基因β-肌动蛋白(β-actin)引物和探针参照文献[8]设计(表1),由上海辉睿生物科技有限公司合成。Mx3000P定量PCR扩增仪(Stratagene公司)。
1.2 方法
1.2.1 DNA提取:采用QIAamp DNA FFPE Tissue Kit试剂盒提取组织DNA,将含有DNA组织的蜡块连切5张10 μm的厚蜡膜,严格按照试剂盒说明步骤进行操作。并用紫外分光光度计测量其纯度和浓度备用。
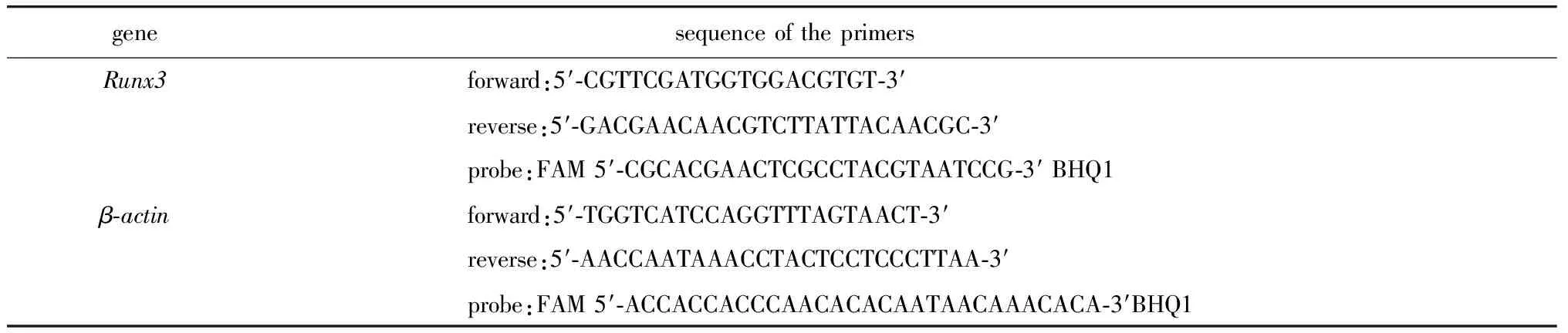
表1 Runx3基因和内参基因的引物及探针序列Table 1 The sequence of the primers and the probes of the Runx3 gene and the Reference gene
1.2.2 甲基化修饰:采用EZ DNA Methylation-GoldTMKit(D5005)试剂盒,严格按照试剂盒说明步骤进行操作。经此步后,DNA序列中未甲基化的胞嘧啶(C)转变为尿嘧啶(U)。
1.2.3 Taqman探针为基础的实时定量PCR分析:PCR反应体系(20 μL):2×Taq PCR Master Mix 10 μL;修饰后的DNA模板2 μL;上、下游引物各1 μL(10 pmol);探针FAM 0.4 μL(10 pmol);ROX 0.3 μL。反应条件:94 ℃ 5 min预变性;94 ℃ 30 s,52 ℃ 45 s, 72 ℃ 45 s,共45个循环(收集荧光信号);72 ℃延伸5 min。经亚硫酸氢盐修饰的人的CpG全甲基基因组DNA作为阳性对照,人的CpG去甲基基因组DNA作为阴性对照。所有PCR均做复孔,取其平均值作为结果。
1.2.4 免疫组织化学染色:所有标本常规石蜡包埋,4 μm厚连续切片,60 ℃温箱烘烤90 min。采用EnVision二步法,实验过程严格按照试剂盒说明书进行,高温高压抗原修复,DAB显色,PBS代替一抗为阴性对照,已知阳性的结肠腺体组织为阳性对照。
1.2.5 结果判断标准:TaqMan探针法RT-PCR结果判断标准[9]:同时扩增目的基因(Runx3)和内参基因(β-actin),根据标准曲线得到两者的原始拷贝数,计算标准甲基化指数(the normalized index of methylation, NIM)其定义为:NIM=[(Runx3 sample/Runx3 positive)/ β-actin sample/β-actin positive)]×100,其中Runx3 sample指样本中甲基化Runx3基因的拷贝数,Runx3 positive指阳性对照中甲基化Runx3基因的拷贝数,β-actin sample和β-actin positve与上述相同。NIM≥4为甲基化,NIMlt;4为非甲基化。
免疫组化判断标准:Runx3阳性定位于细胞核;标记指数计算方法[10]:每张切片低倍镜下选择组织结构良好、比较清晰的5个阳性细胞最为密集的区域,每个区域在高倍镜下,计数100个细胞中的阳性细胞指数(不包括间质细胞和其他非肿瘤细胞),计算阳性细胞数平均值的百分率。标记指数计分:lt;10%为0分,10%~25%为1分,26%~50%为2分,51%~75%为3分,76%~100%为4分。染色强度计分:淡黄色为1分,棕黄色为2分,棕褐色为3分。每张切片两种评分之乘积为该切片最后的表达强度:0分为(-),1~3分为(+),4~6分为(++),≥8分为(+++)。
1.3 统计学分析
所有数据采用SPSS17.0统计软件,结果运用χ2、Fisher确切概率法及Pearson相关进行统计学处理,检验水准α=0.05。
2 结果
2.1 Runx3基因标准曲线分析
将阳性对照按10的倍数稀释成1~1×10-67个浓度梯度制作标准曲线(其拷贝数为103~109/mL),各浓度梯度反应均做复孔。Taqman探针方法的线性范围为104~108拷贝/mL,R2为0.998。
2.2 Runx3基因启动子CpG岛甲基化状态
通过对样品的循环阈值数(Ct值)和扩增曲线分析:Runx3启动子CpG岛甲基化率在正常组织、HP、SSA/P、TSA和CRC分别为12.5%(2/16)、17.2%(5/29)、51.7%(15/29)、63.2%(12/19)和78.6%(11/14),正常组织中未测到Runx3启动子CpG岛甲基化。SSA/P、TSA、CRC和正常组及HP组之间有显著性差异(Plt;0.05);HP与正常组之间,SSA/P、TSA、CRC 3组之间差异均不显著(表2;图1,2)。
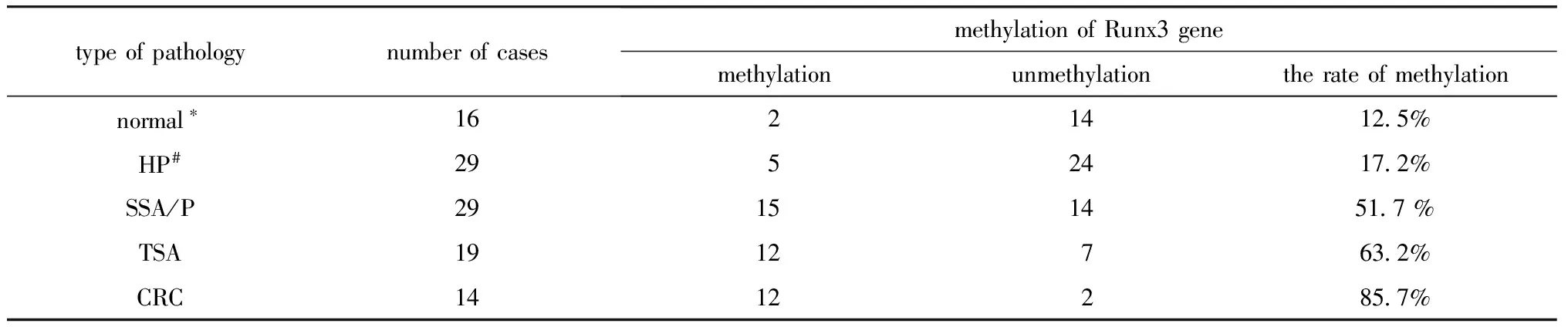
表2 基因CpG岛甲基化统计结果Table 2 The survey results of genes CpG Island methylation
*SSA/P, TSA and CRC group compared with normal group respectively;Plt;0.05;#SSA/P, TSA and CRC group compared with HP group respectively;Plt;0.05
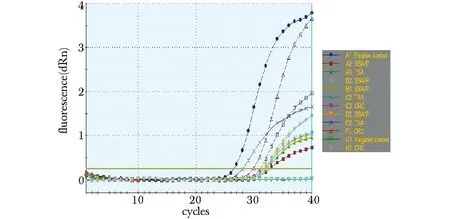
图1 SSA/P、TSA和CRC的MethyLight扩增曲线Fig 1 Expansion curve of MethyLight in SSA/P, TSA and CRC

图2 HP和正常黏膜组织的MethyLight扩增曲线Fig 2 Expansion curve of MethyLight in HP and normal mucosa tissues
2.3 Runx3蛋白表达
Runx3蛋白阳性表达率在正常、HP、SSA/P、TSA 和CRC分别为81.3%(13/16)、65.5%(19/29)、48.3%(14/29)、31.6%(6/19)和14.3%(2/14)。SSA/P、TSA、CRC和正常组之间有显著性差异(Plt;0.05);TSA、CRC和HP组之间有显著性差异(Plt;0.05);SSA/P 与CRC之间有显著性差异(Plt;0.05);HP与正常组、SSA/P,SSA/P 和TSA之间差异不显著(表3;图3,4,5,6)。
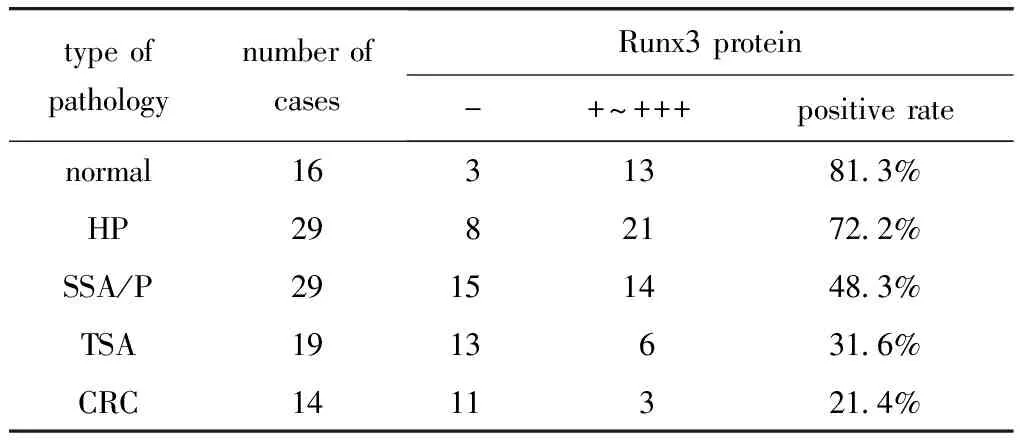
表3 免疫组织化学观察统计结果Table 3 The survey results of the immunohistochemical observation
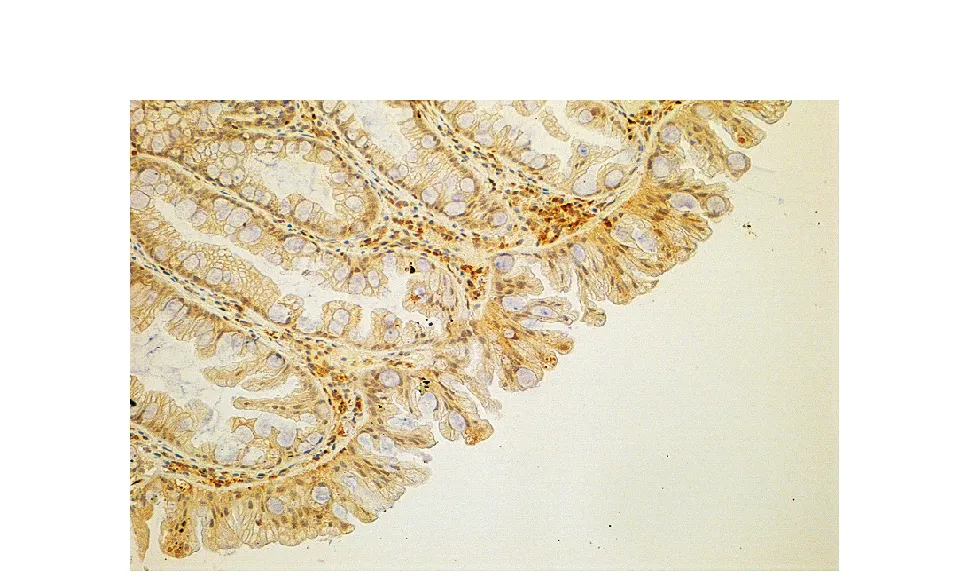
图3 HP中Runx3呈阳性表达Fig 3 Positive expression of Runx3 in HP(×200)
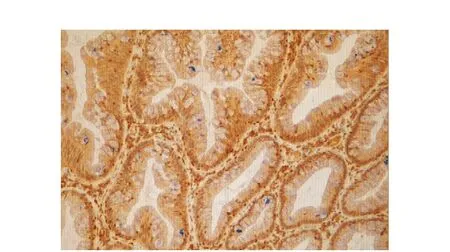
图5 TSA中Runx3呈阳性表达Fig 5 Positive expression of Runx3 in TSA(×200)
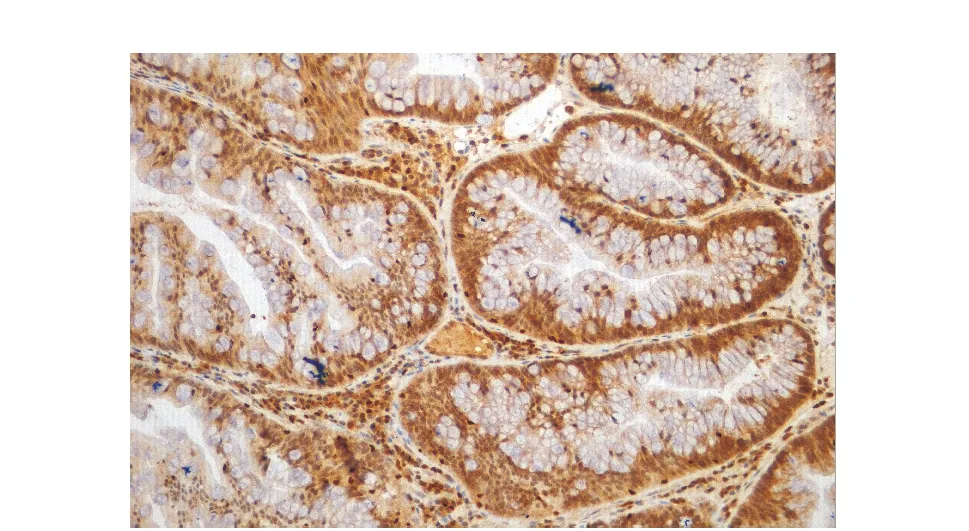
图4 SSA/P中Runx3呈阳性表达Fig 4 Positive expression of Runx3 in SSA/P(×200)
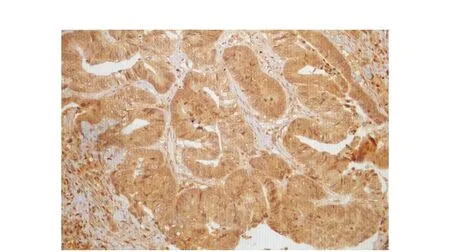
图6 CRC呈阳性表达Fig 6 Positive expression of Runx3 in CRC(×200)
2.4 Runx3基因甲基化与Runx3蛋白相关性分析
经统计学分析显示:SSA/P、TSA和CRC 3组中Runx3甲基化与Runx3蛋白表达结果具有相关性(Plt;0.05),并且为负相关,相关系数分别为γ=-0.724、γ=-0.655和γ=-0.782;而正常结直肠黏膜组织和HP两组中Runx3甲基化与Runx3蛋白表达结果不具有相关性(表4,5)。
3 讨论
本研究发现HP、SSA/P和TSA中均有Runx3启动子CpG岛甲基化,甲基化率为17.2%(5/29)、51.7%(15/29)、63.2%(12/19);而14例CRC中有11例Runx3基因CpG岛甲基化,甲基化率为85.7%(12/14);正常组织中Runx3启动子CpG岛甲基化率为12.5%(2/16)。Kodach等[11]在57.4%(27/47)CRC患者中检测到Runx3基因表达下调或缺失,其中在85.2%(23/27)CRC组织中检测到Runx3基因启动子区域CpG岛甲基化,而在相对应的正常肠组织中未发现Runx3基因甲基化,说明启动子区域的高甲基化导致Runx3基因表达明显下调。袁权利等[12]也发现53.3%(16/30)的CRC组织中存在Runx3基因CpG岛甲基化,正常组织中未发现。但Waki等研究来源于小肠、大肠、胃等不同器官或组织的甲基化相关基因如Runx3、E-cadherin、GSTP1、DAP-kinase、P16、MLH1等发现42岁以后的个体基因启动子CpG岛均以组织特异性的方式发生不同程度的甲基化。我们研究中CRC的甲基化率与已有研究结果基本符合,正常组织中检测到2例存在甲基化,与Waki的研究相吻合,甲基化率的不同可能与样本量、样本来源、引物设计及检测方法有关。至今,国内外尚无对锯齿状病变中Runx3基因甲基化的研究,在我们的研究中,SSA/P、TSA、CRC和正常组及HP组之间有显著性差异(Plt;0.05),这3种病变中Runx3基因的甲基化阳性率明显高于正常组和HP组,且甲基化阳性率在正常、HP、SSA/P、TSA 和CRC组呈递增趋势,推测病变恶变程度越高,Runx3基因的甲基化阳性率越高,从而进一步推测Runx3基因甲基化在锯齿状通路中起推动作用。
有研究者[13]应用MSP技术对人结肠癌细胞系进行检测,发现50%的人结肠癌细胞系Runx3蛋白表达下降或不表达。国外有文献报道[14]通过免疫组织化学方法比较发现HP和TSA中Runx3的阳性表达较正常结肠组织和管状腺瘤(Tubular adenoma,TA)中有所下降。本研究中HP、SSA/P和TSA中Runx3蛋白阳性表达率分别为65.5%(19/29)、48.3%(14/29)、31.6%(6/19),正常结肠黏膜Runx3蛋白阳性表达率达81.3%(13/16),而CRC中Runx3蛋白阳性表达率只有14.3%(2/14)。SSA/P、 TSA和CRC组Runx3蛋白阳性表达率明显低于HP组及正常组,显著性差异(Plt;0.05)。由此推论,Runx3蛋白表达下调或缺失是锯齿状病变恶变进展过程中的一个早期事件,与CRC的发生、发展有关。
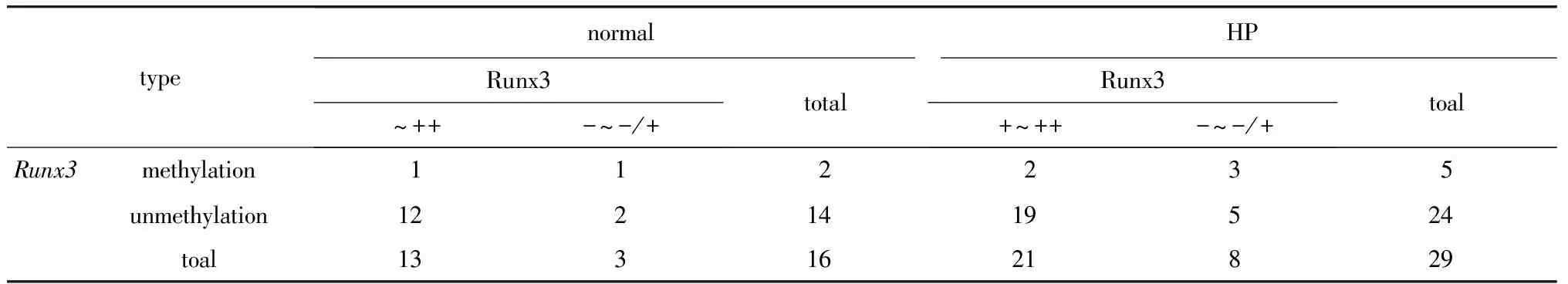
表4 正常黏膜组织和HP组中Runx3蛋白表达与Runx3的甲基化Table 4 The expression of the Runx3 protein and gene methylation in normal mucosa tissues and HP
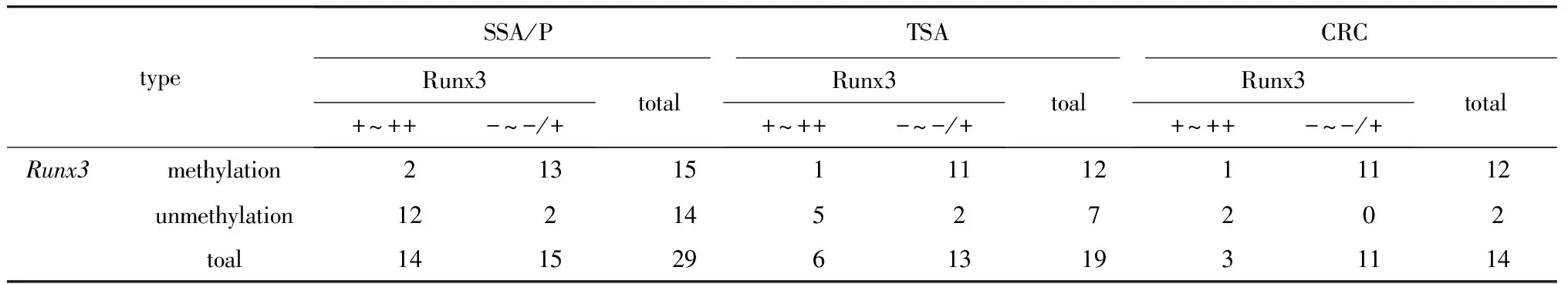
表5 SSA/P、TSA和CRC 3组中Runx3蛋白的表达与Runx3的甲基化Table 5 The expression of the Runx3 protein and gene methylation in SSA/P、TSA and CRC
对Runx3甲基化与Runx3蛋白表达结果进行了相关性分析,结果显示在SSA/P、TSA和CRC中Runx3甲基化与Runx3蛋白表达具有相关性(Plt;0.05),且为负相关,而正常结直肠黏膜组织和HP中Runx3甲基化与Runx3蛋白表达结果不具有相关性。实验结果表明:在SSA/P、TSA和CRC中Runx3基因CpG岛甲基化导致Runx3基因表达沉默,从而导致Runx3蛋白表达下调或缺失。可推测,在锯齿状通路中,Runx3基因CpG岛甲基化推动了SSA/P和TSA向CRC的恶变;Runx3基因CpG岛发生甲基化是从HP开始,提示我们HP也具有的一定的恶变潜能,需引起临床病理医生的关注。
综上所述,Runx3基因启动子区域CpG岛甲基化导致Runx3基因表达下调或缺失,与锯齿状病变的发生、发展密切相关,在锯齿状通路中起重要作用,是CRC发生早期分子事件。
[1] Snover DC. Update on the serrated pathway to colorectal carcinoma[J]. Hum Pathol, 2011,42:1-10.
[2] Makinen MJ. Colorectal serrated adenocarcinoma[J]. Histopathology, 2007,50:131-150.
[3] Chuang LS, Ito Y. RUNX3 is multifunctional in carcinogenesis of multiple solid tumors[J]. Oncogene, 2010,29:2605-2615.
[4] Tong DD, Jiang Y, Li M,etal. RUNX3 inhibits cell proliferation and induces apoptosis by TGF-beta-dependent and-independent mechanisms in human colon carcinoma cells[J]. Pathobiology, 2009,76:163-169.
[5] Peng Z, Tang H, Wang X,etal. Inhibition of the growth and metastasis of human colon cancer by restoration of RUNX3 expression in cancer cells[J]. Int J Oncol, 2008,33:979-984.
[6] HamiltonSR, BosmanFT, BoffettaP. Carcinoma of the colon and rectum. WHO Classification of Tumors of the Digestive System. Pathology and Genetics Tumours and Digestive System[M].4th edition,Switzerland: WHO press, 2010:134-146, 160-165.
[7] Weisenberger DJ, Siegmund KD, Campan M,etal. CpG island methylator phenotype underlies sporadic microsatellite instability and is tightly associated with BRAF mutation in colorectal cancer[J]. Nat Genet, 2006,38:787-793.
[8] 李遇梅, 李政亮, 马红, 等. 两种实时定量PCR方法检测系统性红斑狼疮患者DNA甲基化状态的比较研究[J]. 临床皮肤科杂志, 2010,39:211-213.
[9] Eads CA, Lord RV, Kurumboor SK,etal. Fields of aberrant CpG island hypermethylation in Barrett’s esophagus and associated adenocarcinoma[J]. Cancer Res, 2000,60:5021-5026.
[10] 许良中, 杨文涛. 免疫组织化学反应结果的判断标准[J]. 中国癌症杂志, 1996:229-231.
[11] Kodach LL, Jacobs RJ, Heijmans J,etal. The role of EZH2 and DNA methylation in the silencing of the tumour suppressor RUNX3 in colorectal cancer[J]. Carcinogenesis, 2010,31:1567-1575.
[12] 袁权利, 李华, 白净, 等.Runx3基因甲基化与大肠癌发生和转移的关系[J]. 中国综合临床, 2007,23:345-347.
[13] Ku JL, Kang SB, Shin YK,etal. Promoter hypermethylation downregulates RUNX3 gene expression in colorectal cancer cell lines[J]. Oncogene, 2004,23:6736-6742.
[14] Subramaniam MM, Chan JY, Soong R,etal. RUNX3 inactivation in colorectal polyps arising through different pathways of colonic carcinogenesis[J]. Am J Gastroenterol, 2009,104:426-436.
Methylation and protein expression ofRunx3 gene in serrated lesion tissues
ZHANG Yu-ping, WANG Lu-ping*, FANG Yuan, XU Chun-wei, GE Chang
(Dept. of Pathology, the General Hospital of Beijing Military Region, Beijing 100700, China)
ObjectiveTo investigate the role and significance of methylation status and protein expression of theRunx3 gene in serrated lesions and carcinogenesis pathway.MethodsTheRunx3 gene promoters methylation were detected with the Taqman probe based real-time PCR(Methylight) technology in 77cases serrated lesions(29 cases of hyperplastic polyps, 29 cases of sessile serrated adenoma/polyp and 19 traditional serrated adenoma), 16 cases of normal mucosa tissues and 14 cases of colorectal cancer. At the same time, immunohistochemical staining was utilized to detect the expression of Runx3, and to analyze the relationship of them.ResultsThe rates of methylation of theRunx3 gene in normal mucosa tissues, HP, SSA/P, TSA and CRC were 12.5%(2/16),17.2%(5/29),51.7%(15/29),63.2%(12/19) and 78.6%(11/14).The results of Immunohistochemistry showed that the positive expression of Runx3 in normal mucosa tissues, HP, SSA/P, TSA and CRC were 81.3%(13/16), 72.2%(21/29), 48.3%(14/29), 31.6% (6/19)and 21.4%(3/14). The Runx3 methylayion was negatively correlated with the expression of Runx3 protein in SSA/P, TSA and CRC (Plt;0.05).ConclusionsRunx3 gene promoter region methylation explains the down-regulation of Runx3 protein expression, which may function in the main reasons for the serrated lesions and occurrence of serrated adenoma and cancer.
DNA methylation;Runx3 gene; serrated lesions;immunohistochemistry
2013-06-13
2013-10-09
首都卫生发展科研专项基金(2011-5021-02)
*通信作者(correspondingauthor): BZWLP@yhaoo.com
1001-6325(2014)03-0339-06
研究论文
R 735.3
A
