MUC13腹泻抗性纯合杜洛克种群的选育研究
2014-11-06阮国荣何晓芳肖石军陈金雄李军山刘亚轩孙耀华陈福珍林国忠
阮国荣,何晓芳,黄 晶,肖石军,陈金雄,李军山,刘亚轩,孙耀华,陈福珍,林国忠
(1.福建农业职业技术学院,福建 福州 350119;2.上海农业科学院 畜牧兽医研究所,上海 201106;3.江西农业大学生物技术国家重点实验室培育基地,江西 南昌 330045;4.福建省厦门国寿种猪开发有限公司,福建 厦门 350021;5.福建省光华百斯特生态农牧有限公司,福建 尤溪 365100)
仔猪腹泻是造成仔猪死亡的重要原因。据调查,因腹泻死亡的仔猪占仔猪死亡总数的38.9%[1]。目前应对仔猪腹泻的常见办法主要是饲料中加大抗生素剂量或使用疫苗预防,但防治效果欠佳、且会产生耐药而影响后期生长、甚至导致抗生素残留超标,对食品安全造成影响。为此,改变疾病频发的最好措施是实施抗病选育,从源头上加以改变,培育出先天性抵抗腹泻的种猪品种。
研究表明,产肠毒素大肠杆菌F4(ETEC F4)是引发新生和断奶前仔猪腹泻病的最主要致病菌[2-4]。ETEC F4有3种血清型:F4ab,F4ac和F4ad,其最主要的血清型为F4ac[5]。ETEC F4的致病性取决于其是否能与猪小肠上皮细胞表面刷状缘的受体特异性结合,无受体猪表现为抵抗型,有受体猪则表现为易感型。猪ETEC F4受体是由单基因控制的,呈显隐性遗传模式。携带显性等位基因的猪对ETEC F4的侵染易感,隐性等位基因纯合子猪则表现为抵抗型[6-7]。因此,F4ac受体基因的定位和鉴别是抗腹泻育种的关键。在ETEC F4受体基因的精细定位和主效基因鉴别方面,瑞士Peter Vogeli小组将其精细定位于SW207-MUC4之间[8],黄路生院士[8-12]研究组采用一系列现代遗传分析手段最终鉴别到了编码ETEC F4ac受体的目的基因-MUC13基因,发现了能准确鉴别F4ac易感个体和抗性个体的分子标记(准确率>97%)。而种猪性能测定及BLUP遗传评估选育技术是近20年来国内外猪育种界公认的提高生产性能的有效方法,国内外诸多种猪企业应用该方法进行种猪选育,均取得明显效果[13-16]。为此,将仔猪抗腹泻分子标记育种技术结合种猪性能测定及BLUP遗传评估选育技术开展抗腹泻育种有望培育出生产性能优异的抗腹泻病新品系。
在国外引进瘦肉型猪品种中,杜洛克品种的抗性有利基因频率相对较高[17-18],经少数几代选育将有可能育成抗性品系;而且在商品猪生产上杜洛克常被用作终端父本,具有影响面广的特点。为此,本研究选取两家具有开展性能测定的条件和经验的国家级育种核心场,对其杜洛克种群开展MUC13抗性纯合与性能测定的综合选育研究,希冀培育出生产性能较好的抗F4ac腹泻病猪专门化新品系。
1 材料与方法
1.1 材料
根据血缘、生产性能及体型外貌等表现,分别组建两个杜洛克猪育种核心群,其中厦门国寿220头(20♂、200♀)、光华百斯特55头(5♂、50♀),合计275头(25♂、250♀)。组群后均同步按方案逐代进行检测、测定和选育。两猪场饲养管理相同,营养水平一致。期间两猪场间先后有6头公猪血缘交流。
1.2 选育方案
在两场原有选育群的基础上重新确定育种核心群(25♂,250♀)。记录育种群配种受胎及产仔、哺育个体情况,根据同窝及个体本身表现,对育种群断奶仔猪进行初选(剔除出现遗传疾患的窝别外,其余每窝选择 1~2♂、2~3♀)。
对入选测定(常规性能测定)的仔猪个体进行采样,并进行基因检测。根据生长发育测定结果,经BLUP法计算个体育种值,对综合选择指数高、抗性基因型有利的个体进行外貌鉴定,按指数高低,选留体型外貌符合品种特征的优秀个体留作下一世代育种核心群;允许部分世代重叠。
实施连续3年的反复检测、选留,比较育种群选育前后的生产性能指标变化,以及育种群与未经选育的扩繁群仔猪腹泻的差异。
1.3 MUC13 基因检测
1.3.1 样品采集与保存 按要求对育种核心群每世代选留测定的后代进行耳样采集,以鉴别个体MUC13基因型。
采样部位和方法:用耳号钳在猪耳朵上打下一小块耳组织,放入装有体积分数75%酒精的样品采集管内,拧紧管盖并在管壁和管盖上用标记笔作好耳号记录。
样品保存:采集好的耳组织样品,及时(半天之内)放置在-20℃冰箱内,以防样品腐烂变质。
1.3.2 DNA提取 依照标准酚/氯仿抽提、乙醇沉淀的方法提取,经Nanodrop ND-1000全波长紫外/可见光扫描分光光度计(热电,美国)测定DNA浓度和质量合格后,置于-20℃冰箱保存备用。
1.3.3 基因判别 根据江西农业大学研发的鉴别腹泻易感/抗性个体MUC13分子标记专利技术,委托上海生工生物有限公司合成PCR-Snapshot引物。PCR引物序列(5'-3')为Fp:GGA GAG ACC AAA CCC ACA GA,Rp:CTC CTC ACC AGC TCC TTA GC,目的片段280 bp。PCR反应体系总体积为20 μL,包括模板 DNA 40 ng、10×buffer 2.0 μL、25 mmol/L MgCl21.2 μL、10 mmol/L dNTP 0.3 μL、Fp 0.4 μL、Rp 0.4 μL、Taq聚合酶 0.5 μL、超纯水 13.2 μL。PCR 循环参数:94 ℃变性 3 min,94 ℃ 30 s,退火温度61℃ 30 s,72℃延伸45 s,共36个循环。
SNaPshot判型:采用PCR-SNaPshot判定 MUC13基因型。取1.5 μL纯化后 PCR产物,加入2 μL SNaPshot multiplex mix(含 Taq 聚合酶和荧光标记的 dd-NTPs)、1.2 μL 去离子水和 0.3 μL SNaPshot引物(引物序列为:TTT TTT TTT TTT TTT CCA TGT ACA TTT CAG AGT CTG AGG GAT)。混合均匀后进行SNaPShot反应,反应参数为96℃ 10 s,50℃ 5 s,60℃ 30 s,25个循环。反应结束后在 SNaPshot反应产物中加 0.57 μL 1 × NEB buffer和0.1 μL CIP(NEB,美国),37 ℃ 60 min,75 ℃15 min,进行再次纯化,以清除荧光标记的ddNTPs并灭活纯化酶。最后进行毛细管电泳检测分型,方法是:每1 μL SNaPshot反应产物加入8 μL上样变性剂(由Hi-Di formamide和GeneScan 120 LIZ size standard以20∶1混合而成)95℃变性5 min;然后迅速转置冰水浴中2 min;冷却离心后上样于3130XL遗传分析仪(ABI,美国)进行电泳检测,最后使用GeneMapper 4.0软件(ABI,美国)进行判型分析。判型标准如图1。
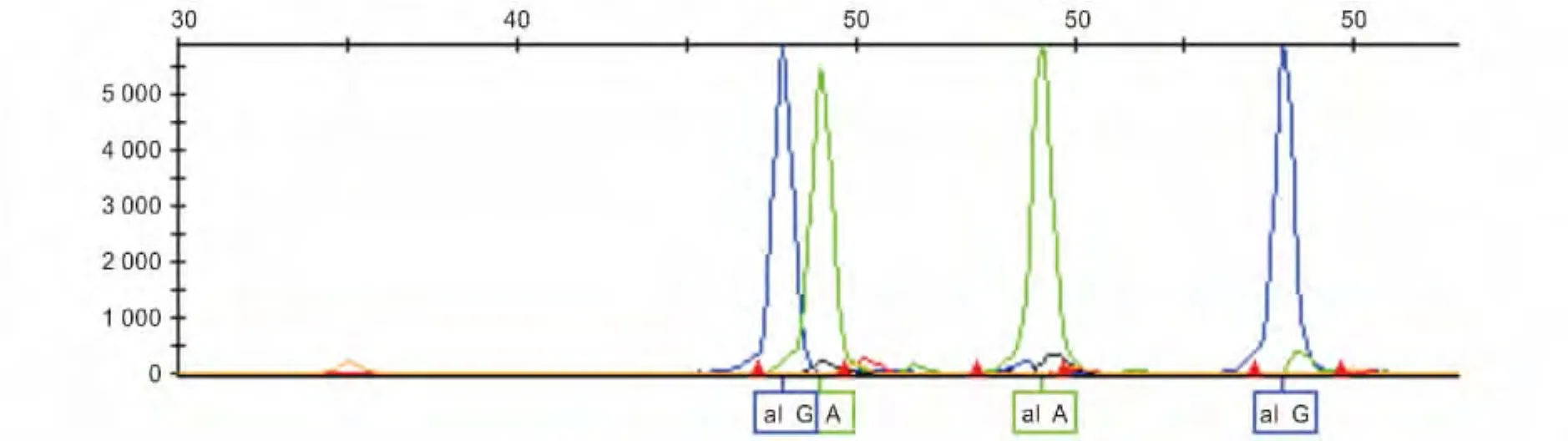
图1 MUC13基因的PCR-SnaPshot检测图Fig.1 SNapshot genotyping patterns at the MUC13 locus
1.4 仔猪腹泻观察
在两场分别选取相同胎龄的核心群与扩繁群杜洛克母猪各10对,安排在相同栏舍,以观察核心群与扩繁群同期出生的仔猪腹泻发生情况。观察实验要求对同期出生(相近1周内)的仔猪记录母猪耳号、分娩日期、同窝仔猪数量,并仔细登记有发生腹泻的每头仔猪耳号、腹泻发生日龄、腹泻延续时间、治疗康复时间等,计算腹泻比例和成活率。
1.5 生长性能测定
根据国家《种猪性能测定技术规程》标准(NY/T 822—2004)要求,剔除繁殖性能低、发生遗传疾患等情况的整窝仔猪外,将每世代断奶、保育选留的待测小猪(每窝选留1~2♂、2~3♀)转入测定舍,按常规饲养到85~105 kg体质量时进行称量、同时用ALOKA 500 B超仪进行活体背膘测量。所有测定数据输入GBS软件,并校正到100 kg体质量时日龄、活体背膘厚。
1.6 不同MUC13基因型个体的生长性能比较
根据个体的生长性能测定结果和MUC13基因型检测结果,分析两场3批计525头杜洛克仔猪MUC13 3种基因型对生长性能的影响效应。
1.7 数据分析
采用SPSS 13.0进行数据统计分析,腹泻发生率与仔猪成活率使用Chi-square test模块;生长发育数据使用One-Way Anova及Post Hoc模块分析。
2 结果与分析
2.1 MUC13 基因检测
由表1可见,通过3个世代的基因检测和筛选,腹泻抗性有利基因G频率逐代明显提高,而不利基因A则在核心群中逐代降低,直至完全剔除。
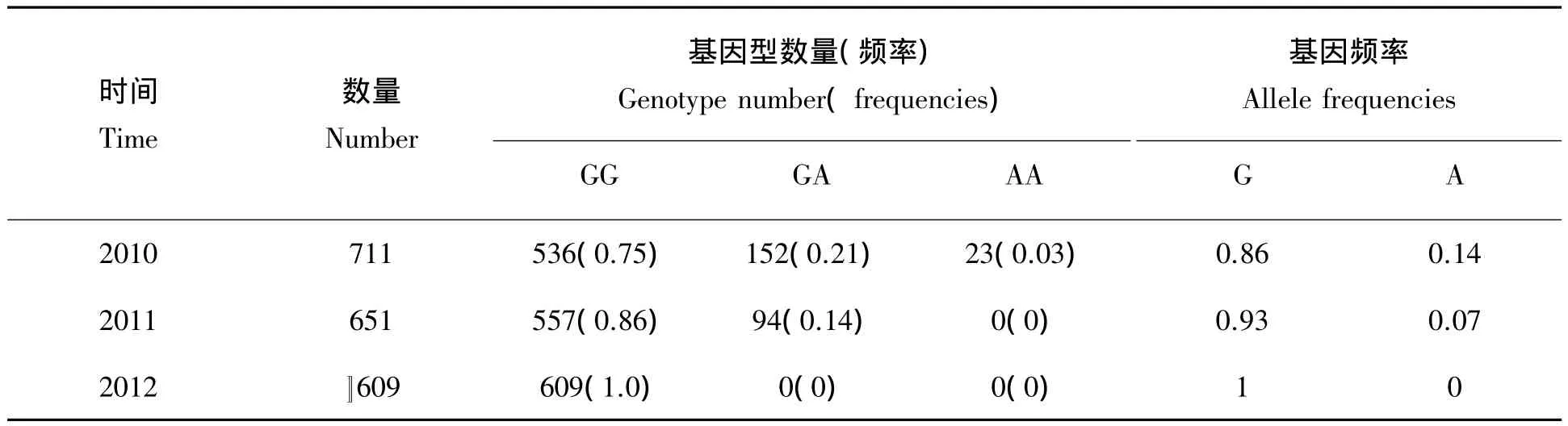
表1 三世代MUC13基因检测结果Tab.1 Genotyping patterns at the MUC13 locus of the three generations pigs
2.2 仔猪腹泻发生情况及哺乳期成活率
由表2可见,在同胎次相同饲养管理条件下,核心群与扩繁群仔猪腹泻发生率分别为8.7%和18.4%,两组间仔猪腹泻发生率差异极显著(P=0.006)。核心群发生腹泻的仔猪治疗后较快康复,扩繁群腹泻仔猪在采取同样治疗措施情况下,腹泻延续时间多需3~4 d以上,两组的腹泻致死率分别为12.5%和16.7%,但差异不显著(P>0.05);哺乳期仔猪成活率分别为94.57%和92.23%,差异也未达到显著水平(P>0.05)。
2.3 生长发育测定选育结果
通过3年来的性能测定与BLUP指数选育,生长速度和活体背膘均有一定改善,其中校正达100 kg体质量日龄由181.34 d降低到177.48 d,但经SPSS 13.0统计分析,各年度间(P>0.05),未达显著水平;而活体背膘由12.23 mm降低到10.77 mm,经分析并比较,2010年与2012年间差异达到显著水平(P<0.05)。
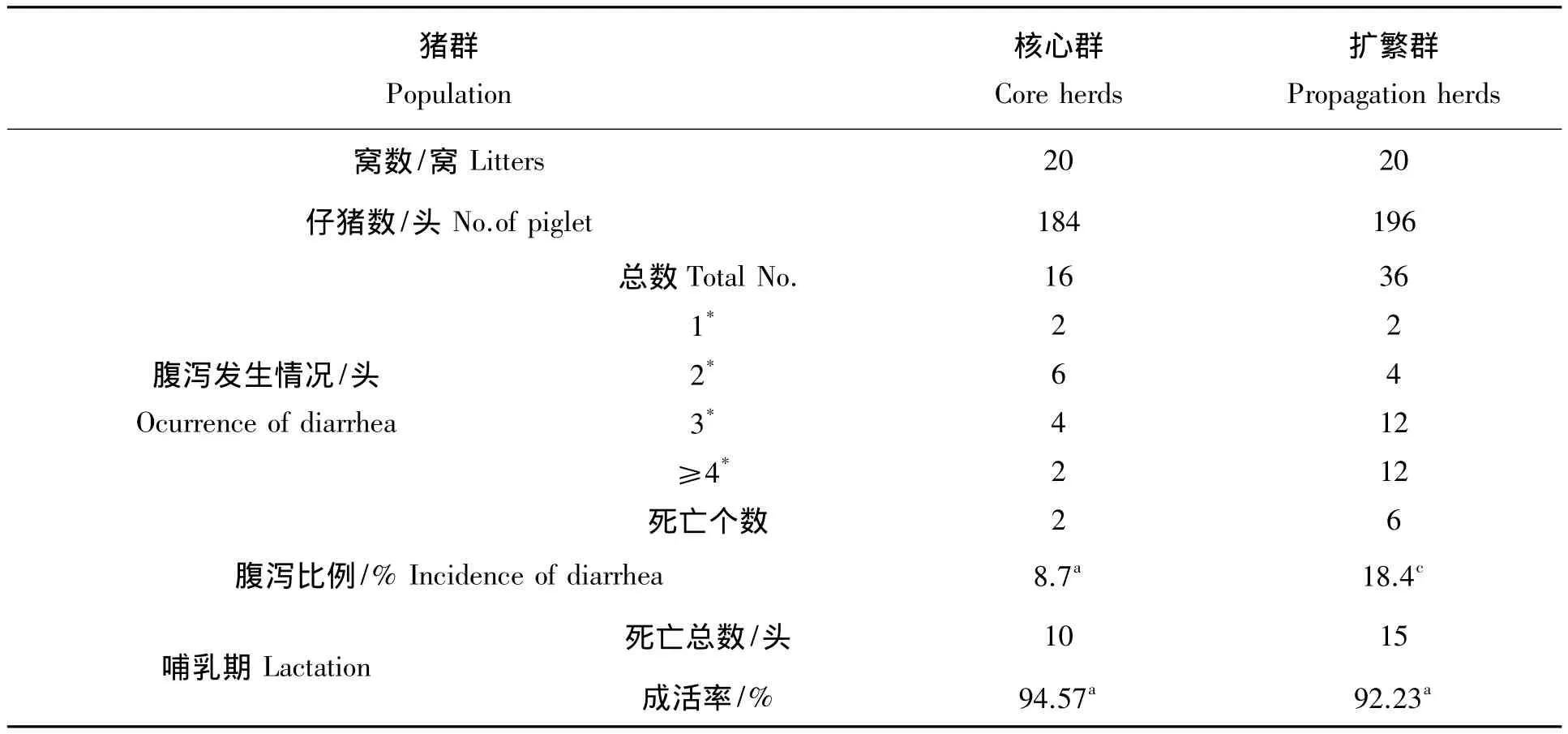
表2 育种核心群与普通扩繁群仔猪腹泻比例Tab.2 Diarrhea rate of piglets in the core breeding group and propagation population
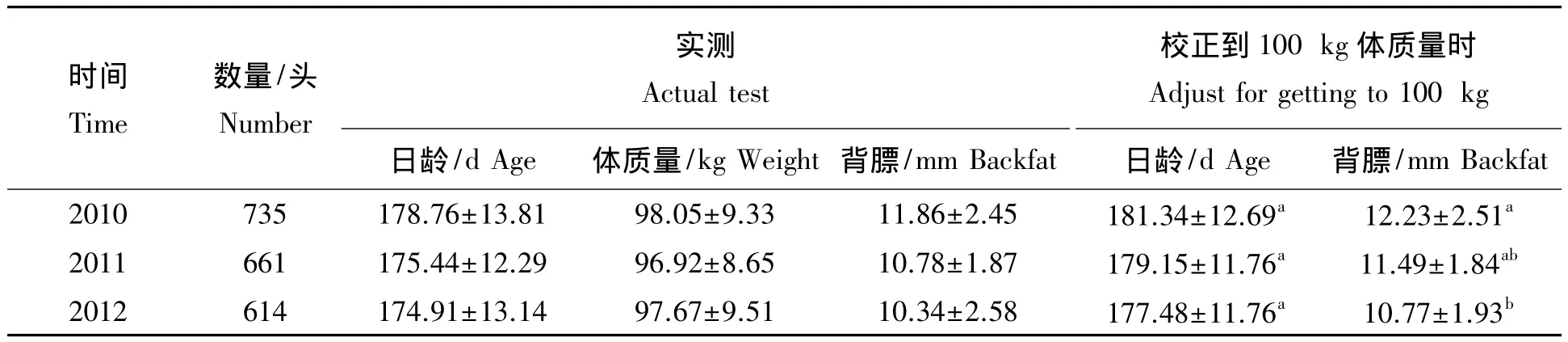
表3 育种群3个世代生长性能Tab.3 Growth performance in the three generations of breeding pigs
2.4 MUC13基因型与生长性能的关联性
从表4发现,易感纯合基因型AA猪只达100 kg体质量日龄较抗性纯合基因型GG猪只平均增加1.56 d,背膘厚也平均增加0.36 mm;易感杂合基因型GA猪只达100 kg体质量日龄较抗性纯合基因型GG猪只平均增加1.34 d,背膘厚也平均增加0.27 mm。但经SPSS 13.0统计分析,均未达到显著水平(P>0.05),说明MUC13各基因型猪只间在生长速度和活体背膘上没有显著差别。
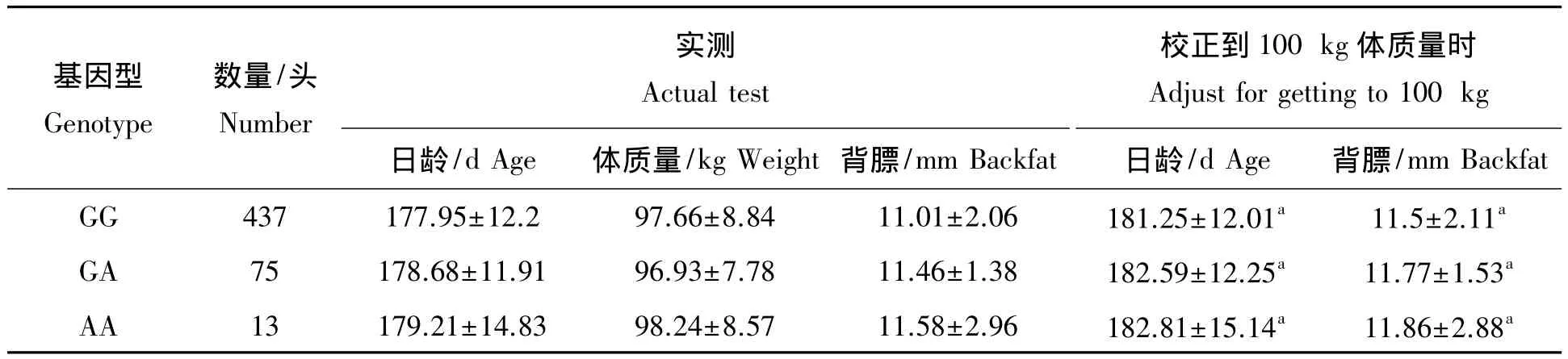
表4 不同基因型猪群生长性能Tab.4 Growth performance in different genotyping patterns of pigs
3 讨论与结论
3.1 MUC13抗性基因型纯合群体的选育
通过连续3个世代的MUC13基因检测筛选以及性能测定后的BLUP指数选择,受试育种群MUC13抗腹泻有利基因G频率由0.86提高到1,有利基因型GG频率由0.75提高到1,实现了抗腹泻有利基因的纯合。同时,生长速度和活体背膘均有一定改善,校正达100 kg体质量日龄由181.34 d降低到177.48 d,减少3.86 d,不过仍未达显著水平;但活体背膘则由12.23 mm降低到10.77 mm,减少1.46 mm,且达到显著水平(P<0.05)。经分析,由于场内测定受实际生产时间影响,猪群入测时间达6个月,且测定个体表现也有较大差异,虽然BLUP模型计算选择指数时能够校正场、年、季等造成的误差,但进行SPSS统计分析时仍有一定影响,为此,在后续的选育测定工作中,应将核心群母猪配种安排在尽可能短时间完成,以减少因时间跨度长而造成的影响。此外,选育群体要能维持腹泻抗性基因的纯合,首先应该坚持在本群体内选留后备种猪;如需要引进血缘时,必须对引进个体进行MUC13基因检测,防止导入易感等位基因。
3.2 MUC13抗性纯合群体与哺乳仔猪腹泻的关系
造成哺乳仔猪腹泻的因素众多,肠毒素大肠杆菌则是引起仔猪腹泻的主要因素。通过MUC13基因型判定与肠毒素大肠杆菌黏附的受体有无,从而确定仔猪对大肠杆菌引起腹泻的抗性或易感,已由江西农大团队相继研发和验证[9-12,19-20],并获得国家发明专利(专利号:200810136425)。经普查,杜洛克群体自然选择情况下,MUC13抗性纯合程度已达70%~80%,因而,本项目通过对育种核心群与未经选育的扩繁群仔猪在相同胎次和饲养管理情况下的腹泻情况观察、统计,发现核心群与扩繁群断奶前仔猪腹泻发生率分别为8.7%和18.4%,经SPSS 13.0 Chi-square test分析,差异极显著(P<0.01);核心群与扩繁群哺乳仔猪断奶成活率分别为94.6%和92.3%,提高2.3个百分点,但经统计分析,未达显著水平。本次测试核心群和扩繁群数量均仅20窝,所得结果虽有一定参考价值,但有待于在更大范围进行试验、统计,以进一步验证MUC13抗性纯合与断奶前仔猪腹泻的关系。
3.3 MUC13抗性纯合基因型与猪生产性能的关系
MUC13抗性纯合基因型能减少仔猪腹泻的发生,但抗性个体的纯合对其他生产性能是否具有不利影响,此前尚未见报道。本研究通过对不同基因型的个体进行生长发育性能测定,发现抗性纯合基因型(GG)、杂合基因型(GA)及易感纯合基因型(AA)个体达100 kg体质量日龄平均分别为181.25,182.59,182.81 d,达100 kg体质量校正背膘分别为11.50,11.77,11.86 mm,经SPSS 13.0统计分析,各组间差异均不显著(P>0.05),说明MUC13基因型抗性纯合选育对生长速度和背膘厚性能没有影响。
通过3年的分子检测与性能测定综合选育研究,验证了MUC13基因抗性纯合有利于降低断奶前仔猪腹泻的发生;生长发育性能测定及BLUP指数评估能有效降低活体背膘;而且MUC13不同基因型选择对生长发育没有明显影响。
致谢:感谢江西农业大学任军研究员对基因检测实验的指导和论文修改。
[1]王明福,彭羽,肖光明.论溶菌酶防治仔猪大肠杆菌性腹泻推广应用前景[J].中国畜禽种业,2009(3):132-135.
[2]Guerin G,Duval-Iflah Y,Bonnean M,et al.Evidence for linkage between k88ab,k88ac intestinal receptors to Escherichia coli and transferring loci in pigs[J].Anim Genet,1993,24:393-396.
[3]Moon H W,Hoffman.Prevalence of some virulence genes among Escherichia coli isolates from swine presented to a diagnostic laboratory in Iowa[J].J Vet Diagn Invest,1999,11:557-560.
[4]陈螺眉,郭金森,黄瑜,等.福州地区哺乳仔猪腹泻防制研究Ⅱ、致病性大肠杆菌的分离及血清型鉴定[J].福建畜牧兽医,2002(6):8.
[5]Guinée P A M,Jansen W H.Behavior of Escherichia coli antigens K88ab,K88ac,and K88ad in immunoelectrophoresis,double diffusion,and hemagglutination[J].Infect Immun,1979,23:700-705.
[6]Sellwood R,Gibbons R A.Adhesion of enteropathogenic Escherichia coli to pig intestinal brush borders:the existence of two pig phenotypes[J].J Med Microbiol,1975,8:405-411.
[7]Gibbons R A,Sellwood R,Burrows,et al.Inheritance of resistance to neonatal E.coli iarrhea in the pig:examination of the genetic system[J].Theor Appl Genet,1977,51:65-70.
[8]Joller D,Jørgensen C B,Bertschinger H U,et al.Refined localization of the Escherichia coli F4ab/F4ac receptor locus on pig chromosome 13[J].Anim Genet,2009,40:749-752.
[9]Peng Q L,Ren J,Yan X M,et al.The g.243A>G mutation in intron 17 of MUC4 is significantly associated with susceptibility/resistance to ETEC F4ab/ac infections in pigs[J].Anim Genet,2007,38:397-400.
[10]Wang Y,Ren J,Lan L,et al.Characterization of polymorphisms of ransferring receptor and their association with susceptibility to ETEC F4ab/ac in pigs[J].J Anim Breed Genet,2007,124:225-229.
[11]Zhang B,Ren J,Yan X,et al.Investigation of the porcine MUC13 gene:isolation,expression,polymorphisms and strong association with susceptibility to enterotoxigenic Escherichia coli F4ab/ac[J],Animal Genetics,2008,39:258-266.
[12]Ren J,Tang H,Yan X M,et al.A pig-human comparative RH map comprising 20 genes on pig chromosome 13q41 that harbours the ETEC F4ac receptor locus[J].Journal of Animal Breeding and Genetics,2009,126:30-36.
[13]刘敬顺,刘小红.全自动种猪性能测定系统(F.I.R.E)在猪育种中的研究和应用[J].今日养猪业,2004(3):72-74.
[14]肖炜.猪育种新技术及其应用现状[J].猪业科学,2006,10:12-15.
[15]Rothschild M F.Genetic and genomic technologies from A-Z,Pig Genetics Workshop[G].2010:25-38.
[16]孙德林,云鹏.2011年中国养猪业发展研究报告[R].中国种猪信息网,2012-09-26.
[17]阮国荣,肖石军,徐盼,等.福建省商业猪种MUC13、IGF2、和RYR1基因主效位点的遗传变异分析[J].江西农业大学学报,2012,34(5):997-1002.
[18]Ruan G R,Xing Y Y,Fan Y,et al.Genetic variation at RYR1,IGF2,FUT1,MUC13,and KPL2 mutations affecting production traits in Chinese commercial pig breeds[J],Czech J Anim Sci,2013,58(2):65-70.
[19]晏学明.猪肠毒素大肠杆菌F4流行病学调查及其受体的遗传学研究[D].南昌:江西农业大学,2008.
[20]Ren J,Yan X,Ai H,et al.Susceptibility towards enterotoxigenic Escherichia coli F4ac diarrhea is governed by the MUC13 gene in pigs[J].PLoS One,2012,7:e44573.
