拟南芥成花关键基因调控网络研究进展
2014-03-22李敬谷慧英王志敏汤青林宋明
李敬 谷慧英 王志敏 汤青林 宋明
(西南大学园艺园林学院 南方山地园艺学教育部重点实验室 重庆市蔬菜学重点实验室,重庆 400715)
拟南芥成花关键基因调控网络研究进展
李敬 谷慧英 王志敏 汤青林 宋明
(西南大学园艺园林学院 南方山地园艺学教育部重点实验室 重庆市蔬菜学重点实验室,重庆 400715)
开花是植物从营养生长转变为生殖生长的重要时期,而开花调控成为近年来植物分子生物学研究的热点。在目前已有的研究中,调控拟南芥开花的基因网络已经发展成一个包含串扰(Crosstalk)、反馈(Feedback)和冗余(Redundancy)的复杂网络,这个网络通过开花整合子来与其他发育过程紧密结合。以调节开花的遗传途径作为基础,重点讨论了顶端分生组织中的信号积累、花发育的时空调节、开花相关基因在拟南芥开花时间或花发育过程以外的其他过程中的功能,并对开花调控网络的深入研究进行了展望。
开花基因调控网络 开花整合子 信号积累 花发育 时空调节
目前,大量基于基因组数据的转录因子靶点定位研究使人们对开花基因网络调控植物发育过渡过程有了新的认识,特别是在顶芽分生组织成花转变的时空动力学(Spatial and temporal dynamics)研究方面有很大进步。单一转录因子以异源二聚体模式促进特定靶基因的表达,同时抑制其他非目的基因的表达,这有力地证明了特定的转录因子通过整合不同的调控过程来协调复杂的发育过渡过程这一理论[1,2]。
目前利用染色体免疫共沉淀法(Chromatin immunoprecipitation,ChIP)探究体内转录因子定位(Transcription factor target mapping)的研究从根本上增加了基因调控网络(Genetic regulatory networks,GRNs)的复杂性,而且已有关于开花调控途径的综述见刊[3,4]。因此应以调节开花的遗传途径为基础,将研究重点放在顶端分生组织(Shoot apical meristems,SAM)中的信号积累、花发育的时空调节、开花相关基因在拟南芥开花时间或花发育过程以外
的其他过程中的功能上。
1 开花整合子
一些对内源信号(赤霉素、自主性和苗龄)和环境信号(光照和温度)有响应的信号转导途径都会聚在一些特定基因上,这些基因会激活花的同源异型基因(图1)。开花整合子通过整合来自不同开花途径的信号,如光周期途径(Photoperiod pathway)、春化途径(Vernalization pathway)、自主途径(Autonomous pathway)、赤霉素途径(Gibberellins pathway)等,进而精确调控花分生组织,从而控制拟南芥的开花过程[5]。开花整合子包括移动信号Flowering locus T(FT),有研究表明,FT 作为长距离运输的开花素信号分子,通过韧皮部从叶片移动到茎端[6,7]。此外,番茄中FT的同源基因Singleflower truss(SFT)能诱导光周期不敏感的番茄和烟草开花。SFT在番茄叶片中表达,而SFT蛋白具有移动性,是开花素信号分子。Eliezer等[8]的试验表明,嫁接传递的SFT信号可以弥补sft突变体的所有发育缺陷,该信号代替了短日照和长日照开花刺激。

图1 开花过渡前的基因调控[3]
FT与特异分生组织bZIP类转录因子Flowering loocus D(FD)共同促进开花,FD在顶端分生组织中表达,而FT在韧皮部表达。此外,FT在顶端分生组织中的异位表达可以挽救ft突变体的表型,这表明FT在茎尖也发挥作用[9]。FT-FD复合物可以激活茎端分生组织中花分生组织基因(如AP1、FUL、CAL)的表达,从而促进成花转变并启动花发育过程[10,11]。
FT作为光周期途径中转录调控因子Constans(CO)的直接靶基因,其在叶片和维管组织中特异表达[10,12]。自主途径,温度,春化和赤霉素(GA)途径均引起FT的上调表达,该上调作用是通过抑制FLOWERING LOCUS C-SHORT VEGETATIVE PHASE(FLC-SVP)复合体的阻遏作用来实现的。在顶端分生组织中,FT-FD复合体激活MADS区域转录因子SUPRESSOR OF OVEREXPRESSION OF CONSTANS1(SOC1),它存在于含AGAMOUS-LIKE 24(AGL24)蛋白的复合体中,这个复合体促进花分生组织决定基因LEAFY(LFY)的表达,并通过LFY促进APETALA1(AP1)表达,而AP1也是FT-FD复合体的直接靶位点。
LFY基因在拟南芥花发育过程中起重要作用,它既是开花时间基因,又是花分生组织基因,LFY不受CO的直接调节,但赤霉素(GAs)可以通过不同于对长日照产生应答的顺式作用元件激活LFY基因的表达,这说明控制开花的环境信号和内在信号可以在LFY启动子上整合[13]。LFY的表达发生在成花转变之前,最早可在幼叶原基中检测到,当花序分生组织出现以后,其表达逐渐增强,LFY mRNA在花序和嫩花中积累的量达到峰值[14]。
光周期途径中CO能够促进SOC1的表达,RNA表达分析表明,CO及FT的过量表达能够诱导激活SOC1[15-18],而且SOC1的表达还受春化途径、自主途径和赤霉素途径信号的正调控。soc1突变体在长日照和短日照下都延迟开花,表明SOC1的失活能抑制各种开花促进途径的信号,而SOC1的过量表达能引起早花,并且还能逆转自主途径和光周期途径基因的突变效果。SOC1主要在叶片和茎尖表达,并且其表达量随着发育过程而提高,在成花转变过程中,它的表达会在茎尖急速增加[19,20]。除了具有开花整合子的功能,SOC1还调节花模式和花分生组织特性[21-24]。另外,SVP蛋白通过结合FT和SOC1
的CArG基序调控其表达[25,26]。SVP蛋白还与FLC蛋白在春化途径中相互作用[26],但svp-32和flc-3的突变体对环境温度的响应是不同的[25],这表明,通过SVP的环境温度的响应与春化响应不同。由于转录因子往往调节多个目标,SVP蛋白可能在环境温度信号中有非单一的靶标。
2 花发育的时空调节
在经典ABC模型中每轮花器官的产生是3类器官特征基因A、B和C不同组合表达的结果。在此模型中,基因A单独表达形成萼片(Sepals);基因A和B都表达形成花瓣(Petale);基因B和C表达形成雄蕊(Stamen);基因C单独表达形成心皮(Carpels)[27](图2)。

图2 ABC模型
随着D功能基因(FBP11)被发现,经典ABC模型从而扩展为ABCD模型,而E功能基因(AGL2-like)的发现,使ABCD模型又进一步扩展为ABCDE模型。至今只在矮牵牛中发现D功能基因,序列相似性分析表明拟南芥的D功能基因是AGL11。FBP11和AGL11都是MADS-box基因,和AG的亲缘关系较近,有相似的基因表达模式[28,29]。
当开花诱导发生时,营养分生组织(Vegetative meristem,VM)首先出现花序分生组织(Inflorescence meristem,IM)的特征,然后花序分生组织会引起生殖器官产生花分生组织(Floral meristem,FM)。当信号从促进开花的途径积累到营养分生组织的时候,这些特征就开始发生变化。在拟南芥中,花序分生组织仍然是不确定的,因为FT的同源基因TERMINAL FLOWER1(TFL1)拮抗LFY和AP1的表达[30,31](图1)。
在花分生组织中发生的发育的变化被编码在ABCE模型中,该模型假设,4大调节功能(A、B、C 和 E)为相应的器官在每一轮的形成组合地发挥作用[32,33](图2)。A类蛋白AP1和AP2在第1轮形成萼片的特征,它们的活性与B类蛋白AP3和PISTILLATA(PI)在第2轮重叠,形成花瓣特征。在第3轮形成雄蕊特征,因为合并了B类蛋白和C类蛋白AGAMOUS(AG)的活性,后者在第4轮确定心皮发育。在所有4个轮中,E类蛋白SEPALLATA1-4(SEP1-4)作为共同调节因子发挥了至关重要的作用。
最近,应用转录因子结合试验探究这个开花调控网络的研究已经明确了顶端分生组织中的基因相互作用,花分生组织(FM)的形成是从植物特有的转录因子LFY和AP1的上调开始的[34](图3):在花序分生组织(IM)侧面的花原基中的LFY的明显上调是VM-IM过渡的开始,而且SOC1-AGL24复合体直接促进LFY表达[35],同时SPL3[36]和GAMYB33也促进其表达[37]。有研究者提出,SOC1-AGL24二聚体与一个由SHOOT MERSTEMLESS(STM)、PENNYWISE(PNY)和POUND-FOOLISH(PNF)组成的异源二聚体一起激活LFY表达[38,39]。因为FT促进SOC1,所以LFY的表达也间接被FT上调[18]。随后,AP1和LFY在一个正反馈回路中彼此上调,其功能是保持花分生组织的特征。FT-FD和STM-PNY/ PNF,以及SPL3和SPL9也引起AP1的表达[36,39,40](图 1)。
在花发育的第1阶段和第2阶段,原基无分化增殖[41]。在A类和E类基因的共同作用下,随着萼片的形成,分化在第3阶段开始发生,而分化在第1阶段未发生是通过保持SEP3沉默实现的。开花时间转录因子 SVP、SOC1和AGL24共同下调SEP3的表达(图3),这3个转录因子中,SVP是在花发育的第1阶段或第2阶段最强烈地表达[22],而SOC1和AGL24的表达在这些阶段不易被检测到[22,42],不过,遗传分析清楚地表明,SOC1和AGL24对最幼嫩花蕾中的SEP3起到了抑制作用,而且SEP3的异
位表达仅仅出现在agl24-1 svp-41 双突变体和soc1-2 agl24-1 svp-41 三突变体中[24]。SVP与TERMINAL FLOWER2/LIKE HETEROCHROMATIN PROTEIN1相互作用,间接引起组蛋白H3赖氨酸27(Histone H3 lysine 27)的三甲基化。而SOC1和AGL24与SAP18(Sin3/组蛋白脱乙酰基酶复合物的组成部分)相互作用来阻止组蛋白H3乙酰化,这种乙酰化是活化转录的标记[24,43]。但B类和C类基因不仅被SEP3调控,相反,异源二聚体AP1-AGL24和AP1-SVP会抑制其早期表达,而且这两个异源二聚体与SEUSS-LEUNIG(SEU-LUG)复合体共同调节[44](图3)。随后,在第3阶段早期,AP1直接或通过LFY间接促进SEP3的表达[45,46](图4)。

图3 花分生组织(FM)的1阶段和2阶段的基因调控[3,67]
现在有很多关于花同源异型转录因子负反馈到开花时间整合子的证据,这个反馈确保一个快速经过VM,IM和FM变化的过程,同时也防止开花逆转。AP1直接抑制SVP、SOC1和AGL24[22],这形成了一个交替的、间接的促进SEP3表达的AP1模式(图4)。AP1也直接抑制另外两种开花整合基因FD和它的旁系同源基因FDP[45],从而确保花器官形态发生的急剧转变和增强。
一旦SEP3表达,它与LFY一起激活B类(AP3和PI)和C类(AG)基因。此外,SEP3和LFY通过上调AP1产生一个正反馈回路[31,47],在这个回路中,SEP3还直接下调SOC1和SVP[47](图4)。由于SEP3-SOC1和SEP3-SVP异源二聚体可以形成,这些开花时间转录因子有助于它们在花分生组织(FM)中的自动负调控[48]。

图4 花分生组织(FM)在第3阶段早期的基因调控
SEP3诱导导致SEP3-AP1异源二聚体的形成,该异源二聚体与SEU-LUG复合体共同抑制AG在外两轮中的表达[49]。这种抑制作用在AP1和AG之间是双向的,由于AG的抑制,AP1在第3阶段从花的中心消失[50],此时AG是与SEP3形成复合体发挥作用[47,48](图5)。然而,AG与另一个A类基因AP2之间的关系是单向的,AP2通过直接结合到AG第2个内含子上来下调分生组织中的AG[51,52],尽管AG在转录水平上没有拮抗AP2(图5)。通过mRNA的裂解和翻译抑制,AP2的表达被miR172调节,由于AP2自身表达上的强烈负反馈和相关的AP2类 miR172靶点的强烈负反馈,目前尚难评估该调节的精确的生物重要性[53-55](图5)。原位杂交(In situ hybridization,ISH) 显 示,AP2和miR172的表达是相辅相成的,从第2阶段开始,AP2在外轮,miR172在内轮,类似AG的表达模式[56]。然而,AP2和miR172在第3轮中花被和生殖器官之间的边界有一些重叠,这符合miR172不能充分抑制AP2的情况[53-56],通过SEU-LUG共抑制复合体的作用,AP2在外轮保持其特定的功能而直接下调miR172的表达[52,57](图5)。另外,miR172表达的变化会导致SCHLAFMÜTZE(SMZ),TARGET OF EAT1(TOE1),
和TARGET OF EAT1(TOE2)[58]的差异表达,环境温度的变化引起的miR172及其靶基因之间的负相关表达模式与这些靶基因在控制开花时间中的作用是一致的[52,59]。
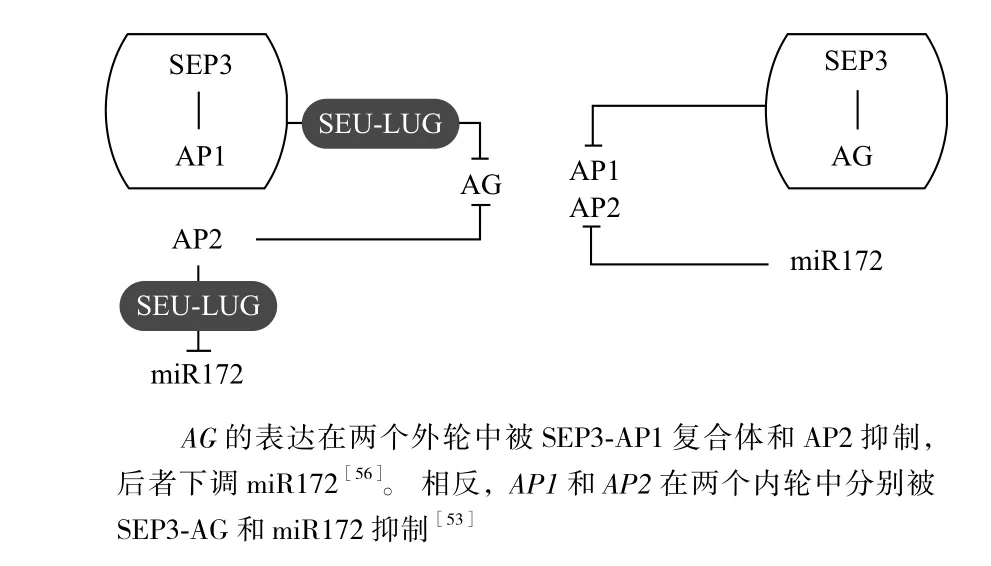
图5 C类基因(AG)与A类基因(AP1、AP2)的相互调控
3 日益复杂的开花基因调控网络
最近转录因子目标定位研究发现:AP2本身作为一个双功能的转录因子,直接抑制其阻遏物MIR172b在一个反馈回路中的表达,并通过直接激活MIR156e(MIR172b的间接阻遏物)的表达来加强这个回路。AP2也通过直接诱导另一个开花阻遏物AGL15的表达来增强它自身的功能,同时直接抑制开花激活子SOC1和FRUITFUL(FUL)[52]。AP1直接抑制开花抑制子TFL1,而直接激活LFY和花的同源异型基因AP2,AP3和SEP3[45]。当LFY直接激活4个轮中的同源异型基因的时候,它也直接抑制TFL1的表达[46]。最后,SEP3表现双功能,抑制开花时间基因SVP和SOC1,而促进花器官特征基因AP1,AP3和AG的表达[47]。虽然其根本机制尚不清楚,但可以假设,共抑制或共激活复合物通过动态组合管理这些功能开关,如AP1[41],FT-FDTFL1[60]和SEU-LUG-AP2复合物[52,57]。
最近的染色质免疫共沉淀测序(ChIP-seq)研究表明,转录因子不但可以直接针对许多位点,还可以通过激活或抑制不同的相关基因来直接促进特征器官的发育。这些基因依赖特殊的发育背景、外源性信号,或者组织类型。区分这些功能转变的调节基础是目前正在进行的一个工作重点,从而明确复杂的发育过程是如何运行的。
4 开花相关基因的新功能
最近基于基因组数据的转录因子靶点定位研究发现,开花转录因子在不同于开花时间和花发育的过程中发挥功能。事实上单个的转录因子参与多种发育过程的相互作用,而且通常被认为是不相关的流程现在却是直接联系的。
两个深入的开花整合研究证实了FLC和LFY在开花以外的新作用,它们是拟南芥响应春化作用的主要调节因子。FLC也具有双功能,它通过直接抑制FT,SOC1和SEP3的表达来抑制开花,但是FLC也促进开花阻遏物SMZ和TOE3的表达。FLC对SOC1和FT表达的抑制是通过与SOC1启动子区直接作用以及与FT的第1个内含子中的CArG盒作用而实现的[61]。另外,FLC还直接调节从幼年到成年阶段变化的基因,如SPL15[62]。而且在FLC位点的自然变异可以通过调控开花时间的遗传途径来调控与温度相关的发芽[63],这一点说明FLC是拟南芥生命周期中普遍的过程整合子。同时,不同的非编码RNA调控FLC的表达,这与染色质重塑的动态性相关[64-66]。
最明显的流程整合例子是LFY靶点的全基因组定位[67],虽然许多研究通过转录因子目标定位已经有效地解释了基因多效性,但有研究发现,LFY通过抗性反应来协调开花过渡,引导资源流向花和果实的发育,从而保证植物生殖健康[67]。LFY直接绑定到微生物相关分子模式(Microbe-associated molecular pattern,MAMP)识别受体FLAGELLINSENSITIVE 2(FLS2)和ABC转运体 PLEIOTROPIC DRUG RESISTANCE 8(PDR8/PEN3)的启动子上,并调控它们的mRNA表达,这些基因产物是植物的基础免疫反应途径的关键。通过测试lfy突变体对鞭毛蛋白衍生肽(Flagellin-derived peptide flg22)的响应进行功能验证,结果表明相对于野生型,lfy突变体表现出一个明显的flg22诱导的胼胝质沉积数量的增加[67]。
5 展望
拟南芥开花调控网络研究的下一阶段将是评估多个转录因子在几个发育阶段中的作用和明确早期花原基的区域。ChIP-seq的应用表明,科学技
术的进一步发展能实现更精细的研究基因调控网络(GRN)的结构。因此,需要先进的细胞分选技术的持续发展来确定每一个转录因子的结合位点和在一个特定细胞子集和发育阶段中的靶点表达谱。激光捕获显微切割(LCM)的应用,以及在体内标记并分离特殊细胞类型的方法的应用[68],再结合RNAseq等技术,就可以在严格定义的细胞群中进行基因表达变化的精确测定。随着更深入的研究,可以期望观察到每个转录因子与一个相同的靶点的动态的、可变的作用,并突出辅助因子参与的重要性。
[1]Niu W, Lu ZJ, Zhong M, et al. Diverse transcription factor binding features revealed by genome-wide ChIP-seq in C. elegans[J]. Genome Research, 2011, 21:245-254.
[2]Redestig H, Weicht D, Selbig J, Hannah MA. Transcription factor target prediction using multiple short expression time series from Arabidopsis thaliana[J]. BMC Bioinformatics, 2007, 8:454.
[3]Srikanth A, Schmid M . Regulation of flowering time:all roads lead to Rome[J]. Cell Mol Life Sci, 2011, 68:2013-2037.
[4]Michaels SD . Flowering time regulation produces much fruit[J]. Curr Opin Plant Biol, 2009, 12:75-80.
[5]Putterill J, Laurie R, Macknight R. It’s time to flower:the genetic control of flowering time[J]. Bioessays, 2004, 26(4):363-373.
[6]Corbesier L, Vincent C, Jang S, et al. FT protein movement contributes to long-distance signaling in floral induction of Arabidopsis[J]. Science, 2007, 316:1030-1033.
[7]Jaeger KE, Wigge PA . FT protein acts as a long-range signal in Arabidopsis[J]. Curr Biol, 2007, 17:1050-1054.
[8]Lifschitz E, Eviatar T, Rozman A, et al. The tomato FT ortholog triggers systemic signals that regulate growth and flowering and substitute for diverse environmental stimuli[J]. PANS, 2006, 103(16):6398-6403.
[9]Imaizumi T, Kay SA. Photoperiodic control of flowering:not only by coincidence[J]. Trends in Plant Science, 2006, 11(11):550-558.
[10]Abe M, Kobayashi Y, Yamamoto S, et al. FD, a bZIP protein mediating signals from the floral pathway integrator FT at the shoot apex[J]. Science, 2005, 309:1052-1056.
[11]Wigge PA, Kim MC, Jaeger KE, et al. Integration of spatial and temporal information during floral induction in Arabidopsis[J]. Science, 2005, 309:1056-1059.
[12]Kardailsky I, Shukla VK, Ahn JH, et al. Activation tagging of the floral inducer FT[J]. Science, 1999, 286:1962-1965.
[13]Blazquez MA, Weigel D. Integration of floral inductive signals in Arabidopsis[J]. Nature, 2000, 404(6780):889-892.
[14]Blazquez MA, Soowal LN, Lee I, Weigel D. LEAFY expression and flower initiation in Arabidopsis[J]. Development, 1997, 124(19):3835-3844.
[15]Michaels SD, Himelblau E, Kim SY, et al. Integration of flowering signals in winter-annual Arabidopsis[J]. Plant Physiol, 2005, 137(1):149-156.
[16]Moon J, Lee H, Kim M, Lee I. Analysis of flowering pathway integrators in Arabidopsis[J]. Plant Cell Physiol, 2005, 46:292-299.
[17]Samach A, Onouchi H, Gold SE, et al. Distinct roles of CONSTANS target genes in reproductive development of Arabidopsis[J]. Science, 2000, 288:1613-1616.
[18]Yoo SK, Chung KS, Kim J, et al. CONSTANS activates SUPPRESSOR OF OVEREXPRESSION OF CONSTANS 1 through FLOWERING LOCUS T to promote flowering in Arabidopsis[J]. Plant Physiol 2005, 139:770-778.
[19]Borner R, Kampmann G, Chandler J, et al. MADS domain gene involved in the transition to flowering in Arabidopsis[J]. Plant J, 2000, 24:591-599.
[20]Lee H, Suh SS, Park E, et al.The AGAMOUS-LIKE 20 MADS domain protein integrates floral inductive pathways in Arabidopsis[J]. Genes Dev, 2000, 14:2366-2376.
[21]Lee J, Lee I. Regulation and function of SOC1, a flowering pathway integrator[J]. J Exp Bot, 2010, 61:2247-2254.
[22]Liu C, Zhou J, Bracha-Drori K, et al. Specification of Arabidopsis floral meristem identity by repression of flowering time genes[J]. Development, 2007, 134:1901-1910.
[23]Melzer S, Lens F, Gennen J, et al. Flowering-time genes modulate meristem determinacy and growth form in Arabidopsis thaliana[J]. Nat Genet, 2008, 40:1489-1492.
[24]Liu C, Xi W, Shen L, et al. Regulation of floral patterning by flowering time genes[J]. Dev Cell, 2009, 16:711-722.
[25]Lee JH, Yoo SJ, Park SH, et al. Role of SVP in the control of flowering time by ambient temperature in Arabidopsis[J]. Genes
Dev, 2007, 21(4):397-402.
[26]Li D. A repressor complex governs the integration of flowering signals in Arabidopsis[J]. Dev Cell, 2008, 15:110-120.
[27]刘建武, 孙成华, 刘宁. 花器官决定的ABC模型和四因子模型[J]. 植物学通报, 2004, 3:346-351.
[28]Theissen G, Becker A, Rosa AD, et al. A short history of MADS-box genes in plants[J]. Plant Mol Biol, 2000, 42:115-149.
[29]Theissen G . Development of floral organ identity:stories from the MADS house[J]. Curr Opin Plant Biol, 2001, 4:75-85.
[30]Ratcliffe OJ, Bradley DJ, Coen ES . Separation of shoot and floral identity in Arabidopsis[J]. Development, 1999, 126:1109-1120.
[31]Liljegren SJ, Gustafson-Brown C, Pinyopich A, et al. Interactions among APETALA1, LEAFY, and TERMINAL FLOWER1 specify meristem fate[J]. Plant Cell, 1999, 11:1007-1018.
[32]Theissen G, Saedler H. Plant biology Floral quartets[J]. Nature, 2001, 409:469-471.
[33]Krizek BA, Fletcher JC. Molecular mechanisms of flower development:an armchair guide[J]. Nat Rev Genet, 2005, 6:688-698.
[34]Benlloch R, Berbel A, Serrano-Mislata A, Madueno F. Floral initiation and in florescence architecture:a comparative view[J]. Annals Bot, 2007, 100:659-676.
[35]Lee J, Oh M, Park H, Lee I. SOC1 translocated to the nucleus by interaction with AGL24 directly regulates LEAFY[J]. Plant J, 2008, 55:832-843.
[36]Yamaguchi A, Wu MF, Yang L, et al. The microRNA-regulated SBP-Box transcription factor SPL3 is a direct upstream activator of LEAFY, FRUITFULL, and APETALA1[J]. Dev Cell, 2009, 17:268-278.
[37]Gocal GF, Sheldon CC, Gubler F, et al. GAMYB-like genes, flowering, and gibberellin signaling in Arabidopsis[J]. Plant Physiol, 2001, 127:1682-1693.
[38]Kanrar S, Bhattacharya M, Arthur B, et al. Regulatory networks that function to specify flower meristems require the function of homeobox genes PENNYWISE and POUND-FOOLISH in Arabidopsis[J]. Plant J, 2008, 54:924-937.
[39]Smith HMS, Ung N, Lal S, Courtier J. Specification of reproductive meristems requires the combined function of SHOOT MERISTEMLESS and floral integrators FLOWERING LOCUS T and FD during Arabidopsis inflorescence development[J]. J Exp Bot, 2011, 62: 583-593.
[40]Wang JW, Czech B, Weigel D. miR156-regulated SPL transcription factors define an endogenous flowering pathway in Arabidopsis thaliana[J]. Cell, 2009, 138:738-749.
[41]Smyth DR, Bowman JL, Meyerowitz EM. Early flower development in Arabidopsis[J]. Plant Cell, 1990, 2:755-767.
[42]Yu H, Ito T, Wellmer F, Meyerowitz EM. Repression of AGAMOUSLIKE 24 is a crucial step in promoting flower development[J]. Nat Genet, 2004, 36:157-161.
[43]Li B, Carey M, Workman JL. The role of chromatin during transcription[J]. Cell, 2007, 128:707-719.
[44]Gregis V, Sessa A, Dorca-Fornell C, Kater M. The Arabidopsis floral meristem identity genes AP1, AGL24 and SVP directly repress class B and C floral homeotic genes[J]. Plant J, 2009, 60:626-637.
[45]Kaufmann K, Wellmer F, Muino JM, et al. Orchestration of floral initiation by APETALA1[J]. Science, 2010, 328:85-89.
[46]Moyroud E, Minguet EG, Ott F, et al. Prediction of regulatory interactions from genome sequences using a biophysical model for the Arabidopsis LEAFY transcription factor[J]. Plant Cell, 2011, 23:1293-1306.
[47]Kaufmann K, Muino JM, Jauregui R, et al. Target genes of the MADS transcription factor SEPALLATA3:integration of developmental and hormonal pathways in the Arabidopsis flower[J]. PLoS Biol, 2009, 7:e1000090.
[48]Folter S, Immink RGH, Kieffer M, et al. Comprehensive interaction map of the Arabidopsis MADS Box transcription factors[J]. Plant Cell, 2005, 17:1424-1433.
[49]Sridhar VV, Surendrarao A, Liu Z. APETALA1:SEPALLATA3 interact with SEUSS to mediate transcription repression during flower development[J]. Development, 2006, 133:3159-3166.
[50]Gustafson-Brown C, Savidge B, Yanofsky MF. Regulation of the Arabidopsis floral homeotic gene APETALA1[J]. Cell, 1994, 76:131-143.
[51]Bomblies K, Dagenais N, Weigel D. Redundant enhancers mediate transcriptional repression of AGAMOUS by APETALA2[J]. Dev Biol, 1999, 216:260-264.
[52]Yant L, Mathieu J, Dinh TT, et al. Orchestration of the floral transition and floral development in Arabidopsis by the bifunctional transcription factor APETALA2[J]. Plant Cell, 2010, 22:2156-
217 0.
[53]Aukerman MJ, Sakai H. Regulation of flowering time and floral organ identity by a microRNA and its APETALA2-like target genes[J]. Plant Cell, 2003, 15:2730-2741.
[54]Chen X. A microRNA as a translational repressor of APETALA2 in Arabidopsis flower development[J]. Science, 2004, 303:2022-2025.
[55]Schwab R, Palatnik JF, Riester M, et al. Specific effects of microRNAs on the plant transcriptome[J]. Dev Cell, 2005, 8:517-527.
[56]Wollmann H, Mica E, Todesco M, et al. On reconciling the interactions between APETALA2, miR172 and AGAMOUS with the ABC model of flower development[J]. Development, 2010, 137:3633-3642.
[57]Grigorova B, Mara C, Hollender C, et al. LEUNIG and SEUSS corepressors regulate miR172 expression in Arabidopsis flowers[J]. Development, 2011, 138:2451-2456.
[58]Lee H, Yoo SJ, Lee JH, et al. Genetic framework for floweringtime regulation by ambient temperature-responsive miRNAs in Arabidopsis[J]. Nucleic Acids Res, 2010, 38:3081-3093.
[59]Mathieu, J, Yant, LJ, Murdter, F, et al. Repression of flowering by the miR172 target SMZ[J]. PLoS Biol, 2009, 7:e1000148.
[60]Ahn JH, Miller D, Winter VJ, et al. A divergent external loop confers antagonistic activity on floral regulators FT and TFL1[J]. EMBO J, 2006, 25:605-614.
[61]Searle I, He YH, Turck F, et al. The transcription factor FLC confers a flowering response to vernalization by repressing meristem competence and systemic signaling in Arabidopsis[J]. Gene Dev, 2006, 20:898-912.
[62]Willmann MR, Poethig RS. The effect of the floral repressor FLC on the timing and progression of vegetative phase change in Arabidopsis[J]. Development, 2011, 138:677-685.
[63]Chiang GCK, Barua D, Kramer EM, et al. Major flowering time gene, flowering locus C, regulates seed germination in Arabidopsis thaliana[J]. Proc Natl Acad Sci USA, 2009, 106:11661-11666.
[64]Swiezewski S, Liu F, Magusin A, Dean C. Cold-induced silencing by long antisense transcripts of an Arabidopsis Polycomb target[J]. Nature, 2009, 462:799-802.
[65]Heo JB, Sung S. Vernalization-mediated epigenetic silencing by a long intronic noncoding RNA[J]. Science, 2011, 331:76-79.
[66]Crevillen P, Dean C. Regulation of the floral repressor gene FLC:the complexity of transcription in a chromatin context[J]. Curr Opin Plant Biol, 2011, 14:38-44.
[67]Winter CM, Austin RS, Blanvillain-Baufume S, et al. LEAFY target genes reveal floral regulatory logic, cis motifs, and a link to biotic stimulus response[J]. Dev Cell, 2011, 20:430-443.
[68]Deal RB, Henikoff S. A simple method for gene expression and chromatin profiling of individual cell types within a tissue[J]. Dev Cell, 2010, 18:1030-1040.
(责任编辑 狄艳红)
Research Progress of Flowering Gene Regulatory Networks in Arabidopsis thaliana
Li Jing Gu Huiying Wang Zhimin Tang Qinglin Song Ming
(College of Horticulture and Landscape Architecture,Southwest University,Key Laboratory of Horticulture Science for Southern Mountainous Regions,Ministry of Education,Chongqing Key Laboratory of Olericulture,Chongqing 400715)
Flowering is one of the most important phase changes during the vegetative to reproductive growth in the life cycle of higher plants. And in recent years, flowering regulation has become a hot research in plant molecular biology. In current study, the gene regulatory network controlling flowering in Arabidopsis thaliana has grown up to a intricate web of crosstalk, feedback, and redundancy, bound tightly with other developmental processes by ‘process integrators’. The paper only briefly contextualizes the genetic pathways involved in regulating flowering, and focuses on the integration of signals at the shoot apical meristem(SAM), the spatial-temporal regulation of flower development, and finally functions that floral-related genes have in processes other than flowering time or flower development in A. thaliana. Also we discuss about the prospects of future research works, according to the present research status.
Flowering gene regulatory networks Flowering integrators Integration of signals Flower development Spatial-temporal regulation
10.13560/j.cnki.biotech.bull.1985.2014.12.001
2014-01-27
李敬,女,硕士,研究方向:蔬菜遗传育种与生物技术;E-mail:519054661@qq.com
