一种基于Voss映射下计算DNA序列3-周期特性的快速算法
2014-01-16黄绿娥鄢化彪
黄绿娥,鄢化彪
(江西理工大学,a.应用科学学院;b.理学院,江西赣州341000)
一种基于Voss映射下计算DNA序列3-周期特性的快速算法
黄绿娥a,鄢化彪b
(江西理工大学,a.应用科学学院;b.理学院,江西赣州341000)
DNA序列信号频谱3-周期特性被认为是用来区分编码区和非编码区的一个重要特征,传统的DNA序列分析中频谱计算量大占用了大量计算时间,使得分析效率极低.为提高DNA序列分析效率,针对传统频谱计算量大的问题,从3-周期特性原理出发,推导出了一种基于Voss映射下快速计算DNA序列3-周期频谱的方法.该方法有效避开计算离散傅立叶变换(DFT),从序列本身直接得到信噪比.实验结果表明快速算法计算效率是DFT方法的百倍之上,极大减小基因的信噪比计算时间,提高DNA序列识别中信噪比的计算效率.
DNA序列;Voss映射;外显子;3-周期性;信噪比
生物信息学(Bioinformatics)是生命科学、计算机科学、信息科学和数学等学科交汇融合形成的一门交叉学科.它以计算机、网络为工具,用数学和信息科学的理论、方法和技术去研究生物大分子,发现生物分子信息组织的规律.DNA序列数据是生物信息学中的主要研究对象之一,通过分析DNA序列的结构特征,不仅可以明晰已有序列,而且有利于发现新序列并预测其功能,而分析DNA序列的结构最基本的方法就是DNA序列分类[1-5]. 2000年全国大学生数学建模竞赛就将其列为竞赛题目,2012年全国研究生数学建模竞赛又将其列为赛题之一,充分说明了分类的难度.近年来,许多生物学家、数学家在这方面也做了大量的研究,分类方法也各异,例如哈尔滨工业大学张德丰等采用传统数理统计方法,沈阳大学岳晓宁等采用数据挖掘方法[6],东北电力大学敖丽敏等采用神经网络方法[7],等等.这些方法对于基因序列较短的问题效果明显,对于比较长的序列都面临计算量大、识别效率低的困境.为解决基因计算量大的问题,南京工业大学邵建峰教授所带领的团队采用DNA序列的3-周期特性分析方法[8-10],大大降低计算量.本文从DNA序列3-周期特性的原理出发,从数学角度推导一种能有效避开计算离散傅立叶变换(DFT)的快速计算方法,以提高DNA识别效率[11-13].
1 映射下DNA序列3-周期特性模型
在DNA序列研究中,首先需要把A、T、G、C四种核苷酸的符号序列,根据一定的规则映射成相应的数值序列,以便于对其作数字处理[1].
令I={A、T、G、C},长度(即核苷酸符号个数,又称碱基对(Base Pair)长度,单位记为bp)为N的任意DNA序列,可表达为:

即A、T、G、C的符号序列S:S(0),S(1),…,S(N-1).

对于任意确定的b∈I,令:对序列ub(n)分别做离散Fourier变换(DFT)有:
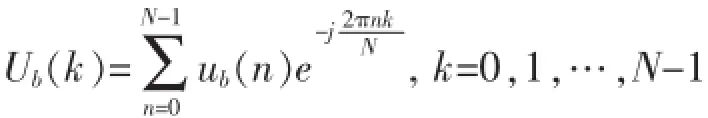
计算每个复数序列Ub(k)的平方功谱,并相加得到整个序列S的功谱序列:

由DNA序列外显因子的3-周期性[8-9],将序列1/3处的功谱值与整个序列的平均功谱做比较,其比率定义为该序列的“信噪比”(Signal Noise Ratio,SNR),即:

2 快速算法模型
根据功率谱定义可得,Ub(k)的平方功谱可以转化为:

DNA序列S的功谱为:

NDA序列S的总功谱为:

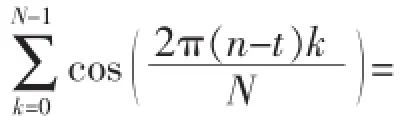

所以NDA序列S的总功谱为E=N2.
设xb、yb、zb分别为核苷酸b在序列的0,3,6,…和1,4,7,…以及2,5,8,…位置上出现的频数.的模平方可表示为:

所以:


3 算法算例分析
选用编号为BK006948.2的酵母基因DNA序列[10]的一段外显子(区间为[81787,82920],长度1134 bp)和一段内含子(区间为[96361,97551],长度1191 bp)的指示序列为例.
用传统DFT变换,并绘制功谱图如图1所示.

图1 BK006948.2的酵母基因一外显子和内含子功谱图
用DFT方法和快速算法对上述DNA序列片段进行分析,得到其效果比较如表1所示.

表1 传统DFT方法和快速算法的效果比较
从计算结果可以看出,快速算法和DFT方法计算结果完全相同,但计算效率是DFT算法的500倍,特别是当序列长度更大时,计算效率差异更明显.
4 结束语
本文建立了Voss映射下信噪比模型,从原理出发推导出了一种能直接计算信噪比的方法,避免计算离散傅立叶变换带来的计算复杂度.并通过实例与传统DFT方法进行比较,实验结果表明,快速算法完全保证了DFT方法的结果,但计算效率提高了上百倍,而且,当序列越长,计算效率比值越大.充分说明该方法适用于Voss映射下的长序列计算辨识问题.
[1]Sharma S D,Shakya K,Sharma S N.Evaluation of DNA mapping schemes for exon detection[C]//Computer,Communication and Electrical Technology (ICCCET),2011 International Conference on.IEEE,2011:71-74.
[2]Burge C,Karlin S.Prediction of complete gene structures in human genomic DNA[J].Journal of molecular biology,1997,268 (1):78-94.
[3]Berryman M J,Allison A,Wilkinson C R,et al.Review of signal processing in genetics[J].Fluctuation and Noise Letters,2005,5 (4):13-35.
[4]Anastassiou D.Frequency-domain analysisofbiomolecular sequences[J].Bioinformatics,2000,16(12):1073-1081.
[5]Kotlar D,Lavner Y.Gene prediction by spectral rotation measure: a new method for identifying protein-coding regions[J].Genome research,2003,13(8):1930-1937.
[6]岳晓宁,井元伟.基于DNA序列数据挖掘算法研究[J].生物数学学报,2009,24(2):363-368.
[7]敖丽敏,罗存金.基于神经网络集成的DNA序列分类方法研究[J].计算机仿真,2012,29(6):171-175.
[8]Yin C,Yau S S T.Prediction of protein coding regions by the 3-base periodicity analysis of a DNA sequence [J].Journal of theoretical biology,2007,247(4):687-694.
[9]邵建峰,严晓华,邵 伟,等.DNA序列信号3-周期特性[J].南京工业大学学报,2012,34(4):133-137.
[10]Goffeau A.TPA_inf:Saccharomyces cerevisiae S288c chromosome XV,complete sequence[EB/OL].[2012-09-18].http://www.ncbi. nlm.nih.gov/nuccore/329138966?report=fasta.x.
[11]黄绿娥,李平康.运动目标自动跟踪系统的控制平台设计[J].江西理工大学学报,2008,29(4):10-13.
[12]余水静,彭艳平,邓扬悟.一株嗜酸氧化亚铁硫杆菌分离及生长特性研究[J].江西理工大学学报,2011,32(5):1-4.
[13]李冬冬,王正志,杜耀华,等.DNA序列中模式发现的一种快速算法[J].生物物理学报,2005,21(2):121-129.
A fast algorithm to calculate the 3-periodicity characteristic in DNA sequence based on Voss mapped
HUANG Lv-ea,YAN Hua-biaob(a.Faculty of Applied Science;b.Faculty of Science,Jiangxi University of Science and Technology,Ganzhou 341000,China)
The 3-periodicity obtained by direct Fourier transform (FDT)was acknowledged as an important feature for distinguishing gene coding regions of a DNA sequence.Aim at the quantity work to calculate the value of DNA sequence by direct Fourier transform in calculate SNR,this paper given an expeditious algorithm based on Voss mapped DNA sequence.This method used DNA sequence's character,direct to calculate SNR without FDT,which acquiring the noise-signal ratio from the sequence.The experimental results show that the expeditious algorithm's computing efficiency is 100 times of DFT,which greatly reduced the gene SNR calculation time.
DNA sequence;Voss mapped;exon;3-periodicity;SNR
Q812;O242
A
2095-3041(2014)00-0098-04
10.13265/j.cnki.jxlgdxxb.2014.01.017
2013-06-21
江西省教育厅资助项目(JXJG-09-6-25)
黄绿娥(1981- ),女,硕士,讲师,主要从事系统控制与建模方法等方面的研究,E-mail:42673284@qq.com.
