一种有效的基于4D图形表示法的DNA序列相似性比较方法
2012-01-05黄大荣
阮 静,黄大荣
(1.云南大学 数学与统计学院,云南 昆明 650009;2.重庆交通大学 信息科学与工程学院,重庆 400074)
随着基因测序技术及数据库的发展,在过去的20年里发布在GenBank数据库(http://www.ncbi.nlm.nih.gov)里的序列数量正在以指数增长.许多序列需要知道它们的功能,或者已知功能同源性等来判定序列,因此序列比较已经成为了一个非常重要的研究领域之一.近年来,出来了很多计算机和统计的方法来对序列的比较.一些DNA序列的图形表示方法也随之出现,比如2D图形表示法,3D图形表示法,4D图形表示法[1-4].Liao等人[5-10]也提出了许多图形表示法.同时,在这些非退化的方法中,几何中心学的概念也被引用进来,结合相似性及不相似性矩阵来对DNA序列进行比较[11-12].然而,Chi等人[13]采用的4D图形表示方法的结果也出现了进化事实不相符合的相似性物种对,如人-山鸡、山鸡-老鼠.在Liao等人[14]用中心几何的方法来比较11个种类的相似性中,从比较的结果来看,存在与进化事实不太相符合的相似性物种对,如山羊-大猩猩、兔子-黑猩猩、牛-黑猩猩等.Tang等人[15]基于Z曲线的4D图形表示法相似性比较,其结果统计显著性不是很明显.
基于以上用4D图形表示方法对DNA序列进行比较的思想,本文提出了一种更有效的计算DNA序列相似性及不相似性的方法,通过这样的方法能够避免出现与进化事实不相符合的相似性物种,并且相似性与不相似性结果差异大,其统计显著性很明显
1 DNA序列的基本概述及中心几何点欧式距离计算方法
DNA序列由4个碱基组成,分别是A腺嘌呤,G鸟噤呤,C胞嘧啶,T胸腺嘧啶.根据不同的化学性质,Zhang[19]将它们分为3类,分别是:
(i)根据碱基化学结构的分类:R(嘌呤)=A,G;Y(嘧啶)=T,C.
(ii)根据碱基官能团的分类:M(氨基)=A,C;K(酮基)=G,T.
(iii)根据碱基配对的氢键强度分类:S(强氢键)=G,C;W=(弱氢键)A,T

对于每一条DNA序列本文把它平均分成两个片段,比如对于一条DNA序列G=g1,g2,…,gN,通过平分可以把它表示成两个片段并映射为A,B,即φ(G)=(A,B).
其中当N为偶数时,A=φ(g1)φ(g2)…φ(gN/2),B=φ(g(N+1)/2)φ(g(N+2)/2)…φ(gN);当N为奇数时,A=φ(g1)φ(g2)…φ(g(N+1)/2),B=φ(g(N+2)/2)φ(g(N+3)/2)…φ(gN).例如对于一条DNA序列G=ATGGCT,通过上述方法可以映射成{[(1,1,1,0),(0,0,1,1/2),(1,0,0,2/3)],[(1,0,0,3/4),(0,1,0,4/5),(0,0,1,5/6)]}.
在这里引入了几何中心点[18]的概念作为序列比较的指标.对任何一条DNA序列,都可以得到点(xi,yi,zi,si)构成的集(i=0,1,2,…,N,其中N是被研究的DNA序列长度).设这个几何中心点的坐标为(x0,y0,z0,s0),则通过公式(1)计算如下:
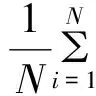
(1)
通过计算,得到几何中心坐标为:
(2)
其中:AN,GN,CN,TN分别表示为所研究的DNA序列中碱基A,G,T和C出现的次数.所以可以看出当x0≤y0时,说明Gn≤Cn否则Gn≥Cn;当y0≤z0时说明Cn≤Tn否则Cn≥Tn;当x0≤z0时说明Gn≤Tn否则Gn≥Tn.
在本文中,对于一条DNA序列G=g1,g2…gN其映射为φ(G)=(A,B),令点集A的几何中心坐标点为(x00,y00,z00,s00);点集B的几何中心坐标点为(x11,y11,z11,s11),其中:
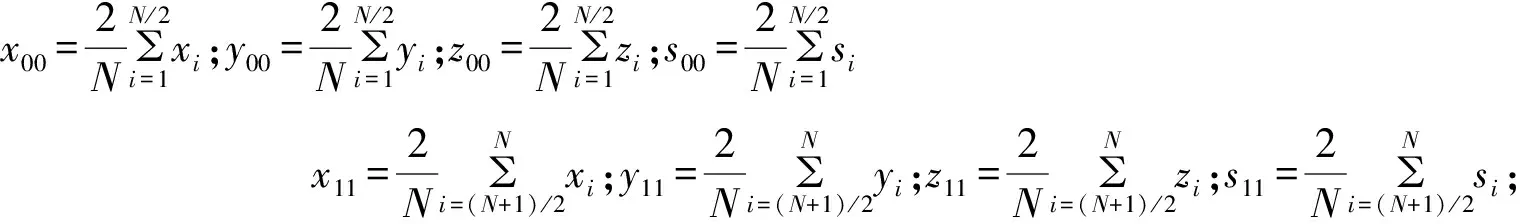
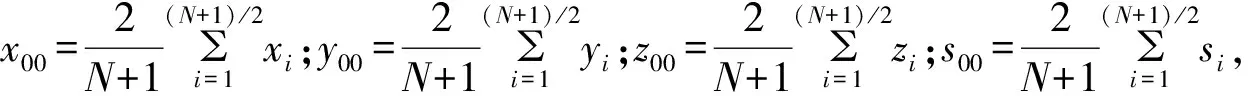
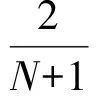
例如对于序列G=ATGGCT,其几何中心坐标为[(2/3,1/3,2/3,7/18),(1/3,1/3,1/3,143/180)].
2 DNA序列的相似性分析模型构建
假设两条序列的几何中心坐为:{[(x00(i),y00(i),z00(i),s00(i)],[x11(i),y11(i),z11(i),s11(i)]}和{[(x00(j),y00(j),z00(j),s00(j)],[x11(j),y11(j),z11(j),s11(j)]},本文用欧式距离来计算向量终点距离dij,dij定义如下:
很显然,欧式距离(dij)越小,说明相似性越大;反之,欧式距离越大,说明相似性越小.

表1 11个不同物种β-球蛋白基因的第一个外显子序列的几何中心坐标
3 实验论证
为了验证本文所论证的DNA序列相似性的有效性,本文选取了11个物种的β-球蛋白基因的第一个外显子作为研究对象,并在表1中给出了这11个不同物种的β-球蛋白基因的第一个外显子序列的几何中心.从表2中可以看出欧式距离越小其DNA序列相似性越高,进化上同源物种的欧式距离比进化上非同源的物种欧式距离小很多.从结果中可以发现山鸡与其他物种的相似性最小,其结果很显著,这是与进化事实相符的.因为山鸡是非哺乳动物,而其他10种物种都是哺乳动物.另还发现负鼠与其他物种的差异也较大,因为它是表中唯一的有袋动物.另外,最相似的物种对是人-黑猩猩、人-大猩猩、大猩猩-黑猩猩、牛-山羊、牛-兔子,从生物进化关系来看,这是符合事实的.
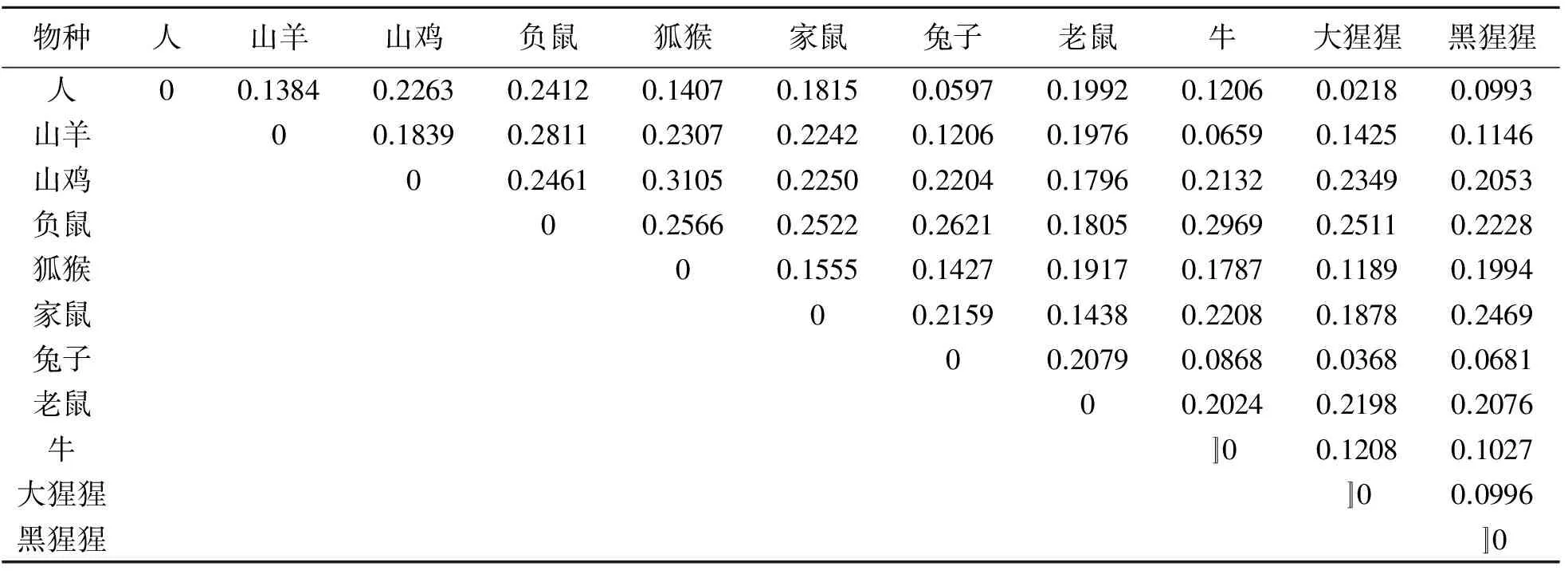
表2 基于向量终点之间欧氏距离的11个物种的相似性矩阵
4 结论
本文在基于4D图形表示方法的基础上提出了一种更有效的DNA序列相似性或不相似性比较的方法,并对11个物种的β-球蛋白基因的第一个外显子进行相似性分析,得到了很好结果.这种方法通过把DNA序列平均分成两个片段,从而把更多的位置信息保存下来,再引用几何中心学说的概念,利用欧式距离对这些几何中心集合进行计算,从而得到更加显著的相似性和不相似性结果.与最近提出的4D方法[13-14,16-17]的相似性分析结果相比,这种方法得到的结果更接近已知的进化事实.Liao等人[14]和Tang等人[15]得出的结果没有统计上的显著性,也就是说11个物种得到的欧式距离都非常小,而本文中的结果(表3)具有相似性的物种其欧式距离均小于0.1,而相似性很小的物种其欧式距离均大于0.1,没有同源性的物种其欧式距离均大于0.2.
[1] Nandya.A new graphical representation and analysis of DNA sequence structure I. methodology and application to globin genes[J].Curr Sci,1994,66:309-314.
[2] Randic M,Vracko M,Lers N,et al.Novel 2-D graphical representation of DNA sequences and their numerical characterization[J].Chem Phys Lett,2003,368:1-6.
[3] Randic M,Vracko M,Nandy A,et al.On 3-D graphical representation of DNA primary sequences and their numerical characterization[J].J Chem Inf Comput Sci,2000,40:1235-1244.
[4] Randic M.Graphical representations of DNA as 2-D map[J].Chem Phys Lett,2004,386:468-471.
[5] Liao B,Wang T M.Analysis of similarity/dissimilarity of DNA sequences based on 3-D graphical representation[J].Chem Phys Lett,2004,388:195-200.
[6] Liu Z B,Liao B,Zhu W,et al.A 2D graphical representation of DNA sequence based on dual nucleotides and its application[J].Int J Quantum Chem,2009,109:948-958.
[7] Liao B,Wang T M.New 2D graphical representations of DNA sequences[J].J Comput Chem,2004,25:1364-1368.
[8] Liao B.A 2D graphical representation of DNA sequence[J].Chem Phys Lett,2005,401:196-199.
[9] Liao B.Analysis of similarity/dissimilarity of DNA sequences based on a condensed curve representation[J].J Mol Struct (THEOCHEM),2005,717:199-203.
[10] Liao B,Wang T M.3-D graphical representation of D NA sequences and their numerical characterization[J].J Mol Struct (THEOCHEM),2004,681:209-212.
[11] Zhang R,Zhang C.Z curves,an intuitive tool for visualizing and analyzing the DNA sequences[J].J Biomol Struct Dyn,1994,11:767-782.
[12] Zhang C T,Zhang R,Ou H Y.The Z curve database: A graphic representation of genome sequence[J]. Bioinformatics,2003,19:593-599.
[13] Chi R,Ding K.Novel 4D numerical representation of DNA sequences[J].Chem Phys Lett,2005,407:63-67.
[14] Liao B,Tan M,Ding K.A 4D representation of DNA sequences and its application[J].Chemical Phys Letter,2005,402:380-383.
[15] Tang X C,Zhou P P,Qiu W Y.On the similarity/dissimilarity of DNA sequences based on 4D graphical representation[J].Chinese Science Bulletin,2010,55(8):701-704.
[16] Zhang C T.A symmetrical theory of DNA sequences and its applications[J].J Theor Biol,1997,187:297-306.
[17] Chenglong YU,Mo DDENG,STEPHEN S T Y.DNA sequence comparison by a novel probabilistic method[J].Information Science,2011,181:1484-1492.
